基于CSP的运动想象EEG分类任务实战
基于运动想象的公开数据集:Data set IVa (BCI Competition III)1
数据描述参考前文:https://blog.csdn.net/qq_43811536/article/details/134224005?spm=1001.2014.3001.5501
EEG 信号时频空域分析参考前文:https://blog.csdn.net/qq_43811536/article/details/134273470?spm=1001.2014.3001.5501
基于CSP的运动想象 EEG 特征提取和可视化参考前文:https://blog.csdn.net/qq_43811536/article/details/134296308?spm=1001.2014.3001.5501
CSP(Common Spatial Patterns)——EEG特征提取方法详解参考前文:https://blog.csdn.net/qq_43811536/article/details/134296840?spm=1001.2014.3001.5501
本文使用公开数据集 Data set IVa 中的部分被试数据,数据已公开可以从网盘获取:
链接:https://pan.quark.cn/s/5425ee5918f4
提取码:hJFz
目录
- 1. 实验介绍
- 2. 运动想象分类
-
- 2.1 分类性能
- 2.2 结论
- 3. 核心Python代码
1. 实验介绍
本任务的实验数据来自一名健康受试者,代号al。受试者在视觉提示出现后3.5s内完成以下3个运动想象中的一个:(L)左手,(R)右手,(F)右脚。分类任务中的数据只包括了右手和右脚两类,共280个试次。实验过程中使用脑电帽记录了118个通道的EEG信号,电极位置如图1所示。采集到的EEG信号首先经过带通滤波(0.05-200Hz),再经过数字化和下采样,得到采样率为100Hz的信号。
![]()
2. 运动想象分类
基于CSP特征,我们使用LDA分类器进行分类,并进行十折交叉验证以评估性能。评价指标为测试集准确率,即分类正确的试次占总试次的比例。
2.1 分类性能
我们比较了不同的带通滤波器和时间窗的结果。
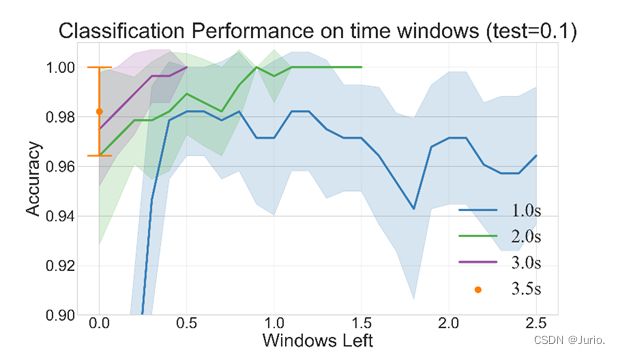
- 图1中,横轴为时间窗相较于提示出现的起始时间。不同的折线代表了不同窗长。我们发现在3s的窗长能获得更高的分类准确率,时间窗从提示出现后0.5s开始效果更好,分类准确率达到1。
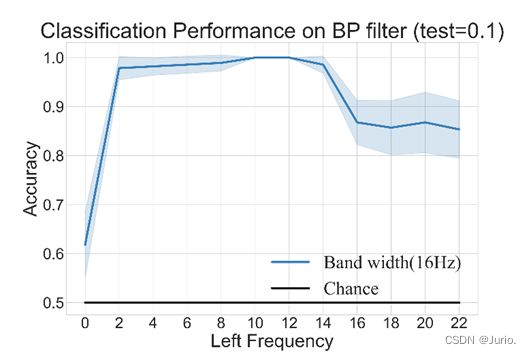
- 图2展示了滤波器截止频率对于准确率的影响,可以看到低频截止频率在10-12Hz时准确率能达到1。
- 我们还比较了LDA分类器与线性回归(LR)和随机森林(RF)方法的性能,结果如表1所示。LDA分类器的准确率高于LR和RF,但分类性能都较高。
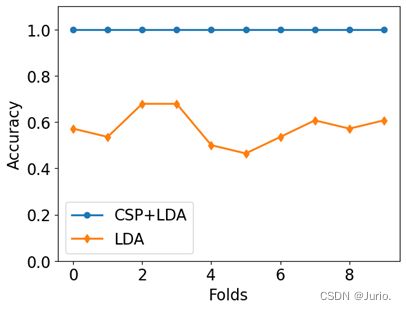
- 最后我们去掉提取CSP特征的模块,直接对原始信号使用LDA分类器,结果如图3所示。去除掉提取CSP模块后,分类准确率由1下降至0.6左右。
| 方法 | 准确率 |
|---|---|
| LDA | 1 |
| LR | 0.99±0.01 |
| RF | 0.99±0.01 |
2.2 结论
实验表明,右手和右脚运动想象的EEG差异集中于μ节律信号(8-15Hz)和β节律(18-24Hz),体现在C3和C4通道,即感觉运动区。使用CSP算法提取到的特征具有较高的线性可分性,使用LDA分类器可以实现准确率为1,能有效区分这两类运动想象。实验发现用于分类任务的时间窗范围和带通滤波范围对分类准确率具有较大影响,最优时间窗为提示出现后0.5s-3.5s,最优频带为12Hz-28Hz。
3. 核心Python代码
- 部分变量说明:
raw:由 mne.io.RawArray() 函数创建,代表原始EEG数据epochs:由 mne.Epochs() 函数创建,代表一个事件(event)对应的所有数据,在该数据集中一个事件即 “右手”或者“脚”的想象运动
# BP Filter
l_fr, h_fr = 12.0, 28.0
tMin, tMax = 0.5, 3.5
# MNE object
info = mne.create_info(
ch_names=[i[0] for i in ch_name],
sfreq=eeg_fs,
ch_types='eeg')
pos_dic = dict(zip(info.ch_names, ch_pos))
montage = mne.channels.make_dig_montage(pos_dic)
info.set_montage(montage)
raw = mne.io.RawArray(eeg_data.T, info)
# Apply band-pass filter
raw.filter(l_fr, h_fr, fir_design="firwin", skip_by_annotation="edge")
# Decoding
events = np.vstack((cues_pos, np.zeros(len(cues_pos)), target_label[0, :])).T.astype(int)
picks = mne.pick_types(raw.info, meg=False, eeg=True, stim=False, eog=False, exclude="bads")
# Epochs
epochs = mne.Epochs(
raw,
events,
events_id,
tMin,
tMax,
proj=True,
picks=picks,
baseline=None,
preload=True,
)
# Prepare data for training
x = epochs.get_data()
y = target_label[0, :]
# ten-fold cross-validation
cv = ShuffleSplit(10, test_size=test_r, random_state=42)
# Classification with LDA on CSP features
lda = LinearDiscriminantAnalysis()
csp = CSP(n_components=10, reg=None, log=True, norm_trace=False)
clf = Pipeline([("CSP", csp), ("LDA", lda)])
from sklearn.metrics import accuracy_score
train_x, test_x = x[:224], x[224:]
train_y, test_y = y[:224], y[224:]
clf.fit(train_x,train_y)
pred1 = clf.predict(train_x)
accuracy1 = accuracy_score(train_y,pred1)
print('在训练集上的精确度: %.4f'%accuracy1)
pred2 = clf.predict(test_x)
accuracy2 = accuracy_score(test_y,pred2)
print('在测试集上的精确度: %.4f'%accuracy2)
# 模型比较
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.ensemble import RandomForestClassifier
lda = LinearDiscriminantAnalysis()
csp = CSP(n_components=10, reg=None, log=True, norm_trace=False)
clf_lda = Pipeline([("CSP", csp), ("LDA", lda)])
scores_lda = cross_val_score(clf_lda, x, y, cv=cv, n_jobs=None)
lr = LogisticRegression()
csp = CSP(n_components=10, reg=None, log=True, norm_trace=False)
clf_lr = Pipeline([("CSP", csp), ("LR", lr)])
scores_lr = cross_val_score(clf_lr, x, y, cv=cv, n_jobs=None)
rfc = RandomForestClassifier()
csp = CSP(n_components=10, reg=None, log=True, norm_trace=False)
clf_rfc = Pipeline([("CSP", csp), ("RFC", rfc)])
scores_rfc = cross_val_score(clf_rfc, x, y, cv=cv, n_jobs=None)
print(scores_lda, scores_lr, 'scores_svc', scores_rfc)
# Without CSP
lda = LinearDiscriminantAnalysis()
scores_lda_only = cross_val_score(lda, x.reshape(-1,118*301), y, cv=cv, n_jobs=None)
print(scores_lda_only)
plt.plot(scores_lda,'-o',linewidth=2)
plt.plot(scores_lda_only,'-d',linewidth=2)
plt.xlabel('Folds',fontsize=16)
plt.ylabel('Accuracy',fontsize=16)
plt.legend(['CSP+LDA','LDA'],fontsize=16)
plt.xticks(fontsize=16)
plt.yticks(fontsize=16)
plt.ylim([0,1.1])
plt.show()
https://bbci.de/competition/iii/desc_IVa.html ↩︎