R语言——taxize(第二部分)
taxize(第二部分)
-
- 3. taxize 文档中译
-
- 3.10. classification(根据类群ID检索分类阶元层级)
-
- 示例1:传递单个ID值
- 示例2:传递多个ID值
- 示例3:传递单个名称
- 示例4:传递多个名称
- 示例5:传递字符型ID
- 示例6:传递带数据源的ID
- 示例7:传递多个数据库
- 示例8:横向/纵向组合
- 示例9:传递多个类群至多个数据库
- 示例10:查找维基物种
- 3.11. comm2sci(根据俗名检索科学名)
- 3.12. sci2comm(根据科学名检索俗名)
- 3.13. eol_dataobjects(根据数据对象的标识符,返回有关该对象的所有元数据)
- 3.14. eol_pages(根据taxonconceptID查找EOL的网页内容)
- 3.15. eol_search(从EOL数据源搜索术语)
- 3.16. eubon_capabilities(EUBON的功能支持)
- 3.17. eubon_children()
- 3.18. eubon_hierarchy()
- 3.19. eubon_search()
- 3.20. fungorum(fg数据库的一系列方法)
- 3.21. gbif_downstream(从GBIF上根据分类信息向下检索所有分类名称)
- 3.22. gbif_name_usage(在 GBIF 的所有分类标准中查找特定名称的详细信息)
- 3.23. gbif_parse(使用 GBIF 名称解析器解析分类群名称)
3. taxize 文档中译
资源https://cran.r-project.org/web/packages/taxize/taxize.pdf
介绍函数方法时,并不严格遵循文档的顺序。
3.10. classification(根据类群ID检索分类阶元层级)
用法:
classification(...)
## Default S3 method:
classification(sci_id, db=NULL, callopts=list(), return_id=TRUE, rows=NA, x=NULL, ...)
## S3 method for class 'tsn'
classification(id, return_id=TRUE, ...)
## S3 method for class 'uid'
classification(id, callopts=list(), return_id=TRUE, batch_size-50, max_tries=3, ...)
## S3 method for class 'eolid'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'tpsid'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'gbifid'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'nbnid'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'tolid'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'wormsid'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'natservid'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'boldid'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'wiki'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'pow'
classification(id, callopts=list(), return_id=TRUE, ...)
## S3 method for class 'ids'
classification(id, ...)
## S3 method for class 'classification'
cbind(...)
## S3 method for class 'classification'
rbind(...)
## S3 method for class 'classification_ids'
cbind(...)
## S3 method for class 'classification_ids'
rbind(...)
参数:
- …:对于classification而言,此项包括传递给get_tsn(),get_uid(),get_eolid(),get_tpsid(),get_gbifid(),get_wormsid(),get_natservid(),get_wiki(),get_pow()的参数。对于rbind.classification和cbind.classification:此项则是classification类的单个或多个对象。
- sci_id:用于检索的类群名称(字符型)或ID(字符型或数值型)向量。当db=“eol”,EOL期望你传递的是一个类群id,即*get_eolid()*返回值。
- db:(字符型)检索数据库。任一ncbi,itis,eol,tropicos,gbif,nbn,worms,natserv,bold,wiki,或pow。每个数据库都有自己的一套标识符,如果提供不属于数据库的标识符,尽管有可能返回结果,但基本上不是期望值。如果使用了ncbi/tropicos,建议使用API密钥;详见taxize-authentication。
- callopts:传递给crul::verb-GET的curl选项。
- return_id:(逻辑值)默认为TRUE,返回类群id和名称以及阶元。数据库natserv将忽略此参数。
- rows:(数值型)从1到无穷大的整数。默认为NA,所有行都有效。如果你传递的是类群id而不是类群名称,此参数被忽略。
- x:已弃用,请使用sci-id。
- id:(数值型)标识符,由 get_tsn(),get_uid(),get_eolid(),get_tpsid(),get_gbifid(),get_wormsid(),get_natservid(),get_wiki(),get_pow() 返回的结果。
- batch_size:(数值型)针对NCBI请求,指定每次请求查找的ID数量。
- max_tries:(数值型)针对NCBI请求,当首次请求失败时,决定再尝试请求的次数。
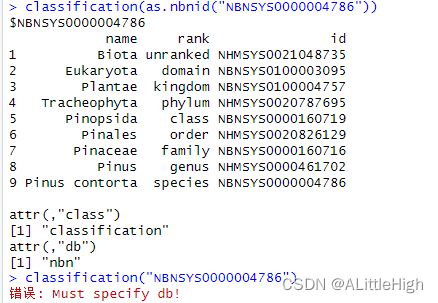
说明:如果你直接传递了ID值(而不是get_*系列方法的返回值),那么你必须指定ID归属的数据库。目前NBN不会在返回的分类信息中包含请求类群。
返回值:由每个请求类群的分类信息的数据框组成的命名列表。
EOL:EOL的无错误处理并不友好。比如,如果你传递了一个并不存在的ID值,那么EOL将返回500 HTTP错误,。
NCBI请求限制:以防万一你触发了NCBI的请求速率限制导致出错,参考taxize_options(),你可以设置ncbi_sleep。
NCBI请求的HTTP版本:本方法硬编码了http_version=2L以便让HTTP请求使用HTTP/1.1访问Entrez API。详见curl::curl_symbols(“CURL_HTTP_VERSION”)。
示例1:传递单个ID值
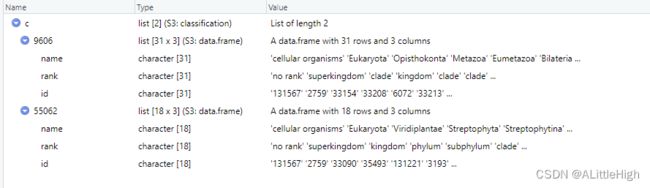
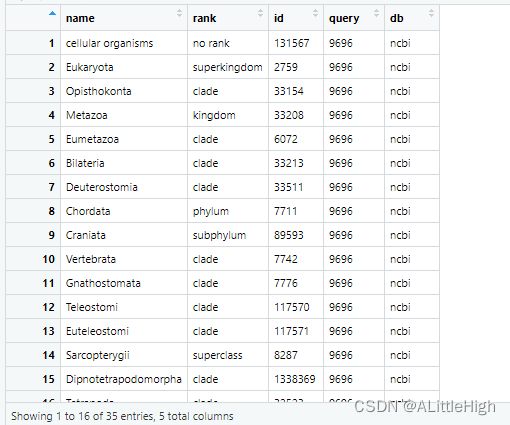
classification(9606, db='ncbi')
示例2:传递多个ID值
c <- classification(c(9606, 55062), db='ncbi')
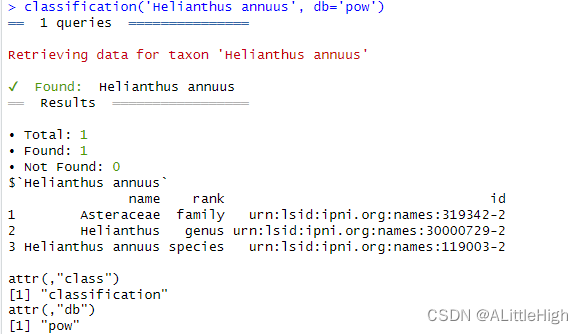
示例3:传递单个名称
classification('Helianthus annuus', db='pow')
示例4:传递多个名称
c <- classification(c("Helianthus annuus", "Mahonia bealei"), db='pow')
示例5:传递字符型ID
classification("134717", db="natserv")
示例6:传递带数据源的ID
classification(as.nbnid("NBNSYS0000004786"))
c <- classification(get_gbifid(c("Poa annua", "Bison bison")))
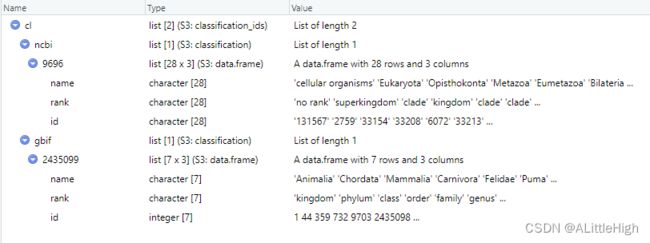
示例7:传递多个数据库
out <- get_ids("Puma concolor", db = c('ncbi','gbif'))
cl <- classification(out)
示例8:横向/纵向组合
cb <- cbind(cl)
rb <- rbind(cl)
示例9:传递多个类群至多个数据库
out <- get_ids(c("Puma concolor","Accipiter striatus"), db = c('ncbi','itis'))
cl <- classification(out)
示例10:查找维基物种
classification("Malus domestica", db = "wiki")
✔ Found: Malus domestica
══ Results ═════════════════
• Total: 1
• Found: 1
• Not Found: 0
$`Malus domestica`
name rank
1 Eukaryota Superregnum
2 Plantae Regnum
3 Angiosperms Cladus
4 Eudicots Cladus
5 Core eudicots Cladus
6 Rosids Cladus
7 Eurosids I Cladus
8 Rosales Ordo
attr(,"class")
[1] "classification"
attr(,"db")
[1] "wiki"
attr(,"wiki_site")
[1] "species"
attr(,"wiki")
[1] "en"
3.11. comm2sci(根据俗名检索科学名)
用法:
comm2sci(...)
## Default S3 method:
comm2sci(com, db="ncbi", itisby="search", simplify=TRUE, commnames=NULL, ...)
## S3 method for class "tsn"
comm2sci(id, db="ncbi", itisby="search", simplify=TRUE, ...)
## S3 method for class "uid"
comm2sci(id, db="ncbi", itisby="search", simplify=TRUE, ...)
参数:
- …:传递给内部调用函数的参数。
- com:单个或多个俗名或部分名称。
- db:数据源,任一ncbi(默认),itis,tropicos,eol或worms。我们建议通过获取API密钥使用ncbi数据库,参考taxize_authentication。
- itisby:在整个名称中搜索俗名(默认,search),在名称起始位置搜索(begin),在名称末尾搜索(end)。
- simplify:(逻辑值)为TRUE时,返回名称向量。为FALSE时,从各种数据源返回所有可能的结果,通常是一个数据框。
- commnames:已弃用,请使用com。
- id:类群标识符,由*get_tsn()或get_uid()*返回。
说明:当针对ITIS和NCBI数据库时,可以直接传递俗名,或者通过*get_uid()或get_tsn()*先获取id再传递给此函数。而针对其他数据库,仅能传递俗名。
返回值:如果simplify=TRUE,一列科学名会出现在传入的名称的后面。如果simplify=FALSE,返回一个数据框,根据不同的数据源而包含不同的列。如果没有成功匹配,则返回character(0)。
NCBI请求限制:以防万一你触发了NCBI的请求速率限制导致出错,参考taxize_options(),你可以设置ncbi_sleep。
NCBI请求的HTTP版本:本方法硬编码了http_version=2L以便让HTTP请求使用HTTP/1.1访问Entrez API。详见curl::curl_symbols(“CURL_HTTP_VERSION”)。
示例:
-
只传递一个俗名
comm2sci(com='american black bear')$`american black bear` [1] "Ursus americanus" -
传递多个俗名
comm2sci(com=c('annual blue grass','tree of heaven'), db='tropicos')$`annual blue grass` [1] "Poa annua" $`tree of heaven` [1] "Toxicodendron altissimum" -
传递ID
x <- get_uid("western capercaillie", modifier = "Common Name") comm2sci(x)$`100830` [1] "Tetrao urogallus"
3.12. sci2comm(根据科学名检索俗名)
用法:
sci2comm(...)
## Default S3 method:
sci2comm(sci, db="ncbi", simplify=TRUE, scinames=NULL, ...)
## S3 method for class 'uid'
sci2comm(id, ...)
## S3 method for class 'tsn'
sci2comm(id, simplify=TRUE, ...)
## S3 method for class 'wormsid'
sci2comm(id, simplify=TRUE, ...)
## S3 method for class 'iucn'
sci2comm(id, simplify=TRUE, ...)
参数:
- …:传递给*get_uid()和get_tsn()*的参数。
- sci:(字符型)单个或多个科学名或部分名称。
- db:(字符型)数据源,任一ncbi(默认)、itis、eol、worms或iucn。每个数据库都有自己的一套标识符,如果提供不属于数据库的标识符,尽管有可能返回结果,但基本上不是期望值。我们建议在使用ncbi或iucn时添加API密钥,参考taxize-authentication。
- simplify:(逻辑值)为TRUE时,只返回名称向量。为FALSE时,返回一个数据框,数据源不同导致包含的列不同。仅适用于eol和itis。指定FALSE以获取eol和itis的输出中每种方言的语言。
- scinames:已弃用,请使用sci。
- id:(字符型)类群标识符,由*get_tsn()或get_uid()*返回。
返回值:字符向量列表,根据传入的类群名称或类群ID命名。匹配失败返回character(0)。
NCBI请求的HTTP版本:本方法硬编码了http_version=2L以便让HTTP请求使用HTTP/1.1访问Entrez API。详见curl::curl_symbols(“CURL_HTTP_VERSION”)。
示例:
-
传递单个科学名
sci2comm(sci='Helianthus annuus')$`Helianthus annuus` [1] "common sunflower" -
传递多个科学名
sci2comm(sci=c('Helianthus annuus', 'Poa annua'))$`Helianthus annuus` [1] "common sunflower" $`Poa annua` character(0) -
传递id值
sci2comm(get_uid('Helianthus annuus'))$`4232` [1] "common sunflower"
3.13. eol_dataobjects(根据数据对象的标识符,返回有关该对象的所有元数据)
用法:
eol_dataobjects(id, taxonomy=TRUE, language=NULL, ...)
参数:
- id:(字符值)EOL数据对象标识符。
- taxonomy:(逻辑值)决定是否返回类群阶元层级的详细信息,存储在taxonconcepts数组。
- language:(字符型)结果使用哪种语言。可用值有:ms、de、en、es、fr、gl、it、nl、nb、oc、pt-BR、sv、tl、mk、sr、uk、ar、zh-Hans、zh-Hant和ko。
- …:传递给crul::HttpClient的可选参数。
说明:EOL API支持返回JSON或XML,而此方法只能返回JSON。
返回值:一个列表,当taxonomy=TRUE时还会返回一个数据框。
3.14. eol_pages(根据taxonconceptID查找EOL的网页内容)
用法:
eol_pages(taxonconceptID, images_per_page = NULL, images_page = NULL, videos_per_page = NULL, videos_page = NULL, sounds_per_page = NULL, sounds_page = NULL, maps_per_page = NULL, maps_page = NULL, texts_per_page = NULL, texts_page = NULL, subjects = "overview", licenses = "all", details = FALSE, common_names = FALSE, synonyms = FALSE, references = FALSE, taxonomy = TRUE, vetted = 0, cache_ttl = NULL, ...)
参数:
- taxonconceptID:(数值型)一个taxonconceptID,也是页码。
- images_per_page:(整数型)返回的图片数量(0-75)。
- images_page:(整数型)图片页。
- videos_per_page:(整数型)返回的影像数量(0-75)。
- videos_page:(整数型)影像页。
- sounds_per_page:(整数型)返回的录音数量(0-75)。
- sounds_page:(整数型)录音页。
- maps_per_page:(整数型)返回的地图数量(0-75)。
- maps_page:(整数型)地图页。
- texts_per_page:(整数型)返回的文本数量(0-75)。
- texts_page:(整数型)文本页。
- subjects:为overview时(默认),返回概述(只要存在),管道符 | 通过标题名进行筛选。为all时,返回所有文本。
- licenses:通过管道符筛选许可证,或者all选择所有许可证。
- details:默认为FALSE。包含所有元数据。
- common_names:默认为FALSE。返回所有俗名。
- synonyms:默认为FALSE。返回所有异名。
- references:默认为FALSE。返回所有文献。
- taxonomy:(逻辑型)默认为TRUE。决定是否返回任何详细的分类信息,存储在taxonconcepts数组。
- vetted:默认为FALSE。如果为*‘1’,只返回可信的内容。如果为‘2’*,只返回可信的和未审阅的内容(而不可信内容不会返回)。默认返回所有内容。
- cache_ttl:希望缓存响应的秒数。
- …:传递给crul::HttpClient的参数。
说明:EOL API支持返回JSON或XML,而此方法只能返回JSON。
返回值:JSON列表或数据库。
示例:
pageid <- eol_search('Pomatomus')$pageid[1]
x <- eol_pages(taxonconceptID = pageid)
z <- eol_pages(taxonconceptID = pageid, synonyms = TRUE)
z <- eol_pages(taxonconceptID = pageid, common_names = TRUE)
z$vernacular
vernacularname language eol_preferred
1 bluefish en TRUE
3.15. eol_search(从EOL数据源搜索术语)
用法:
eol_search(sci, page=1, exact=NULL, filter_tid=NULL, filter_heid=NULL, filter_by_string=NULL, cache_ttl=NULL, terms=NULL, ...)
参数:
- sci:(字符型)科学名。
- page:每页最多返回30条结果。此参数允许你获取多个页面,以防结果大于30条(默认为1)。
- exact:查找类群页面,只要优先名称、任何异名或俗名与检索术语完全匹配。
- filter_tid:根据EOL页面ID,将搜索结果限制在分类群的成员内。
- filter_heid:根据分类层级条目ID,将搜索结果限制在分类群的成员内。
- filter_by_string:根据搜索术语进行绝对检索,匹配页面将作为分类群对搜索结果进行筛选。
- cache_ttl:希望缓存响应的秒数。
- …:传递给crul::HttpClient的参数。
说明:EOL API支持返回JSON或XML,而此方法只能返回JSON。
返回值:
- 包含四列的数据框:
- pageid:页面ID,与使用get_eolid() 返回的eolid相同。
- name:分类名称,不一定包含命名人。
- link:请求URL。
- content:由分号分隔的名称组成的字符串。需要由用户来发掘它是否包含有用的信息。
示例:
e <- eol_search(sci="Homo")
3.16. eubon_capabilities(EUBON的功能支持)
用法:eubon_capabilities(...)
参数:…:传递给crul::verb-GET的curl可选项参数。
示例:eubon_capabilities()
3.17. eubon_children()
用法:eubon_children(id, providers = NULL, timeout = 0, ...)
参数:
- id:(字符型)类群的ID。包括LSID,DOI,URI,或任何由清单中的供应商提供的标识符。
- providers:(字符型)由逗号字符连接的供应商id字符串列表。默认为pesi,bgbm-cdm-server[col]。可以从’ /capabilities '服务端点获得所有可用供应商id的列表。供应商可以嵌套,也就是说,父供应商可以有子供应商。如果提供了父供应商的id,将查询所有子供应商。还可以使用以下语法将查询限制为一个或多个子供应商:parent-id[sub-id-1,sub-id2,…]
- timeout:(数值型)等待任何供应商的响应的最大毫秒数。如果超过了响应时间,服务将只返回到目前为止已经接收到的响应。默认超时为0毫秒(永远等待)。
- …:传递给crul::verb-GET的curl可选项参数。
返回值:一个数据框,或者没有找到结果的话就返回一个空列表。
说明:此方法中没有分页,因此您可能会也可能不会获得搜索的所有结果。
示例:x <- eubon_children(id = "urn:lsid:marinespecies.org:taxname:126141", providers = 'worms')
3.18. eubon_hierarchy()
用法:eubon_hierarchy(id, providers = "pesi", timeout = 0, ...)
参数:
- id:(字符型)类群的ID。包括LSID,DOI,URI,或任何由清单中的供应商提供的标识符。
- providers:(字符型)由逗号字符连接的供应商id字符串列表。默认为pesi,bgbm-cdm-server[col]。可以从’ /capabilities '服务端点获得所有可用供应商id的列表。供应商可以嵌套,也就是说,父供应商可以有子供应商。如果提供了父供应商的id,将查询所有子供应商。还可以使用以下语法将查询限制为一个或多个子供应商:parent-id[sub-id-1,sub-id2,…]
- timeout:(数值型)等待任何供应商的响应的最大毫秒数。如果超过了响应时间,服务将只返回到目前为止已经接收到的响应。默认超时为0毫秒(永远等待)。
- …:传递给crul::verb-GET的curl可选项参数。
返回值:一个数据框,或者没有找到结果的话就返回一个空列表。
说明:此方法中没有分页,因此您可能会也可能不会获得搜索的所有结果。
示例:eubon_hierarchy(id = "urn:lsid:marinespecies.org:taxname:126141", 'worms')
3.19. eubon_search()
用法:
eubon_search(query, providers="pesi", searchMode="scientificNameExact", addSynonymy=FALSE, addParentTaxon=FALSE, timeout=0, dedup=NULL, limit=20, page=1, ...)
参数:
- query:(字符型)搜索的科学名。这是一个精确搜索,因此不支持通配符。
- providers:(字符型)逗号字符连接的供应商id字符串列表。默认为pesi,bgbm-cdm-server[col]。可以从’ /capabilities '服务端点获得所有可用供应商id的列表。供应商可以嵌套,也就是说,父供应商可以有子供应商。如果提供了父供应商的id,将查询所有子供应商。还可以使用以下语法将查询限制为一个或多个子供应商:parent-id[sub-id-1,sub-id2,…]。
- searchMode:(字符型)指定搜索模式。可能的搜索模式有:scientificNameExact, scientificNameLike(以开头),vernacularnamexact, vernacularNameLike(contains), findByIdentifier。如果供应商不支持所选的搜索模式,它将被跳过,并且tnrClientStatus中的状态消息将被设置为“不支持的搜索模式”。
- addSynonymy:(逻辑型)指示是否应将接受的分类单元的同义词包含在响应中。打开此选项可能会增加响应时间。默认为FALSE。
- addParentTaxon:(逻辑型)表示所接受分类群的父分类群是否应包含在响应中。打开此选项可能会导致响应时间略有增加。默认为FALSE。
- timeout:(数值型)等待任何供应商的响应的最大毫秒数。如果超过了响应时间,服务将只返回到目前为止已经接收到的响应。默认超时为0毫秒(永远等待)。
- dedp:(数值型)通过使用重复数据删除策略,可以对结果进行重复数据删除。重复计算是通过比较分类群的特定属性来完成的:
- id:对比taxon.identifier。
- id_name:对比taxon.identifier和taxon.taxonName.scientificName。
- name:对比taxon.taxonName.scientificName。不推荐使用此策略。
- limit:(数值型/整数型)返回的记录数量。默认为20。此参数只影响scientificNameLike和vernacularNameLike;其余搜索模式每个检查列表只返回一条记录。
- page:(数值型/整数型)返回的页面。默认为1.此参数只影响scientificName和vernacularNameLike;其余搜索模式每个检查列表只返回一条记录。
- …:传递给crul::verb-GET的curl可选项参数。
示例:eubon_search("Prionus")
3.20. fungorum(fg数据库的一系列方法)
描述:在全球真菌名录中搜索发分类名称。
用法:
fg_name_search(q, anywhere=TRUE, limit=10, ...)
fg_author_search(q, anywhere=TRUE, limit=10, ...)
fg_epithet_search(q, anywhere=TRUE, limit=10, ...)
fg_name_by_key(key, ...)
fg_name_full_by_lsid(lsid, ...)
fg_all_updated_names(date, ...)
fg_deprecated_names(date, ...)
参数:
- q:(字符型)查询词。
- anywhere:(逻辑型)默认为TRUE。
- limit:(整数型)返回的结果数量。最大值为6000.
- …:传递给crul::verb-GET的curl可选项参数。
- key:(字符型)一个全球真菌名录的类群key。
- lsid:(字符型)一个LSID,例如urn:lsid:indexfungorum.org:names:81085。
- date:(字符型)日期,格式如下:YYYMMDD。
返回值:一个数据框,或者如果没有结果就返回NULL。
示例:
f <- fg_name_search(q = "Gymnopus")
f <- fg_epithet_search(q = "phalloides")
f <- fg_name_by_key(17703)
f <- fg_name_full_by_lsid("urn:lsid:indexfungorum.org:names:81085")
f <- fg_all_updated_names(date = gsub("-", "", Sys.Date() - 2))
f <- fg_deprecated_names(date=20151001)
f <- fg_author_search(q = "Fayod", limit = 2)
3.21. gbif_downstream(从GBIF上根据分类信息向下检索所有分类名称)
用法:
gbif_downstream(id, downto, intermediate=FALSE, limit=100, start=NULL, key=NULL, ...)
参数:
- id:一个分类学序列号。
- downto:您希望深入到的分类阶元。请参见下面的示例。分类级别区分大小写,必须拼写正确。拼写请参见数据 (rank_ref)。
- intermediate:(逻辑型)为TRUE时,则返回包含目标分类群等级名称的长度为 2 的列表、
附加的中间分类群 data.frame 列表。默认为FALSE。 - limit:要返回的记录数。默认值:100,最大值:1000: 与start参数结合使用。
- start:返回记录的起始编号。默认为0。与limit参数结合使用。
- key:已弃用,请使用id。
- …:传递给gbif_name_usage() 的参数。
说明:有时,记录中没有我们要查找的规范名称条目。在这种情况下,我们会抓取scientificName。您可以在 name_type 列中看到所收集的名称类型。
返回值:从目、类等到科的下游分类信息的 data.frame,或者如果 intermediated=TRUE,则返回长度为 2 的列表,包含目标分类群等级名称和中间名称。
示例:
gbif_downstream(id = 198, downto="genus")
name rank key name_type
1 Allogonium genus 2651958 canonicalname
2 Bangia genus 2653776 canonicalname
3 Bangiomorpha genus 4907128 canonicalname
4 Boreophyllum genus 6102075 canonicalname
5 Clymene genus 8203169 canonicalname
6 Conchocelichnus genus 12349604 canonicalname
7 Dione genus 7952618 canonicalname
8 Fuscifolium genus 6784971 canonicalname
9 Kuwaitiella genus 12275417 canonicalname
10 Lysithea genus 6784972 canonicalname
11 Minerva genus 4376334 canonicalname
12 Neomiuraea genus 10933480 canonicalname
13 Neoporphyra genus 10840017 canonicalname
14 Neopyropia genus 11224418 canonicalname
15 Neothemis genus 10040559 canonicalname
16 Palaeoconchocelis genus 4922773 canonicalname
17 Phycocalidia genus 11328097 canonicalname
18 Porphyra genus 2653483 canonicalname
19 Porphyrea genus 4602743 canonicalname
20 Porphyrella genus 2653774 canonicalname
21 Pseudobangia genus 4376337 canonicalname
22 Pyropia genus 4908866 canonicalname
23 Spermogonia genus 4907552 canonicalname
24 Themis genus 8274639 canonicalname
25 Uedaea genus 11329911 canonicalname
26 Wildemania genus 2653758 canonicalname
27 Wildemania genus 9738674 canonicalname
28 Zachariasia genus 2653756 canonicalname
29 Granufilum genus 7632635 canonicalname
30 Chroodactylon genus 2665908 canonicalname
31 Stylonema genus 7864726 canonicalname
3.22. gbif_name_usage(在 GBIF 的所有分类标准中查找特定名称的详细信息)
描述:这是 rgbif 软件包中相同函数的税化版本,因此无需导入 rgbif,也就无需安装 GDAL 二进制程序。
用法:
gbif_name_usage(key=NULL, name=NULL, data='all', language=NULL, datasetKey=NULL, uuid=NULL, sourceId=NULL, rank=NULL, shortname=NULL, start=NULL, limit=20, ...)
参数:
- key:(数值型)一个类群的GBIF 密钥。
- name:(字符型)通过不区分大小写的规范名称字符串进行筛选。
- data:(字符型)指定一个选项,以选择返回哪些数据。
- language:(字符型)语言类型。默认为英语。
- datasetKey:(字符型)通过数据集的键(uuid)进行筛选。
- uuid:(字符型)数据集的 uuid。结果应与 datasetKey 完全相同。
- sourceId:(数值型)根据源标识符进行筛选。目前尚未使用。
- rank:(字符型)分类等级。根据以下分类等级之一进行筛选:CLASS, CULTIVAR, CULTIVAR_GROUP, DOMAIN, FAMILY, FORM, GENUS, INFORMAL, INFRAGENERIC_NAME, INFRAORDER, INFRASPECIFIC_NAME,INFRASUBSPECIFIC_NAME, KINGDOM, ORDER, PHYLUM, SECTION,SERIES, SPECIES, STRAIN, SUBCLASS, SUBFAMILY, SUBFORM, SUBGENUS, SUBKINGDOM, SUBORDER, SUBPHYLUM, SUBSECTION, SUBSERIES, SUBSPECIES, SUBTRIBE, SUBVARIETY, SUPERCLASS, SUPERFAMILY, SUPERORDER, SUPERPHYLUM, SUPRAGENERIC_NAME, TRIBE,UNRANKED, VARIETY
- shortname:(字符型)简称。
- shart:返回记录的起始编号。
- limit:要返回的记录数。
- …:传递给crul::HttpClient的可选参数。
返回值:长度为 2 的列表。第一个元素是元数据。第二个元素是 data.frame(verbose=FALSE,默认)或列表(verbose=TRUE)。
3.23. gbif_parse(使用 GBIF 名称解析器解析分类群名称)
用法:gbif_parser(scientificname, ...)
参数:
- scientificname:(字符型)科学名。
- …:传递给crul::verb-POST的可选参数。
返回值:包含从已解析的分类群名称中提取的字段的 data.frame。返回的字段是从科学名称(scientificname)中的所有物种名称中提取的字段的组合。
示例:
gbif_parse(scientificname='x Agropogon littoralis')
scientificname type genusorabove specificepithet notho parsed parsedpartially canonicalname
1 x Agropogon littoralis SCIENTIFIC Agropogon littoralis GENERIC TRUE FALSE Agropogon littoralis
canonicalnamewithmarker canonicalnamecomplete rankmarker
1 ×Agropogon littoralis ×Agropogon littoralis sp.