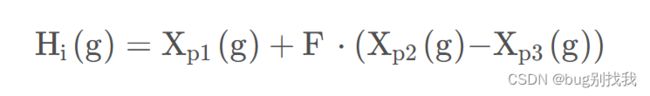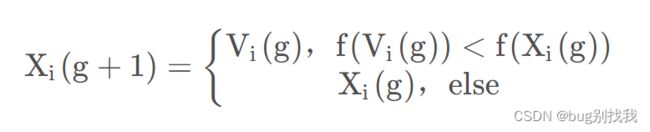- 机器学习与深度学习间关系与区别
ℒℴѵℯ心·动ꦿ໊ོ꫞
人工智能学习深度学习python
一、机器学习概述定义机器学习(MachineLearning,ML)是一种通过数据驱动的方法,利用统计学和计算算法来训练模型,使计算机能够从数据中学习并自动进行预测或决策。机器学习通过分析大量数据样本,识别其中的模式和规律,从而对新的数据进行判断。其核心在于通过训练过程,让模型不断优化和提升其预测准确性。主要类型1.监督学习(SupervisedLearning)监督学习是指在训练数据集中包含输入
- 网易严选官方旗舰店,优质商品,卓越服务
高省_飞智666600
网易严选官方旗舰店是网易旗下的一家电商平台,以提供优质商品和卓越服务而闻名。作为一名SEO优化师,我将为您详细介绍网易严选官方旗舰店,并重点强调其特点和优势。大家好!我是高省APP最大团队&联合创始人飞智导师。相较于其他返利app,高省APP的佣金更高,模式更好,最重要的是,终端用户不会流失!高省APP佣金更高,模式更好,终端用户不流失。【高省】是一个自用省钱佣金高,分享推广赚钱多的平台,百度有几
- Goolge earth studio 进阶4——路径修改与平滑
陟彼高冈yu
Googleearthstudio进阶教程旅游
如果我们希望在大约中途时获得更多的城市鸟瞰视角。可以将相机拖动到这里并创建一个新的关键帧。camera_target_clip_7EarthStudio会自动平滑我们的路径,所以当我们通过这个关键帧时,不是一个生硬的角度,而是一个平滑的曲线。camera_target_clip_8路径上有贝塞尔控制手柄,允许我们调整路径的形状。右键单击,我们可以选择“平滑路径”,这是默认的自动平滑算法,或者我们可
- 基于社交网络算法优化的二维最大熵图像分割
智能算法研学社(Jack旭)
智能优化算法应用图像分割算法php开发语言
智能优化算法应用:基于社交网络优化的二维最大熵图像阈值分割-附代码文章目录智能优化算法应用:基于社交网络优化的二维最大熵图像阈值分割-附代码1.前言2.二维最大熵阈值分割原理3.基于社交网络优化的多阈值分割4.算法结果:5.参考文献:6.Matlab代码摘要:本文介绍基于最大熵的图像分割,并且应用社交网络算法进行阈值寻优。1.前言阅读此文章前,请阅读《图像分割:直方图区域划分及信息统计介绍》htt
- 121. 买卖股票的最佳时机
薄荷糖的味道_fb40
给定一个数组,它的第i个元素是一支给定股票第i天的价格。如果你最多只允许完成一笔交易(即买入和卖出一支股票),设计一个算法来计算你所能获取的最大利润。注意你不能在买入股票前卖出股票。示例1:输入:[7,1,5,3,6,4]输出:5解释:在第2天(股票价格=1)的时候买入,在第5天(股票价格=6)的时候卖出,最大利润=6-1=5。注意利润不能是7-1=6,因为卖出价格需要大于买入价格。示例2:输入:
- 每日算法&面试题,大厂特训二十八天——第二十天(树)
肥学
⚡算法题⚡面试题每日精进java算法数据结构
目录标题导读算法特训二十八天面试题点击直接资料领取导读肥友们为了更好的去帮助新同学适应算法和面试题,最近我们开始进行专项突击一步一步来。上一期我们完成了动态规划二十一天现在我们进行下一项对各类算法进行二十八天的一个小总结。还在等什么快来一起肥学进行二十八天挑战吧!!特别介绍小白练手专栏,适合刚入手的新人欢迎订阅编程小白进阶python有趣练手项目里面包括了像《机器人尬聊》《恶搞程序》这样的有趣文章
- 回溯算法-重新安排行程
chirou_
算法数据结构图论c++图搜索
leetcode332.重新安排行程这题我还没自己ac过,只能现在凭着刚学完的热乎劲把我对题解的理解记下来。本题我认为对数据结构的考察比较多,用什么数据结构去存数据,去读取数据,都是很重要的。classSolution{private:unordered_map>targets;boolbacktracking(intticketNum,vector&result){//1.确定参数和返回值//2
- Faiss:高效相似性搜索与聚类的利器
网络·魚
大数据faiss
Faiss是一个针对大规模向量集合的相似性搜索库,由FacebookAIResearch开发。它提供了一系列高效的算法和数据结构,用于加速向量之间的相似性搜索,特别是在大规模数据集上。本文将介绍Faiss的原理、核心功能以及如何在实际项目中使用它。Faiss原理:近似最近邻搜索:Faiss的核心功能之一是近似最近邻搜索,它能够高效地在大规模数据集中找到与给定查询向量最相似的向量。这种搜索是近似的,
- insert into select 主键自增_mybatis拦截器实现主键自动生成
weixin_39521651
insertintoselect主键自增mybatisdelete返回值mybatisinsert返回主键mybatisinsert返回对象mybatisplusinsert返回主键mybatisplus插入生成id
前言前阵子和朋友聊天,他说他们项目有个需求,要实现主键自动生成,不想每次新增的时候,都手动设置主键。于是我就问他,那你们数据库表设置主键自动递增不就得了。他的回答是他们项目目前的id都是采用雪花算法来生成,因此为了项目稳定性,不会切换id的生成方式。朋友问我有没有什么实现思路,他们公司的orm框架是mybatis,我就建议他说,不然让你老大把mybatis切换成mybatis-plus。mybat
- k均值聚类算法考试例题_k均值算法(k均值聚类算法计算题)
寻找你83497
k均值聚类算法考试例题
?算法:第一步:选K个初始聚类中心,z1(1),z2(1),…,zK(1),其中括号内的序号为寻找聚类中心的迭代运算的次序号。聚类中心的向量值可任意设定,例如可选开始的K个.k均值聚类:---------一种硬聚类算法,隶属度只有两个取值0或1,提出的基本根据是“类内误差平方和最小化”准则;模糊的c均值聚类算法:--------一种模糊聚类算法,是.K均值聚类算法是先随机选取K个对象作为初始的聚类
- 关于Mysql 中 Row size too large (> 8126) 错误的解决和理解
秋刀prince
mysqlmysql数据库
提示:啰嗦一嘴,数据库的任何操作和验证前,一定要记得先备份!!!不会有错;文章目录问题发现一、问题导致的可能原因1、页大小2、行格式2.1compact格式2.2Redundant格式2.3Dynamic格式2.4Compressed格式3、BLOB和TEXT列二、解决办法1、修改页大小(不推荐)2、修改行格式3、修改数据类型为BLOB和TEXT列4、其他优化方式(可以参考使用)4.1合理设置数据
- Python实现简单的机器学习算法
master_chenchengg
pythonpython办公效率python开发IT
Python实现简单的机器学习算法开篇:初探机器学习的奇妙之旅搭建环境:一切从安装开始必备工具箱第一步:安装Anaconda和JupyterNotebook小贴士:如何配置Python环境变量算法初体验:从零开始的Python机器学习线性回归:让数据说话数据准备:从哪里找数据编码实战:Python实现线性回归模型评估:如何判断模型好坏逻辑回归:从分类开始理论入门:什么是逻辑回归代码实现:使用skl
- 推荐算法_隐语义-梯度下降
_feivirus_
算法机器学习和数学推荐算法机器学习隐语义
importnumpyasnp1.模型实现"""inputrate_matrix:M行N列的评分矩阵,值为P*Q.P:初始化用户特征矩阵M*K.Q:初始化物品特征矩阵K*N.latent_feature_cnt:隐特征的向量个数max_iteration:最大迭代次数alpha:步长lamda:正则化系数output分解之后的P和Q"""defLFM_grad_desc(rate_matrix,l
- K近邻算法_分类鸢尾花数据集
_feivirus_
算法机器学习和数学分类机器学习K近邻
importnumpyasnpimportpandasaspdfromsklearn.datasetsimportload_irisfromsklearn.model_selectionimporttrain_test_splitfromsklearn.metricsimportaccuracy_score1.数据预处理iris=load_iris()df=pd.DataFrame(data=ir
- 数据结构 | 栈和队列
TT-Kun
数据结构与算法数据结构栈队列C语言
文章目录栈和队列1.栈:后进先出(LIFO)的数据结构1.1概念与结构1.2栈的实现2.队列:先进先出(FIFO)的数据结构2.1概念与结构2.2队列的实现3.栈和队列算法题3.1有效的括号3.2用队列实现栈3.3用栈实现队列3.4设计循环队列结论栈和队列在计算机科学中,栈和队列是两种基本且重要的数据结构,它们在处理数据存储和访问顺序方面有着独特的规则和应用。本文将详细介绍栈和队列的概念、结构、实
- 系统架构设计师 需求分析篇二
AmHardy
软件架构设计师系统架构需求分析面向对象分析分析模型UML和SysML
面向对象分析方法1.用例模型构建用例模型一般需要经历4个阶段:识别参与者:识别与系统交互的所有事物。合并需求获得用例:将需求分配给予其相关的参与者。细化用例描述:详细描述每个用例的功能。调整用例模型:优化用例之间的关系和结构,前三个阶段是必需的。2.用例图的三元素参与者:使用系统的用户或其他外部系统和设备。用例:系统所提供的服务。通信关联:参与者和用例之间的关系,或用例与用例之间的关系。3.识别参
- [Python] 数据结构 详解及代码
AIAdvocate
算法python数据结构链表
今日内容大纲介绍数据结构介绍列表链表1.数据结构和算法简介程序大白话翻译,程序=数据结构+算法数据结构指的是存储,组织数据的方式.算法指的是为了解决实际业务问题而思考思路和方法,就叫:算法.2.算法的5大特性介绍算法具有独立性算法是解决问题的思路和方式,最重要的是思维,而不是语言,其(算法)可以通过多种语言进行演绎.5大特性有输入,需要传入1或者多个参数有输出,需要返回1个或者多个结果有穷性,执行
- matlab mle 优化,MLE+: Matlab Toolbox for Integrated Modeling, Control and Optimization for Buildings...
Simon Zhong
matlabmle优化
摘要:FollowingunilateralopticnervesectioninadultPVGhoodedrat,theaxonguidancecueephrin-A2isup-regulatedincaudalbutnotrostralsuperiorcolliculus(SC)andtheEphA5receptorisdown-regulatedinaxotomisedretinalgan
- Python算法L5:贪心算法
小熊同学哦
Python算法算法python贪心算法
Python贪心算法简介目录Python贪心算法简介贪心算法的基本步骤贪心算法的适用场景经典贪心算法问题1.**零钱兑换问题**2.**区间调度问题**3.**背包问题**贪心算法的优缺点优点:缺点:结语贪心算法(GreedyAlgorithm)是一种在每一步选择中都采取当前最优或最优解的算法。它的核心思想是,在保证每一步局部最优的情况下,希望通过贪心选择达到全局最优解。虽然贪心算法并不总能得到全
- Android应用性能优化
轻口味
Android
Android手机由于其本身的后台机制和硬件特点,性能上一直被诟病,所以软件开发者对软件本身的性能优化就显得尤为重要;本文将对Android开发过程中性能优化的各个方面做一个回顾与总结。Cache优化ListView缓存:ListView中有一个回收器,Item滑出界面的时候View会回收到这里,需要显示新的Item的时候,就尽量重用回收器里面的View;每次在getView函数中inflate新
- 《 C++ 修炼全景指南:九 》打破编程瓶颈!掌握二叉搜索树的高效实现与技巧
Lenyiin
C++修炼全景指南技术指南c++算法stl
摘要本文详细探讨了二叉搜索树(BinarySearchTree,BST)的核心概念和技术细节,包括插入、查找、删除、遍历等基本操作,并结合实际代码演示了如何实现这些功能。文章深入分析了二叉搜索树的性能优势及其时间复杂度,同时介绍了前驱、后继的查找方法等高级功能。通过自定义实现的二叉搜索树类,读者能够掌握其实际应用,此外,文章还建议进一步扩展为平衡树(如AVL树、红黑树)以优化极端情况下的性能退化。
- 深入浅出 -- 系统架构之负载均衡Nginx的性能优化
xiaoli8748_软件开发
系统架构系统架构负载均衡nginx
一、Nginx性能优化到这里文章的篇幅较长了,最后再来聊一下关于Nginx的性能优化,主要就简单说说收益最高的几个优化项,在这块就不再展开叙述了,毕竟影响性能都有多方面原因导致的,比如网络、服务器硬件、操作系统、后端服务、程序自身、数据库服务等,对于性能调优比较感兴趣的可以参考之前《JVM性能调优》中的调优思想。优化一:打开长连接配置通常Nginx作为代理服务,负责分发客户端的请求,那么建议开启H
- 【RabbitMQ 项目】服务端:数据管理模块之绑定管理
月夜星辉雪
rabbitmq分布式
文章目录一.编写思路二.代码实践一.编写思路定义绑定信息类交换机名称队列名称绑定关键字:交换机的路由交换算法中会用到没有是否持久化的标志,因为绑定是否持久化取决于交换机和队列是否持久化,只有它们都持久化时绑定才需要持久化。绑定就好像一根绳子,两端连接着交换机和队列,当一方不存在,它就没有存在的必要了定义绑定持久化类构造函数:如果数据库文件不存在则创建,打开数据库,创建binding_table插入
- 非对称加密算法原理与应用2——RSA私钥加密文件
私语茶馆
云部署与开发架构及产品灵感记录RSA2048私钥加密
作者:私语茶馆1.相关章节(1)非对称加密算法原理与应用1——秘钥的生成-CSDN博客第一章节讲述的是创建秘钥对,并将公钥和私钥导出为文件格式存储。本章节继续讲如何利用私钥加密内容,包括从密钥库或文件中读取私钥,并用RSA算法加密文件和String。2.私钥加密的概述本文主要基于第一章节的RSA2048bit的非对称加密算法讲述如何利用私钥加密文件。这种加密后的文件,只能由该私钥对应的公钥来解密。
- 粒子群优化 (PSO) 在三维正弦波函数中的应用
subject625Ruben
机器学习人工智能matlab算法
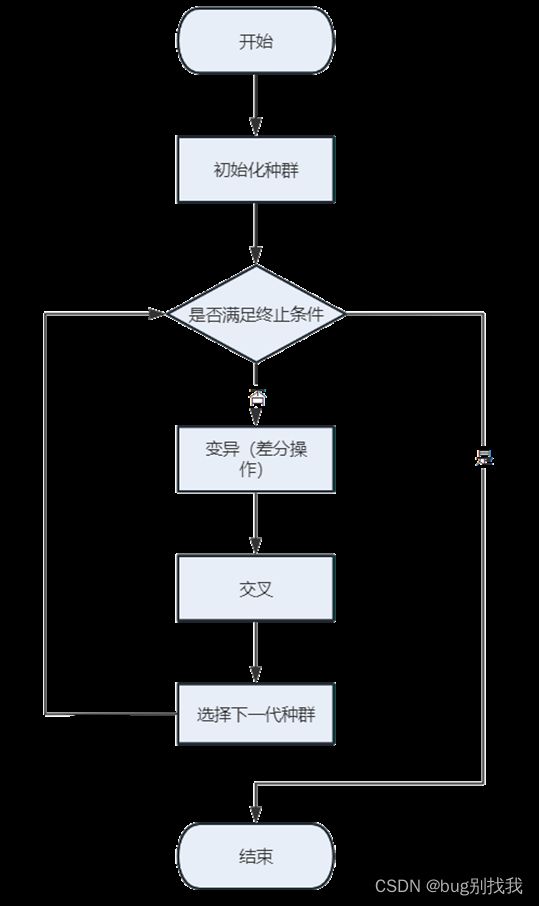
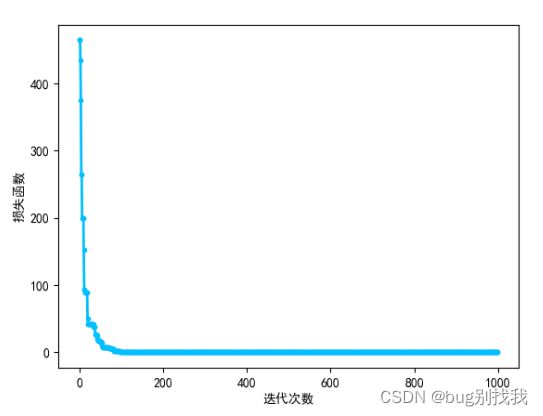
在这篇博客中,我们将展示如何使用粒子群优化(PSO)算法求解三维正弦波函数,并通过增加正弦波扰动,使优化过程更加复杂和有趣。本文将介绍目标函数的定义、PSO参数设置以及算法执行的详细过程,并展示搜索空间中的动态过程和收敛曲线。1.目标函数定义我们使用的目标函数是一个三维正弦波函数,定义如下:objectiveFunc=@(x)sin(sqrt(x(1).^2+x(2).^2))+0.5*sin(5
- 补充元象二面
Redstone Monstrosity
前端面试
1.请尽可能详细地说明,防抖和节流的区别,应用场景?你的回答中不要写出示例代码。防抖(Debounce)和节流(Throttle)是两种常用的前端性能优化技术,它们的主要区别在于如何处理高频事件的触发。以下是防抖和节流的区别和应用场景的详细说明:防抖和节流的定义防抖:在一段时间内,多次执行变为只执行最后一次。防抖的原理是,当事件被触发后,设置一个延迟定时器。如果在这个延迟时间内事件再次被触发,则重
- 自动写论文的网站推荐这5款实用类工具
小猪包333
写论文人工智能深度学习计算机视觉AI写作
在当今学术研究和写作领域,AI论文写作工具的出现极大地提高了写作效率和质量。这些工具不仅能够帮助研究人员快速生成论文草稿,还能进行内容优化、查重和排版等操作。以下是五款实用类工具推荐,特别是千笔-AIPassPaper。1.千笔-AIPassPaper千笔-AIPassPaper是一款功能强大且全面的AI论文写作助手,用户只需输入基本的研究需求和关键词,便能迅速生成一篇完整的论文。该工具利用先进的
- 推荐3家毕业AI论文可五分钟一键生成!文末附免费教程!
小猪包333
写论文人工智能AI写作深度学习计算机视觉
在当前的学术研究和写作领域,AI论文生成器已经成为许多研究人员和学生的重要工具。这些工具不仅能够帮助用户快速生成高质量的论文内容,还能进行内容优化、查重和排版等操作。以下是三款值得推荐的AI论文生成器:千笔-AIPassPaper、懒人论文以及AIPaperPass。千笔-AIPassPaper千笔-AIPassPaper是一款基于深度学习和自然语言处理技术的AI写作助手,旨在帮助用户快速生成高质
- AI论文写作推荐哪个好?分享5款AI论文写作带数据图表网站
小猪包333
写论文人工智能深度学习计算机视觉
在当今学术研究和写作领域,AI论文写作工具的出现极大地提高了写作效率和质量。这些工具不仅能够帮助研究人员快速生成论文草稿,还能进行内容优化、查重和排版等操作。以下是五款推荐的AI论文写作工具,包括千笔-AIPassPaper。千笔-AIPassPaper千笔-AIPassPaper是一款功能强大的AI论文写作助手,旨在帮助用户快速生成高质量的论文内容。AI论文,免费大纲,10分钟3万字https:
- MyBatis 详解
阿贾克斯的黎明
javamybatis
目录目录一、MyBatis是什么二、为什么使用MyBatis(一)灵活性高(二)性能优化(三)易于维护三、怎么用MyBatis(一)添加依赖(二)配置MyBatis(三)创建实体类和接口(四)使用MyBatis一、MyBatis是什么MyBatis是一个优秀的持久层框架,它支持自定义SQL、存储过程以及高级映射。MyBatis免除了几乎所有的JDBC代码以及设置参数和获取结果集的工作。它可以通过简
- mysql主从数据同步
林鹤霄
mysql主从数据同步
配置mysql5.5主从服务器(转)
教程开始:一、安装MySQL
说明:在两台MySQL服务器192.168.21.169和192.168.21.168上分别进行如下操作,安装MySQL 5.5.22
二、配置MySQL主服务器(192.168.21.169)mysql -uroot -p &nb
- oracle学习笔记
caoyong
oracle
1、ORACLE的安装
a>、ORACLE的版本
8i,9i : i是internet
10g,11g : grid (网格)
12c : cloud (云计算)
b>、10g不支持win7
&
- 数据库,SQL零基础入门
天子之骄
sql数据库入门基本术语
数据库,SQL零基础入门
做网站肯定离不开数据库,本人之前没怎么具体接触SQL,这几天起早贪黑得各种入门,恶补脑洞。一些具体的知识点,可以让小白不再迷茫的术语,拿来与大家分享。
数据库,永久数据的一个或多个大型结构化集合,通常与更新和查询数据的软件相关
- pom.xml
一炮送你回车库
pom.xml
1、一级元素dependencies是可以被子项目继承的
2、一级元素dependencyManagement是定义该项目群里jar包版本号的,通常和一级元素properties一起使用,既然有继承,也肯定有一级元素modules来定义子元素
3、父项目里的一级元素<modules>
<module>lcas-admin-war</module>
<
- sql查地区省市县
3213213333332132
sqlmysql
-- db_yhm_city
SELECT * FROM db_yhm_city WHERE class_parent_id = 1 -- 海南 class_id = 9 港、奥、台 class_id = 33、34、35
SELECT * FROM db_yhm_city WHERE class_parent_id =169
SELECT d1.cla
- 关于监听器那些让人头疼的事
宝剑锋梅花香
画图板监听器鼠标监听器
本人初学JAVA,对于界面开发我只能说有点蛋疼,用JAVA来做界面的话确实需要一定的耐心(不使用插件,就算使用插件的话也没好多少)既然Java提供了界面开发,老师又要求做,只能硬着头皮上啦。但是监听器还真是个难懂的地方,我是上了几次课才略微搞懂了些。
- JAVA的遍历MAP
darkranger
map
Java Map遍历方式的选择
1. 阐述
对于Java中Map的遍历方式,很多文章都推荐使用entrySet,认为其比keySet的效率高很多。理由是:entrySet方法一次拿到所有key和value的集合;而keySet拿到的只是key的集合,针对每个key,都要去Map中额外查找一次value,从而降低了总体效率。那么实际情况如何呢?
为了解遍历性能的真实差距,包括在遍历ke
- POJ 2312 Battle City 优先多列+bfs
aijuans
搜索
来源:http://poj.org/problem?id=2312
题意:题目背景就是小时候玩的坦克大战,求从起点到终点最少需要多少步。已知S和R是不能走得,E是空的,可以走,B是砖,只有打掉后才可以通过。
思路:很容易看出来这是一道广搜的题目,但是因为走E和走B所需要的时间不一样,因此不能用普通的队列存点。因为对于走B来说,要先打掉砖才能通过,所以我们可以理解为走B需要两步,而走E是指需要1
- Hibernate与Jpa的关系,终于弄懂
avords
javaHibernate数据库jpa
我知道Jpa是一种规范,而Hibernate是它的一种实现。除了Hibernate,还有EclipseLink(曾经的toplink),OpenJPA等可供选择,所以使用Jpa的一个好处是,可以更换实现而不必改动太多代码。
在play中定义Model时,使用的是jpa的annotations,比如javax.persistence.Entity, Table, Column, OneToMany
- 酸爽的console.log
bee1314
console
在前端的开发中,console.log那是开发必备啊,简直直观。通过写小函数,组合大功能。更容易测试。但是在打版本时,就要删除console.log,打完版本进入开发状态又要添加,真不够爽。重复劳动太多。所以可以做些简单地封装,方便开发和上线。
/**
* log.js hufeng
* The safe wrapper for `console.xxx` functions
*
- 哈佛教授:穷人和过于忙碌的人有一个共同思维特质
bijian1013
时间管理励志人生穷人过于忙碌
一个跨学科团队今年完成了一项对资源稀缺状况下人的思维方式的研究,结论是:穷人和过于忙碌的人有一个共同思维特质,即注意力被稀缺资源过分占据,引起认知和判断力的全面下降。这项研究是心理学、行为经济学和政策研究学者协作的典范。
这个研究源于穆来纳森对自己拖延症的憎恨。他7岁从印度移民美国,很快就如鱼得水,哈佛毕业
- other operate
征客丶
OSosx
一、Mac Finder 设置排序方式,预览栏 在显示-》查看显示选项中
二、有时预览显示时,卡死在那,有可能是一些临时文件夹被删除了,如:/private/tmp[有待验证]
--------------------------------------------------------------------
若有其他凝问或文中有错误,请及时向我指出,
我好及时改正,同时也让我们一
- 【Scala五】分析Spark源代码总结的Scala语法三
bit1129
scala
1. If语句作为表达式
val properties = if (jobIdToActiveJob.contains(jobId)) {
jobIdToActiveJob(stage.jobId).properties
} else {
// this stage will be assigned to "default" po
- ZooKeeper 入门
BlueSkator
中间件zk
ZooKeeper是一个高可用的分布式数据管理与系统协调框架。基于对Paxos算法的实现,使该框架保证了分布式环境中数据的强一致性,也正是基于这样的特性,使得ZooKeeper解决很多分布式问题。网上对ZK的应用场景也有不少介绍,本文将结合作者身边的项目例子,系统地对ZK的应用场景进行一个分门归类的介绍。
值得注意的是,ZK并非天生就是为这些应用场景设计的,都是后来众多开发者根据其框架的特性,利
- MySQL取得当前时间的函数是什么 格式化日期的函数是什么
BreakingBad
mysqlDate
取得当前时间用 now() 就行。
在数据库中格式化时间 用DATE_FORMA T(date, format) .
根据格式串format 格式化日期或日期和时间值date,返回结果串。
可用DATE_FORMAT( ) 来格式化DATE 或DATETIME 值,以便得到所希望的格式。根据format字符串格式化date值:
%S, %s 两位数字形式的秒( 00,01,
- 读《研磨设计模式》-代码笔记-组合模式
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
import java.util.ArrayList;
import java.util.List;
abstract class Component {
public abstract void printStruct(Str
- 4_JAVA+Oracle面试题(有答案)
chenke
oracle
基础测试题
卷面上不能出现任何的涂写文字,所有的答案要求写在答题纸上,考卷不得带走。
选择题
1、 What will happen when you attempt to compile and run the following code? (3)
public class Static {
static {
int x = 5; // 在static内有效
}
st
- 新一代工作流系统设计目标
comsci
工作算法脚本
用户只需要给工作流系统制定若干个需求,流程系统根据需求,并结合事先输入的组织机构和权限结构,调用若干算法,在流程展示版面上面显示出系统自动生成的流程图,然后由用户根据实际情况对该流程图进行微调,直到满意为止,流程在运行过程中,系统和用户可以根据情况对流程进行实时的调整,包括拓扑结构的调整,权限的调整,内置脚本的调整。。。。。
在这个设计中,最难的地方是系统根据什么来生成流
- oracle 行链接与行迁移
daizj
oracle行迁移
表里的一行对于一个数据块太大的情况有二种(一行在一个数据块里放不下)
第一种情况:
INSERT的时候,INSERT时候行的大小就超一个块的大小。Oracle把这行的数据存储在一连串的数据块里(Oracle Stores the data for the row in a chain of data blocks),这种情况称为行链接(Row Chain),一般不可避免(除非使用更大的数据
- [JShop]开源电子商务系统jshop的系统缓存实现
dinguangx
jshop电子商务
前言
jeeshop中通过SystemManager管理了大量的缓存数据,来提升系统的性能,但这些缓存数据全部都是存放于内存中的,无法满足特定场景的数据更新(如集群环境)。JShop对jeeshop的缓存机制进行了扩展,提供CacheProvider来辅助SystemManager管理这些缓存数据,通过CacheProvider,可以把缓存存放在内存,ehcache,redis,memcache
- 初三全学年难记忆单词
dcj3sjt126com
englishword
several 儿子;若干
shelf 架子
knowledge 知识;学问
librarian 图书管理员
abroad 到国外,在国外
surf 冲浪
wave 浪;波浪
twice 两次;两倍
describe 描写;叙述
especially 特别;尤其
attract 吸引
prize 奖品;奖赏
competition 比赛;竞争
event 大事;事件
O
- sphinx实践
dcj3sjt126com
sphinx
安装参考地址:http://briansnelson.com/How_to_install_Sphinx_on_Centos_Server
yum install sphinx
如果失败的话使用下面的方式安装
wget http://sphinxsearch.com/files/sphinx-2.2.9-1.rhel6.x86_64.rpm
yum loca
- JPA之JPQL(三)
frank1234
ormjpaJPQL
1 什么是JPQL
JPQL是Java Persistence Query Language的简称,可以看成是JPA中的HQL, JPQL支持各种复杂查询。
2 检索单个对象
@Test
public void querySingleObject1() {
Query query = em.createQuery("sele
- Remove Duplicates from Sorted Array II
hcx2013
remove
Follow up for "Remove Duplicates":What if duplicates are allowed at most twice?
For example,Given sorted array nums = [1,1,1,2,2,3],
Your function should return length
- Spring4新特性——Groovy Bean定义DSL
jinnianshilongnian
spring 4
Spring4新特性——泛型限定式依赖注入
Spring4新特性——核心容器的其他改进
Spring4新特性——Web开发的增强
Spring4新特性——集成Bean Validation 1.1(JSR-349)到SpringMVC
Spring4新特性——Groovy Bean定义DSL
Spring4新特性——更好的Java泛型操作API
Spring4新
- CentOS安装Mysql5.5
liuxingguome
centos
CentOS下以RPM方式安装MySQL5.5
首先卸载系统自带Mysql:
yum remove mysql mysql-server mysql-libs compat-mysql51
rm -rf /var/lib/mysql
rm /etc/my.cnf
查看是否还有mysql软件:
rpm -qa|grep mysql
去http://dev.mysql.c
- 第14章 工具函数(下)
onestopweb
函数
index.html
<!DOCTYPE html PUBLIC "-//W3C//DTD XHTML 1.0 Transitional//EN" "http://www.w3.org/TR/xhtml1/DTD/xhtml1-transitional.dtd">
<html xmlns="http://www.w3.org/
- POJ 1050
SaraWon
二维数组子矩阵最大和
POJ ACM第1050题的详细描述,请参照
http://acm.pku.edu.cn/JudgeOnline/problem?id=1050
题目意思:
给定包含有正负整型的二维数组,找出所有子矩阵的和的最大值。
如二维数组
0 -2 -7 0
9 2 -6 2
-4 1 -4 1
-1 8 0 -2
中和最大的子矩阵是
9 2
-4 1
-1 8
且最大和是15
- [5]设计模式——单例模式
tsface
java单例设计模式虚拟机
单例模式:保证一个类仅有一个实例,并提供一个访问它的全局访问点
安全的单例模式:
/*
* @(#)Singleton.java 2014-8-1
*
* Copyright 2014 XXXX, Inc. All rights reserved.
*/
package com.fiberhome.singleton;
- Java8全新打造,英语学习supertool
yangshangchuan
javasuperword闭包java8函数式编程
superword是一个Java实现的英文单词分析软件,主要研究英语单词音近形似转化规律、前缀后缀规律、词之间的相似性规律等等。Clean code、Fluent style、Java8 feature: Lambdas, Streams and Functional-style Programming。
升学考试、工作求职、充电提高,都少不了英语的身影,英语对我们来说实在太重要