EEGLAB
eeglab 代码
eeglab redraw 更新eeglab
EEG.hisotry eeglab历史代码查看
clc 清屏
Part1 准备工作
下载软件
本教程使用的Matlab版本号为R2021a,EEGLAB为2023最新版。
不建议安装版本特别古老的Matlab,至少是Matlab2016a以上的。
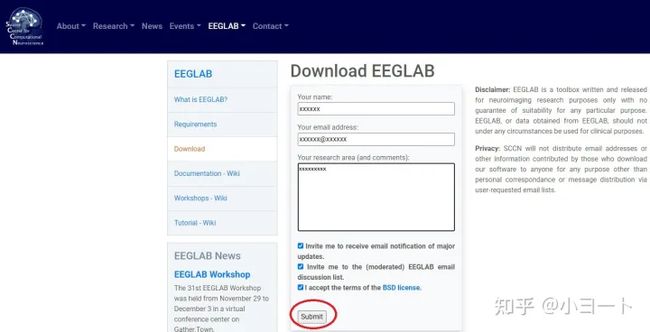
EEGLAB最新版,网址如下:
https://sccn.ucsd.edu/eeglab/download.php
把该填的信息填了,然后点击submit。
然后点击click here就可以下载最新版本的EEGLAB了。
添加路径
下载好了EEGLAB,还需要添加路径,让Matlab知道EEGLAB工具包在哪里,才能顺利的开展后续工作。
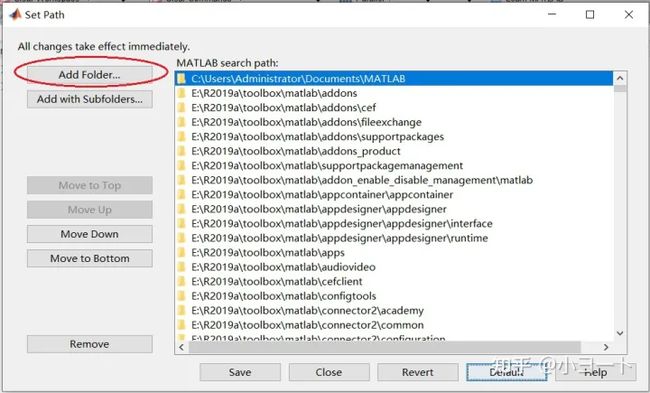
添加路径,选择Matlab菜单栏上面的Set Path。
点击Add Folder,不要点击Add with Subfolders。
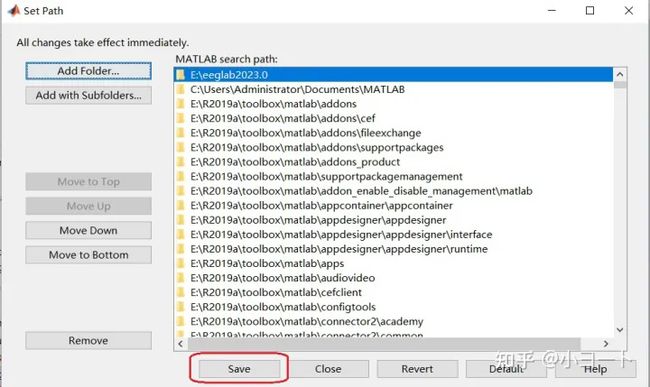
找到我们刚刚下载好并解压好的EEGLAB2023,选中eeglab2023.0,然后点下面的选择文件夹。
点击Save。
点击close关闭。
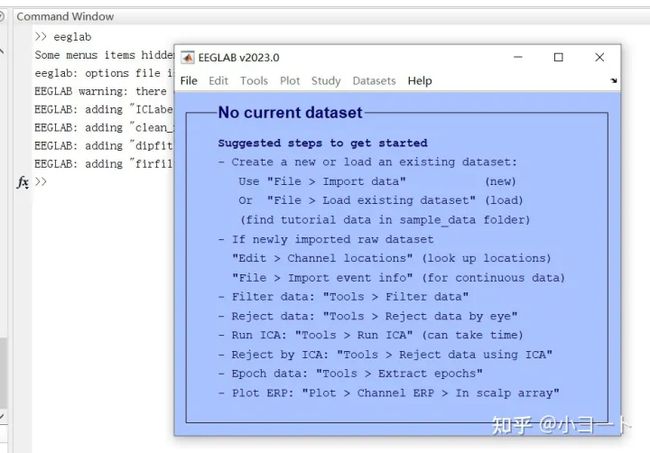
在Matlab的Command Window输入eeglab。
软件准备工作顺利完成。
示例数据准备
单被试预处理使用的示例数据为静息态EEG数据,采集软件为Neuroscan的curry8软件,采样率为1000Hz。
采集数据后的原始格式为:
导入该格式的数据到EEGLAB需要loadcurry插件,默认EEGLAB是没有安装的,此时需要安装该插件。
点击File->Manage EEGLAB extensions:
搜索curry并选择loadcurry然后点击下载。
安装其他EEGLAB默认没有带的插件一样可以通过上述方式下载
Part2 单被试EEG数据预处理
1.导入示例数据
点击如下菜单:
选择我们的样本数据并打开:
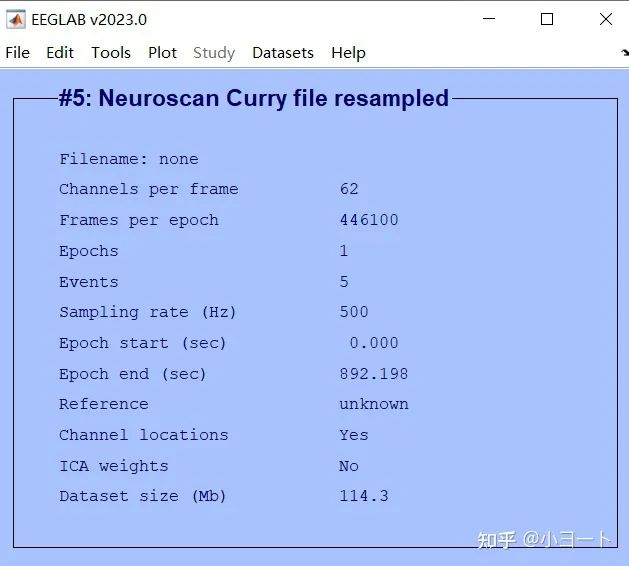
现在我们成功的导入了示例数据,EEGLAB界面如下所示:
从该界面,我们可以看到示例数据的文件名称,一共有67个channels,有892200个数据点,因为示例数据的采样率为1000Hz,表示1秒钟可以采集1000个信号点,总共采集了892秒的数据。
Reference为unknown表示我们现在还没有设定离线参考,channel location为yes表示我们采集的数据自带有电极位置信息,ICA weight为No表示我们尚未进行ICA分析。
以上菜单操作等效于以下代码:
EEG = loadcurry('D:\eeglab批处理模板\sub1.cdt', 'KeepTriggerChannel', 'True', 'CurryLocations', 'False');
2.降采样
降采样可以降低数据所占空间,我们实际分析通常用不到数据采集时那么高的精度,所以本例降采样为500Hz。
点击Tools->Change sampling rate:
输入500点击OK。
很快降采样就完成了,弹出一个窗口点击OK。
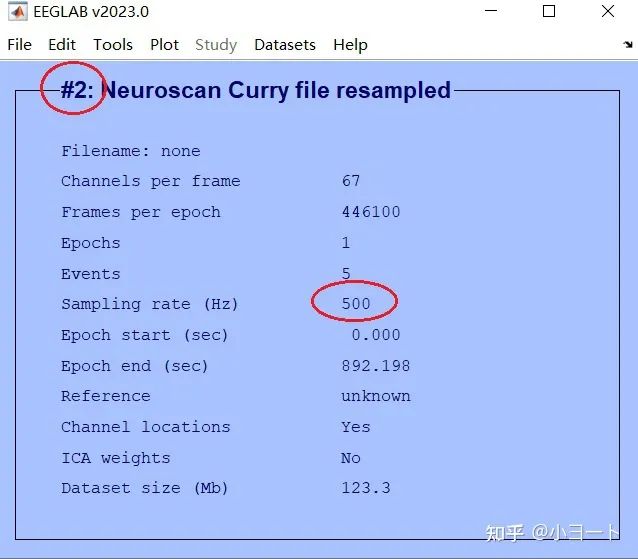
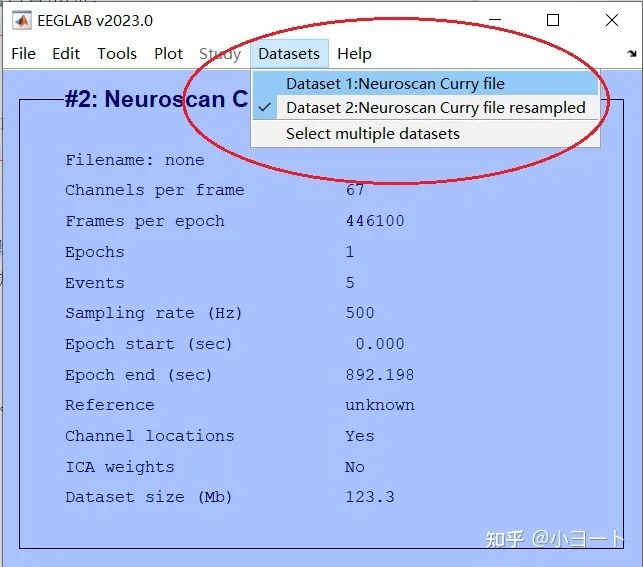
可以发现界面产生了一些变化,如下图所示:
#2是我们刚刚降采样完成的数据集,之前的数据集需要点击Datasets,再选择Dataset1才可以激活为当前数据集:
但我们不需要这么做,只需要在#2的数据集上进行下一步操作就好了。
上述为降采样的菜单操作,实际上这也可以通过代码完成。降采样为500Hz的代码为:
EEG = pop_resample( EEG, 500);
有同学会问,菜单操作多好,为啥还要学代码。这是因为代码对于批处理是必不可少的。
3.带通滤波
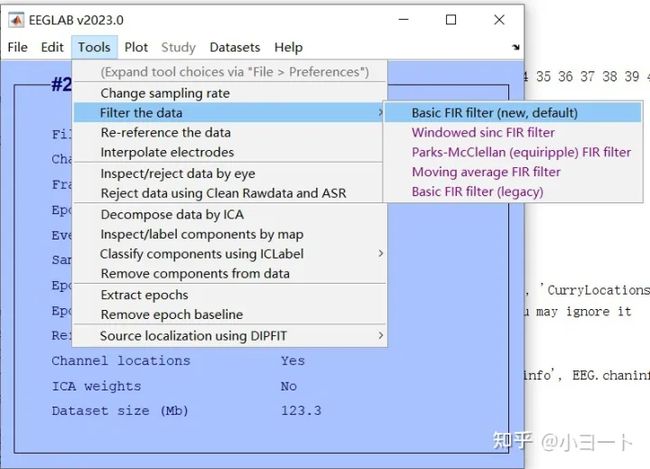
本例中我们选择带通滤波1-80Hz,点击Tools->Filter the data->Basic FIR filter:
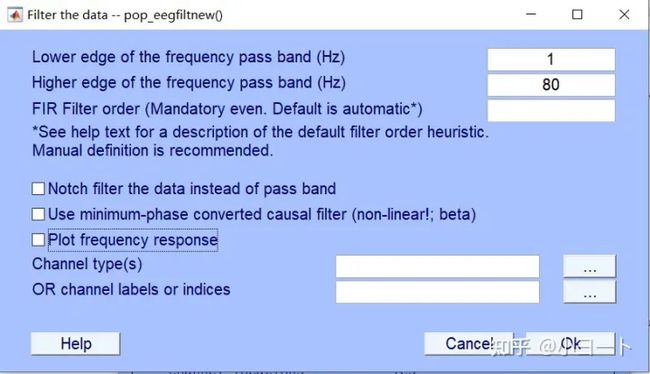
分别输入1,80,然后点击OK,在弹出的new dataset对话框一样点击OK,以下不再赘述:
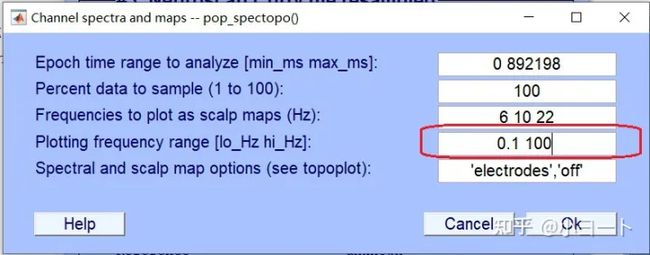
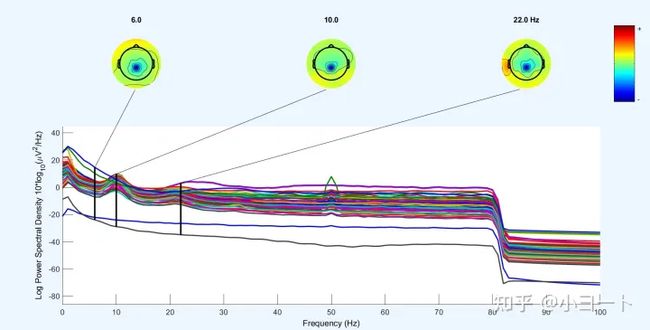
这时候我们就做完了带通滤波,当然在主界面是看不出来数据的频率范围的,我们需要点击Plot->Channel spectra and maps,然后在弹出的框里输入想要看到的频率范围如下图所示,点击OK:
稍等片刻,完成后会显示下图:
可以看到80Hz之后的功率谱密度有一个明显的衰减,那是因为我们把更高频的滤掉了。
上述带通滤波的代码为:
EEG = pop_eegfiltnew(EEG, 'locutoff',1,'hicutoff',80);
4.陷波滤波
细心的同学可以发现,在上图中50Hz的位置有一些尖尖,这是由于工频干扰所致,国内的交流电频率为50Hz。若在国外,那么就是60Hz。
为了将这个工频干扰去掉,我们可以使用陷波滤波。同样点击Tools->Filter the data->Basic FIR filter,然后如下图所示:
一定要记得在Notch前打勾。
点击OK就可完成滤掉工频。再次查看功率谱图可以看到50Hz那里的尖尖消失了,取而代之的是凹陷。
所以称之为陷波滤波真的是名副其实。
陷波滤波的代码为:
EEG = pop_eegfiltnew(EEG, 'locutoff',48,'hicutoff',52,'revfilt',1);
5.去掉无关电极
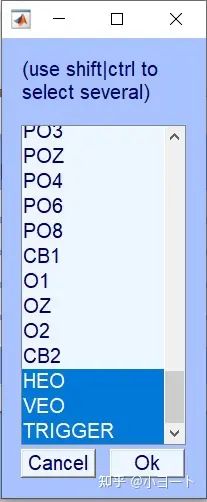
有些多余的电极需要去掉,这一步在之前做也可以。比如我们这里去掉M1、M2、HEO和VEO。点击Edit->Select data,在弹出的对话框里点击红圈按钮:
在弹出来的对话框里按住Ctrl将想要去掉的电极都选中,然后点击OK。
注意:如果想要以M1和M2平均参考的同学请不要去掉M1和M2,如果想全脑平均重参考的同学,可以去掉所有头皮之外的电极。
然后,一定要注意下图这个地方一定要打勾,不然就不是删掉这些电极,而是只剩下你想要去掉的电极了。
我们可以看到现在的电极数目变成了62个,如下图所示:
上述操作等效于以下代码:
EEG = pop_select( EEG, 'rmchannel',{'M1','M2','HEO','VEO','TRIGGER'});
6.检查有无坏导坏段
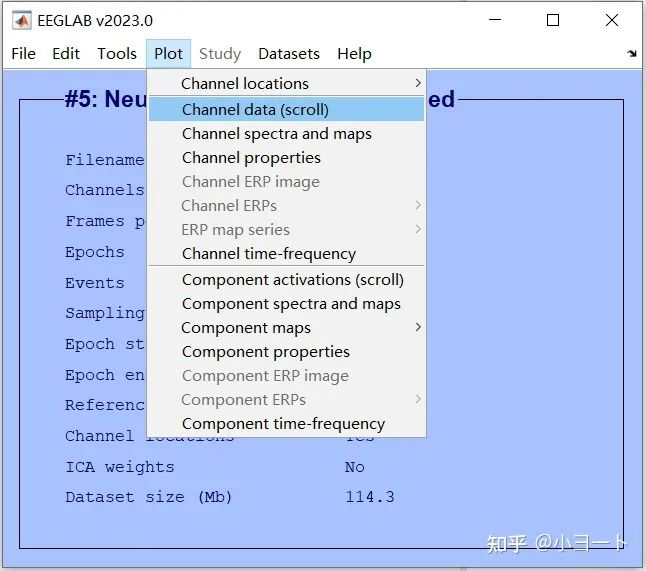
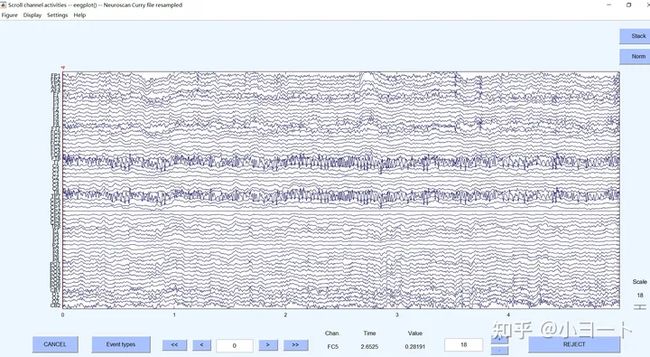
可以点击Plot->Channel data,就可以看到我们采集的脑电图:
菜单栏的Setting->Time range to display可以调整一个屏幕所显示的时间,默认是5秒。而Setting->Number of channels to display可以调整显示的电极数目。如果导联数太多,那么可能看不清楚,可以设置这个参数小一点即可。
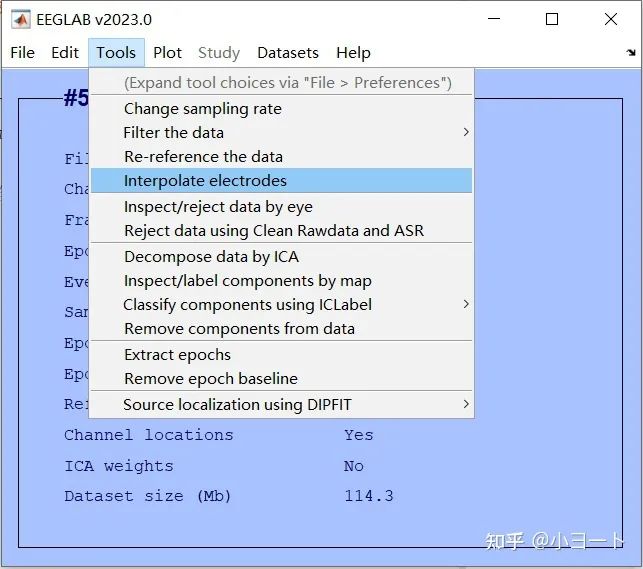
我们例子数据可以看到T7和TP7这两个电极的波形似乎与众不同,那么可以考虑把它插值。关掉图形界面,点击Tools->interpolate electrodes:
在弹出的对话框点击Select from data channels,然后选择T7和TP7这两个电极(注意选多个电极要按Ctrl),点击OK。
再次打开图形界面就看到那两个乱糟糟的电极被插值掉了。
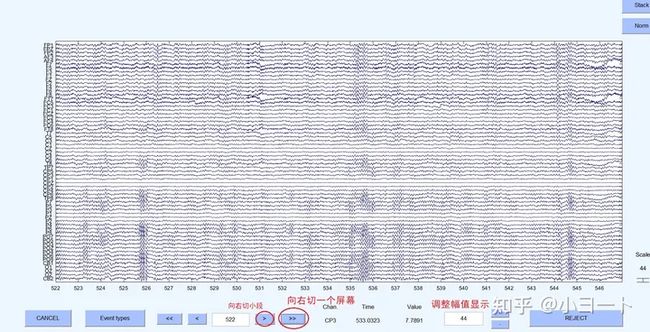
接下来浏览所有时间线的脑电图,发现有无坏段,如果有坏段,就把它标记并删掉,如果没有坏段,那么就不用做任何操作。在浏览的时候,可以适当把一个屏幕的时间调的稍微大点,不然默认的5s一个屏幕要看很长时间。点击下图所示菜单往后浏览,必要时调整幅值。
若发现坏段,那么按住鼠标左键,拖动鼠标标记坏段,到坏段结束时松开鼠标,这时并没有把它删掉,只是作为标记,先标记完所有坏段,最后点击右下角的REJECT才会把它们删掉:
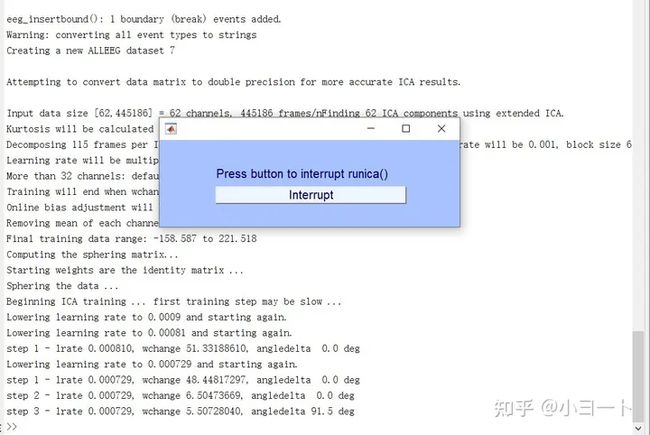
7.跑ICA
这一步一般来说是比较耗时的步骤。那么为啥要跑ICA?
我的理解是为了在不损失数据的前提下把相应成分的伪迹给剥离出来。
当然,如果伪迹很少,直接像去坏段那样把它标记并去掉,也未尝不可。但是某些伪迹几乎贯穿整个脑电时间线,全部把它暴力删除那么我们就没有什么数据可以进行分析了。
所以还是要跑ICA。
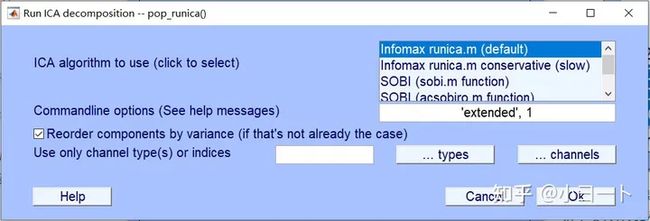
点击Tools->Decompose data by ICA,在弹出的对话框里默认参数不用更改,点击OK即可。
已经开始ICA了,耐心等待。
上述菜单操作等效于以下代码:
EEG = pop_runica(EEG, 'icatype', 'runica', 'extended',1,'interrupt','on');
当ICA跑完后,我们的界面会变成如下所示:
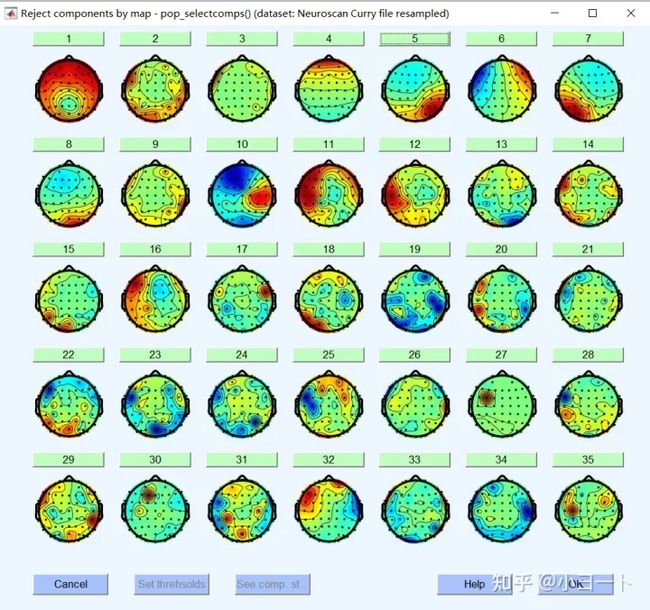
那么现在我们来查看一下跑出来的ICA成分,点击Tools->Inspect/label components by map,然后弹出来的对话框点击OK,然后再点OK(超过35个ICA成分会让你再确认一遍),然后会看到:
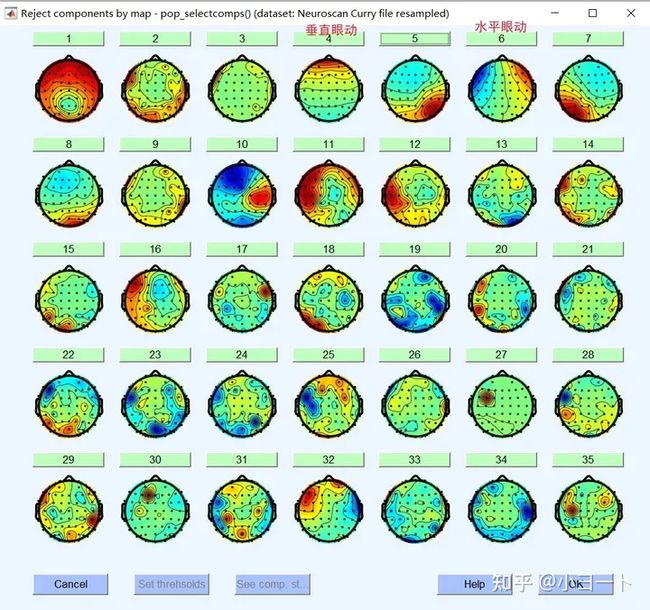
对于这个例子数据,一眼就看出来水平眼动和垂直眼动:
那么啥叫水平眼动和垂直眼动?
水平眼动就是左右看,垂直眼动就是眨眼。我们分别打开ICA成分看看。
水平眼动
垂直眼动
这个被试做的静息态脑电采集,前半时间是睁眼,后半时间是闭眼。所以前半时间水平眼动和垂直眼动都有,但后半时间就几乎没有垂直眼动了。
有了这些知识,相信你判断眼动ICA成分的后验概率提高了不少。但是眼动ICA成分有时候不一定能很好的被提取出来,比如你的数据很糟糕,采集时间很短,伪迹成分过多等等。
那么其他成分呢?还有那么多!
要是只有60个ICA成分,那么一个一个的点开看还好;那如果你用的是128个电极、256个电极呢?看一个被试就要鼠标点256次?前提是你得会分辨每一个成分是不是伪迹。让你处理100个被试你会怎么样?
难以想象,脑电数据的预处理真的很痛苦吗?
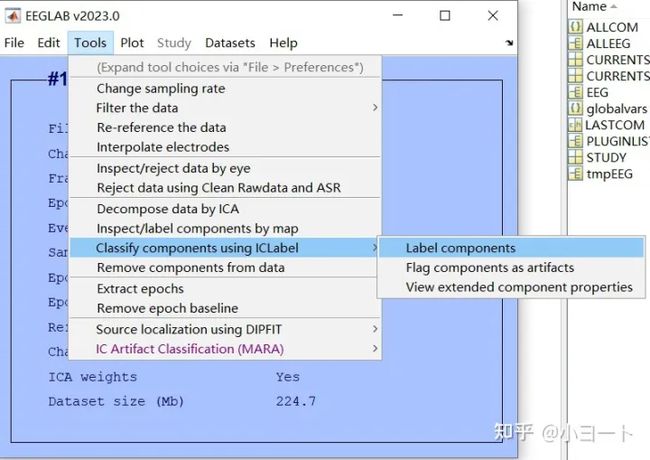
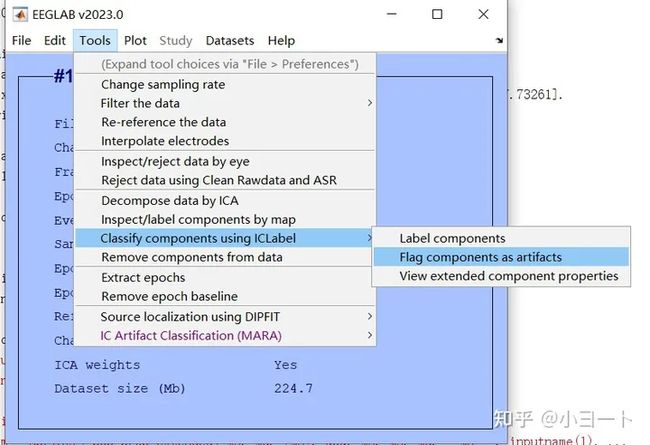
不要慌,现在有一些基于某些算法的插件可以自动给出判断是否是伪迹,甚至是什么伪迹的概率,比如ICLabel这个插件。我们马上开始用一下,点击Tools->Classify components using ICLabel->Label components,如下所示:
还弹出了一个对话框,点击OK;
ICLabel: extracting features...
ICLabel: calculating labels...
ICLabel: saving results...
Done.
很快计算完成,下图继续点击OK。
然后会看到:
可以看到ICLabel可以给出是什么ICA成分的概率,比如我们刚刚看到的水平眼动和垂直眼动,分别被标记为是eye的概率为98.2%和98.3%。
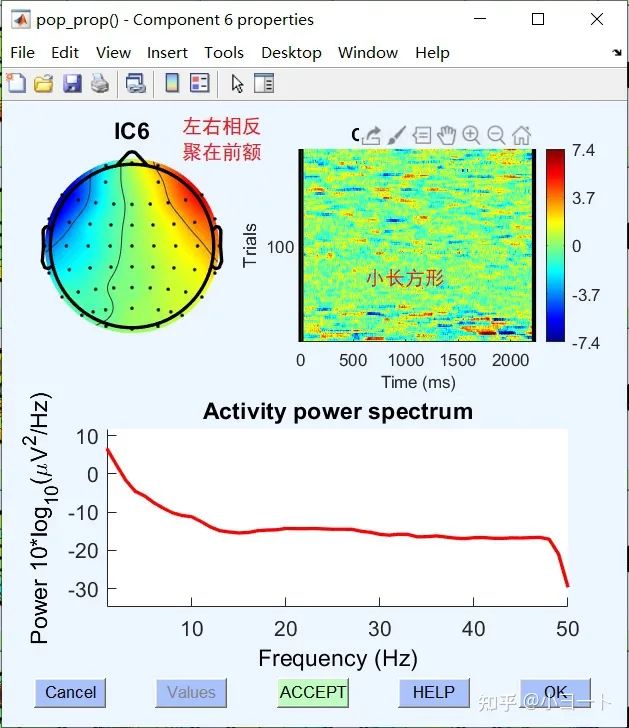
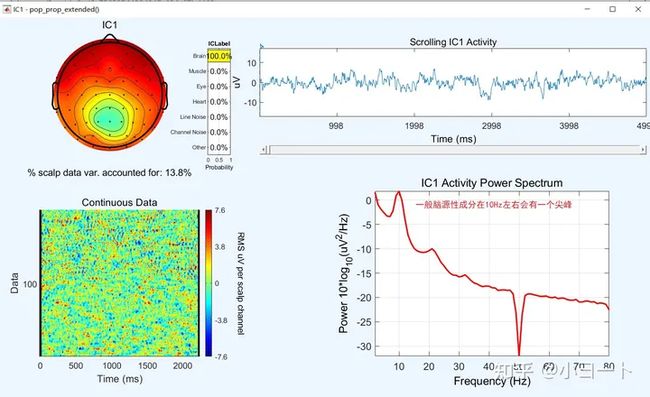
第一个ICA成分被标记为Brain的概率是100%,我们打开可以看到下图:
一般脑源性ICA成分在10Hz左右会有一个尖峰,如上图所示。
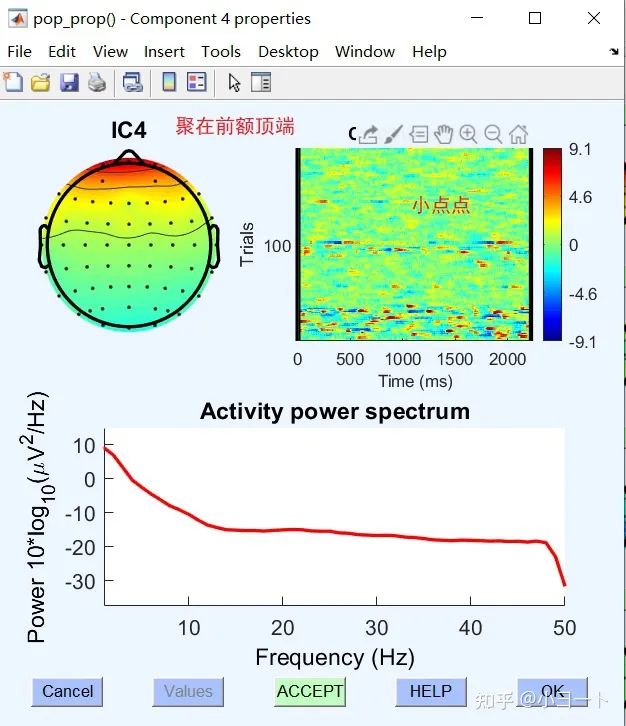
大家注意看,第46个ICA被标记为Muscle的概率为82.4%,我们打开看一下:
可以看到,在右上角的幅值-时间图像中,波峰波谷之间特别的拥挤,频率高。所以肌电信号的高频部分Power值比较高,如右下方所示。这个肌电伪迹还不是特别的典型,更加典型的右下角图像会像一个数学的根号。
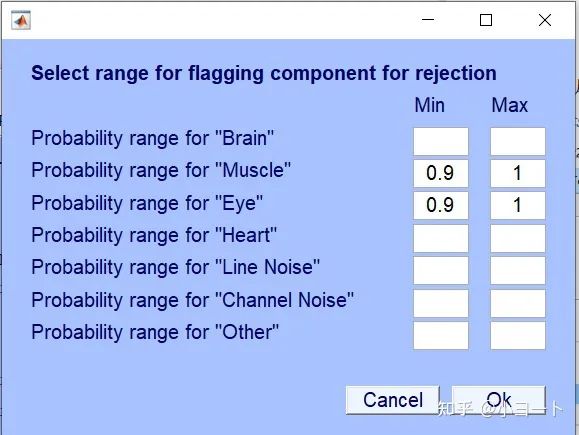
而且还可以让ICLabel自动判断并标记哪些ICA成分是伪迹,点击Tools->Classify components using ICLabel->Flag components as artifacts,如下图:
这里你可以设置一个阈值,默认是90%以上概率的眼电和肌电伪迹会被标记为伪迹准备去除,你也可以设置80%,70%都可以。设置50%也没人管你。
ICA去除没有绝对的金标准,有的人严格,有的人宽松。但有的ICA成分分离不完全,那么在去掉伪迹的时候也会去掉一定程度的脑源性信号。
在这里我们保持默认,点击OK,在MATLAB的窗口可以看到:
ICLabel: Install the viewprops eeglab plugin to create IC visualizations like these elsewhere.
IClabel class "Brain": 5/62 components at 90% threshold
IClabel class "Muscle": 0/62 components at 90% threshold
IClabel class "Eye": 2/62 components at 90% threshold
IClabel class "Heart": 0/62 components at 90% threshold
IClabel class "Line Noise": 0/62 components at 90% threshold
IClabel class "Channel Noise": 1/62 components at 90% threshold
IClabel class "Other": 10/62 components at 90% threshold
2 components flagged for rejection, to reject them use Tools > Remove components from data
Done.
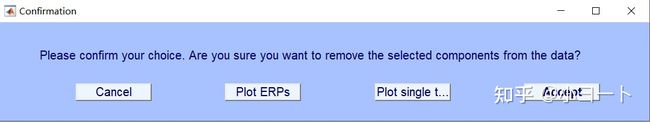
这时就有两个成分被标记为准备rejection的,我们继续点击Tools->Remove components from data。
提示我们4,6是被ICLabel自动标记为伪迹的成分,点击Yes。
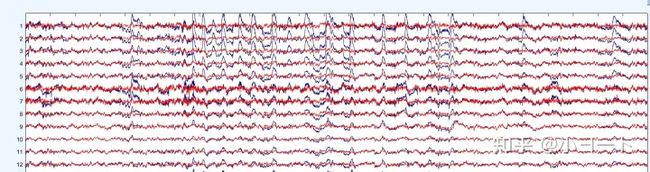
因为我们这里处理的是连续数据,所以点击Plot single trials。
红色的是我们去掉伪迹之后的,黑色的是之前的,可以看到眼电伪迹都被很好的去掉了。总而言之,去掉水平眼动、垂直眼动、肌电伪迹ICA成分的对比图,都会让我有一种极度舒适的感觉,就像成功的把信号中的灰尘擦掉一样。
把这个关掉,然后点击Accept,那么我们就成功的去掉了上述标注的ICA成分。
上述使用ICLabel识别伪迹并去掉伪迹的菜单操作,等效于以下代码:
EEG = pop_iclabel(EEG, 'default');
EEG = pop_icflag(EEG, [NaN NaN;0.9 1;0.9 1;NaN NaN;NaN NaN;NaN NaN;NaN NaN]);
EEG = pop_subcomp( EEG, [], 0)
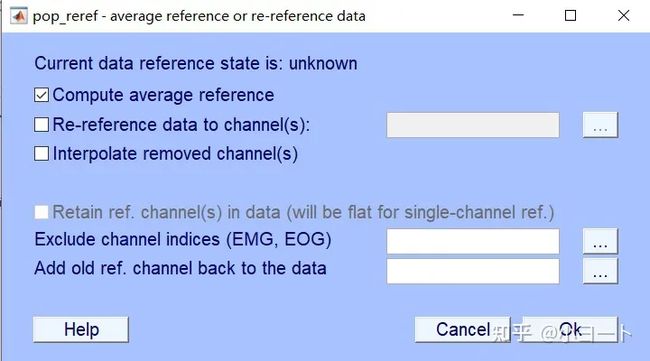
8.重参考
其实去掉ICA成分之后,我就觉得预处理已经基本结束了,什么重参考、ERP的分段都可以在后面正式数据处理分析的时候一块弄。不过很多预处理包括了重参考,这里也跟大家讲一下。
这里选择全脑平均重参考,点击Tools->Re-reference the data,在弹出来的对话框直接点击OK就行,因为默认的就是计算全脑平均重参考。
上述菜单操作等效于代码:
EEG = pop_reref( EEG, []);
Part3 多个被试的预处理-批处理
讲到这里有的同学是不是已经懵掉了,这脑电预处理怎么这么繁琐,一个被试都要点半天,100个被试可还行?
没关系,大部分工作都是可以电脑自动处理的。
做预处理的每一步,我都建议大家要保存在一个文件夹。万一哪个步骤你觉得没弄对,还可以重新从上一步再来,而不必从头再来。尤其是ICA后的文件务必保存。
那么我们首先新建一个文件夹如下图所示:
在这里我新建了4个空的文件夹:
- 文件夹_step1用于存放我们经过降采样、带通滤波、陷波滤波、去除无关电极之后的数据;
- 文件夹_preica用于存放我们经过检查有无坏段坏导之后的数据;
- 文件夹_ica用于存放我们跑完ICA之后的数据;
- 文件夹_rm_ica用于存放ICLabel自动去完伪迹以及全脑平均重参考之后的数据;
- 母文件夹demo下存放原始采集的数据。
准备完成之后,运行以下代码:
clc;clear;
%% Specify Basic information of different groups 将cdt文件转换成set文件,降采样为500,去除无用电极,保存。
group_dir = 'D:\demo'; % 此处路径需要设置为自己的文件目录
group_files = dir([group_dir, filesep, '*.cdt']); %filesep是\的意思
for i=1:length(group_files)
subj_fn = group_files(i).name;
EEG = loadcurry(strcat(group_dir, filesep, subj_fn), 'CurryLocations', 'False'); %导入原始数据
EEG = pop_resample( EEG, 500); %降采样
EEG = pop_eegfiltnew(EEG, 'locutoff',1,'hicutoff',80); %带通滤波
EEG = pop_eegfiltnew(EEG, 'locutoff',48,'hicutoff',52,'revfilt',1); %陷波滤波
EEG = pop_select( EEG, 'rmchannel',{'M1','M2','HEO','VEO','TRIGGER'}); %去除无关电极
EEG = pop_saveset( EEG, 'filename',strcat(group_files(i).name(1:end-4), '.set'), 'filepath',strcat(group_dir, filesep, '_step1')); %注意需要在运行代码之前,文件目录下建一个_resam_remch的文件夹,以下雷同
end
菜单操作:打开eeglab导入_step1的文件,看有无坏导坏段,若有坏导则插值;若有坏段则删掉。完了之后保存数据到_preica文件夹,文件名称保持不变。
- 文件导入:打开eeglab,点击File->load existing dataset;
- 文件保存:点击File->save current dataset as。
接着继续代码:
%% 运行 ICA
group1_dir = 'D:\demo'; % 此处路径需要设置为自己的文件目录
group1_dir1 = 'D:\demo\_preica'; % 此处路径需要设置为自己的文件目录
group1_files = dir([group1_dir1, filesep, '*.set']); %filesep是\的意思
for i=1:length(group1_files)
subj_fn = group1_files(i).name;
EEG = pop_loadset('filename',strcat(subj_fn(1:end-4), '.set'), 'filepath', strcat(group1_dir, filesep, '_preica')); %导入数据
EEG = pop_runica(EEG, 'icatype', 'runica', 'extended',1,'interrupt','on'); % 跑ICA
EEG = pop_saveset( EEG, 'filename',strcat(group1_files(i).name(1:end-4), '.set'), 'filepath',strcat(group1_dir, filesep, '_ica')); %保存数据
end
%% 使用ICLabel自动去除ICA成分
group1_dir = 'D:\demo'; % 此处路径需要设置为自己的文件目录
group1_dir2 = 'D:\demo\_ica';
group1_files = dir([group1_dir2, filesep, '*.set']); %filesep是\的意思
for i=1:length(group1_files)
subj_fn = group1_files(i).name;
EEG = pop_loadset('filename',strcat(subj_fn(1:end-4), '.set'), 'filepath', group1_dir2);
EEG = pop_iclabel(EEG, 'default');
EEG = pop_icflag(EEG, [NaN NaN;0.9 1;0.9 1;NaN NaN;NaN NaN;NaN NaN;NaN NaN]); % 标记伪迹成分。这里可以自定义设定阈值,依次为Brain, Muscle, Eye, Heart, Line Noise, Channel Noise, Other.
EEG = pop_subcomp( EEG, [], 0) %去除上述伪迹成分
EEG = pop_reref( EEG, []); %全脑平均重参考
EEG = eeg_checkset( EEG );
EEG = pop_saveset( EEG, 'filename',strcat(group1_files(i).name(1:end-4), '.set'), 'filepath',strcat(group1_dir, filesep, '_rm_ica'));
end