X-tile的使用选择最适cut-off值
数据准备:
将此数据存成txt(文本文件,制表符分隔)。
打开x-tile并点击分析:
然后File-open-选择数据导入。
Censor对应OS即生存状态,Survivaltime对应OS.time生存时间,marker1就是要研究的变量(基因表达,患者年龄,性别,分期等都可以)。
此处注意,如果随访的数据是天,就都改成Days,然后随访天数最长的加入是五千天,cutoff at写5000(加入4999天,也写5000) 
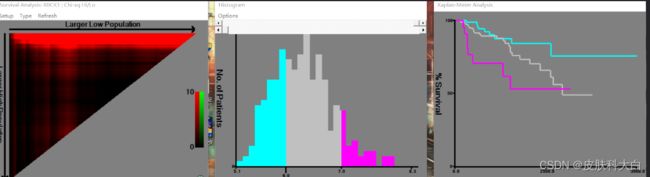
点击左上角Do-kaplan-Maier-maker1,出现如下界面:
点击红框的倒三角,能够确认最适的两个cut-off值,下面的长方形能确定最适的一个cut-off值。
我们先进行一下单因素cox回归,代码如下:
这是来自TCGA—BLCA的数据,OS.time生存时间,OS生存状态:1表示死亡,0表示存活,NSUN6是随意筛选的某个基因的表达量。下面开始测试代码:
setwd("D:\\data")
dir()
data <- read.csv("survival.csv",header = T,sep = ",")
head(data)##查看数据
> head(data)
Sample OS.time OS NSUN6
1 TCGA.2F.A9KO.01A 734 1 4.015870
2 TCGA.2F.A9KP.01A 364 1 3.922716
3 TCGA.2F.A9KQ.01A 2886 0 4.120226
4 TCGA.2F.A9KR.01A 3183 1 2.909812
5 TCGA.2F.A9KT.01A 3314 0 3.160342
6 TCGA.2F.A9KW.01A 254 1 2.2035342.对需要分析的数据进行分组,这里以NSUN6中位值为界限,高于中位值的设置为高表达组,低于中位值的设置为地表达组。当然,我们也可以用一些软件确定自变量的cut-off值,如x-tile。x-tile的使用教程将在下期分享。
data$a <- ifelse(data$NSUN6>median(data$NSUN6),"High NSUN6 expression","Low NSUN6 expression")
> head(data)
Sample OS.time OS NSUN6 a
1 TCGA.2F.A9KO.01A 734 1 4.015870 High NSUN6 expression
2 TCGA.2F.A9KP.01A 364 1 3.922716 High NSUN6 expression
3 TCGA.2F.A9KQ.01A 2886 0 4.120226 High NSUN6 expression
4 TCGA.2F.A9KR.01A 3183 1 2.909812 Low NSUN6 expression
5 TCGA.2F.A9KT.01A 3314 0 3.160342 High NSUN6 expression
6 TCGA.2F.A9KW.01A 254 1 2.203534 Low NSUN6 expression3.接下来绘制生存曲线,代码如下
library(coin)
library(survminer)
library(survival)
fit <- survfit(Surv(OS.time,OS) ~ data$a, data = data)
d <- ggsurvplot_list(fit,data = data,pval = T,##是否添加P值
conf.int = F,### 是否添加置信区间
risk.table = T, # 是否添加风险表
risk.table.col = "strata",
###linetype = "strata",
surv.median.line = "hv", # 是否添加中位生存线
risk.table.y.text.col = F,risk.table.y.text = FALSE,
ggtheme = theme_bw()+theme(legend.text = element_text(colour = c("red", "blue")))
+theme(panel.grid.major = element_blank(),panel.grid.minor = element_blank())
+theme(plot.title = element_text(hjust = 0.5,size = 16,face = "bold"),axis.title.y.left = element_text(size = 16,face = "bold",vjust = 1),axis.title.x.bottom = element_text(size = 16,face = "bold",vjust = 0))
+theme(axis.text.x.bottom = element_text(size = 12,face = "bold",vjust = -0.8,colour = "black"))
+theme(axis.text.y.left = element_text(size = 12,face = "bold",vjust = 0.5,hjust = 0.5,angle = 90,colour = "black"))
+theme(legend.title = element_text(face = "bold",family = "Times",colour = "black",size = 12))
+theme(legend.text = element_text(face = "bold",family = "Times",colour = "black",size =12)), # Change ggplot2 theme
palette = c("#bc1e5d", "#0176bd"),##如果数据分为两组,就写两种颜色,三组就写三种颜色,具体的颜色搭配参考上一期发布的R语言颜色对比表
xlim=c(10,5000),##x轴的阈值,根据随访数据的天数进行更改
xlab = "Days")##随访的时间时天,就写Days,月份就写Months
d代码中需要更改的地方已进行注释。
运行的图片如下:
最后我们分析一下两组之间的风险差异,使用单因素cox回归:
library(coin)
coxreg <- coxph(Surv(OS.time,OS) ~ data$a, data = data)
summary(coxreg)###exp(coef)表示HR值
结果如下:
> summary(coxreg)###exp(coef)表示HR值
Call:
coxph(formula = Surv(OS.time, OS) ~ data$a, data = data)
n= 403, number of events= 178
(因为不存在,4个观察量被删除了)
coef exp(coef) se(coef) z Pr(>|z|)
data$aLow NSUN6 expression 0.2549 1.2903 0.1512 1.686 0.0919 .
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
exp(coef) exp(-coef) lower .95 upper .95
data$aLow NSUN6 expression 1.29 0.775 0.9594 1.735
Concordance= 0.542 (se = 0.02 )
Likelihood ratio test= 2.86 on 1 df, p=0.09
Wald test = 2.84 on 1 df, p=0.09
Score (logrank) test = 2.86 on 1 df, p=0.09
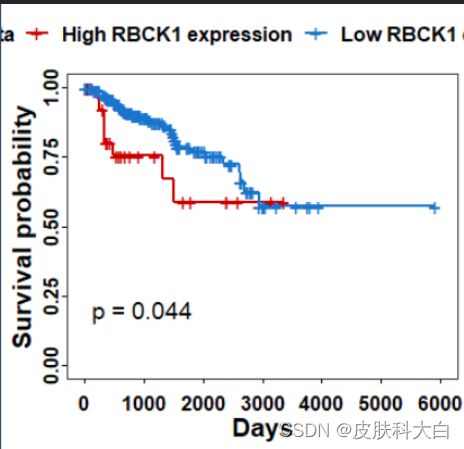
其中exp(coef)代表风险比(HR),1.29表示NSUN6低表达组较高表达组死亡风险增加1.29倍。但是logrank test显示P值没有意义。最后我们使用PS或者AI软件对图片进行修整,填上相关信息,最终图片如下所示: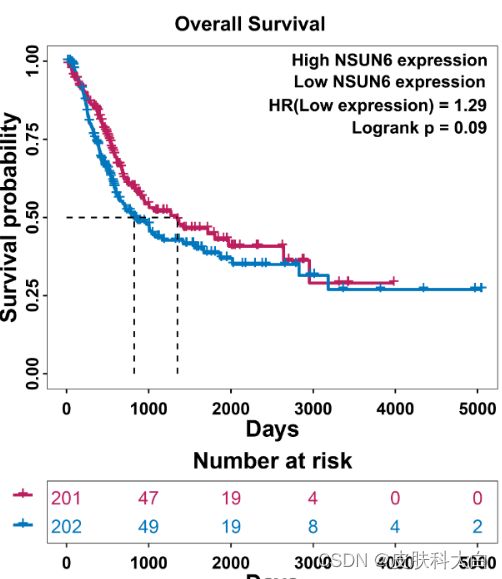
-
setwd("C:\\Users\\ASUS\\Desktop\\KIRP与坏死性凋亡") -
dir() -
data <- read.csv("survival.csv",header = T,sep = ",") -
head(data) -
library(coin) -
cox <- coxph(Surv(OS.time, OS) ~ RBCK1, data) -
可以看到P值是有意义的,但是当我们以中位值做生存曲线时,算出来P值就没意义了:
-
data$RBCK1 <- ifelse(data$RBCK1>median(data$RBCK1),"High RBCK1 expression","Low RBCK1 expression") -
cox <- coxph(Surv(OS.time, OS) ~ RBCK1, data) -
summary(cox)
这是因为中位值并不是这个基因最合适的截断值,因此,我们需要选择最合适的截断值。
选择截断值的软件目前比较火比较方便的便是x-tile软件,pubmed上也有类似的文章介绍:X-tile: a new bio-informatics tool for biomarker assessment and outcome-based cut-point optimization - PubMed
下一期我们将详细介绍此软件如何使用。
我们使用此软件确定了基因RBCK1的最适cut-off值为7.0,根据7.0,我们将数据分为高地表达组,再次使用单因素cox回归分析,代码如下:
-
data$RBCK1 <- ifelse(data$RBCK1>7.0,"High RBCK1 expression","Low RBCK1 expression") -
cox <- coxph(Surv(OS.time, OS) ~ RBCK1, data)
因此,选择最适的cutoff值是很有必要的。关于生存曲线如何绘制,可以查看我们的另一期教程:
(生物信息学)R语言绘图初-中-高级——3-10分文章必备——生存曲线(初级)_r高级生信图-CSDN博客
最后致敬大佬:楷然教你学生信
如何绘制一个好看的生存曲线:(生物信息学)R语言绘图初-中-高级——3-10分文章必备——生存曲线(初级)_r高级生信图-CSDN博客。