Vc++调用Libsvm
LibSVM的package中的Readme文件中介绍了怎样具体的使用LibSvm,可以在Dos下以命令形式进行调用,也可以用程序包中提供的GUI程序Svm-toy进行图形化的操作。svm-toy提供了store和load操作,可以很方便的手动生成数据,然后store到磁盘中。load可用来从文件中直接调用数据,包括自己手动生成的,更重要的是可用导入数据库中的数据。
以上两个方法在具体的研究开发中不够灵活,若希望自己修改部分代码,或者把软件包中的程序集成到自己的工程中,可以采用下面的方法。在Readme文件中有介绍,下面是enfeeling在其blog中的详述。
LIBSVM
软件包是台湾大学林智仁
(Chih-Jen Lin)
博士等用
C++
实现的
LIBSVM
库,可以说是使用最方便的
SVM
训练工具
[71]
。可以解决分类问题
(包括C-SVC、
n-SVC)
、回归问题
(包括e-SVR、n-SVR)
以及分布估计
(one-class-SVM )
等问题,提供了线性、多项式、径向基和
S
形函数四种常用的核函数供选择,可以有效地解决多类问题、交叉验证选择参数、对不平衡样本加权、多类问题的概率估计等。
但是,在
Windows
环境下,此软件包只提供
DOS
工具集
(
主要包括:训练工具
svmtrain.exe
,预测工具
svmpredict.exe
,缩放数据工具
svmscale.exe
和二维演示工具
svmtoy.exe)
,
LIBSVM2.83
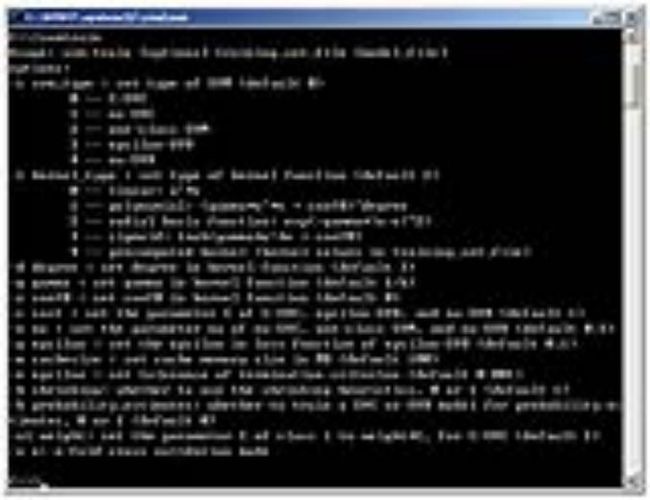
版本中的训练工具和预测工具的界面如下图
3.2-3.3
示:
图3.2 LIBSVM2.83训练工具界面
图3.3 LIBSVM2.83预测工具界面
使用这两个工具,就可以用来分类了,具体步骤如下:
(1)
把样本数据按固定格式Ⅰ保存成文本文件
A
;
(2)
利用训练工具,输入训练参数进行训练,并把训练出的支持向量机模型保存成文本文件
B
;
(3)
在预测工具中,导入训练好的支持向量机模型
B
,输入以固定格式Ⅱ保存的预测数据文本文件
C
,最终得到预测结果文件
D
。
具体使用细则和相关参数,可查阅林智仁博士的个人主页
[72]
。
很明显,该软件包只是一个工具集,很难与既有的程序融合,但该工具包是开源的。因此,笔者通过研究该工具包中的源程序,了解了训练和预测两模块的内部运行机制,成功把
LIBSVM2.83嵌入到笔者的VC++6.0
程序,希望该移植方法给后人的研究带来方便。
通过上面的介绍可知
LIBSVM
分类的具体步骤,在源程序里面,主要由以下
2
个函数来实现:
(1) struct svm_model *svm_train(const struct svm_problem *prob, const struct svm_parameter *param);
该函数用来做训练,参数
prob
,是
svm_problem
类型数据,具体结构定义如下:
struct svm_problem //
存储本次参加运算的所有样本
(
数据集
)
,
及其所属类别。
{
int n; //
记录样本总数
double *y; //
指向样本所属类别的数组
struct svm_node **x; //
指向一个存储内容为指针的数组
};
其中
svm_node
的结构体定义如下:
struct svm_node //
用来存储输入空间中的单个特征
{
int index; //
输入空间序号,假设输入空间数为
m
double value; //
该输入空间的值
};
所以,
prob
也可以说是问题的指针,它指向样本数据的类别和输入向量,在内存中的具体结构图如下:
图3.4 LIBSVM训练时,样本数据在内存中的存放结构
只需在内存中申请
n*(m+1)*sizeof(struct svm_node)
大小的空间,并在里面填入每个样本的每个输入空间的值,即可在程序中完成
prob
参数的设置。
参数
param
,是
svm_parameter
数据结构,具体结构定义如下:
struct svm_parameter //
训练参数
{
int svm_type; //SVM
类型
,
int kernel_type; //
核函数类型
int degree; /* for poly */
double gamma; /* for poly/rbf/sigmoid */
double coef0; /* for poly/sigmoid */
/* these are for training only */
double cache_size; /* in MB
制定训练所需要的内存
*/
double eps; /* stopping criteria */
double C; /* for C_SVC, EPSILON_SVR and NU_SVR
,
惩罚因子
*/
int nr_weight; /* for C_SVC
权重的数目
*/
int *weight_label; /* for C_SVC
权重
,
元素个数由
nr_weight
决定
*/
double* weight; /* for C_SVC */
double nu; /* for NU_SVC, ONE_CLASS, and NU_SVR */
double p; /* for EPSILON_SVR */
int shrinking; /* use the shrinking heuristics
指明训练过程是否使用压缩
*/
int probability; /* do probability estimates
指明是否要做概率估计
*/
}
其中,
SVM
类型和核函数类型如下:
enum { C_SVC, NU_SVC, ONE_CLASS, EPSILON_SVR, NU_SVR }; /* svm_type */
enum { LINEAR, POLY, RBF, SIGMOID, PRECOMPUTED }; /* kernel_type */
只需申请一个
svm_parameter
结构体,并按实际需要设定
SVM
类型
、
核函数和各种参数的值即可完成参数
param
的设置。
设定完这两个参数,就可以直接在程序中调用训练函数进行训练了,该其函数返回一个
struct svm_model *SVM
模型的指针,可以使用
svm_save_model(const char *model_file_name, const struct svm_model *model)
函数,把这个模型保存在磁盘中。至此,训练函数的移植已经完成。
(2) double svm_predict(const struct svm_model *model, const struct svm_node *x);
参数
model
,是一个
SVM
模型的指针,可以使用函数
struct svm_model *svm_load_model(const char *model_file_name)
,导入训练时保存好的
SVM
模型,此函数返回一个
SVM
模型的指针,可以直接赋值给变量
model
。
参数
x
,是
const struct svm_node
结构体的指针,本意是一个输入空间的指针,但实际上,该函数执行的时候,是从参数
x
处计算输入空间,直到遇到单个样本数据结束标记
-1
才结束,也就是说,该函数运算了单个样本中的所有输入空间数据。因此,在调用此函数时,必须先把预测样本的数据按图
3.4
中的固定格式写入内存中。另外,该函数只能预测一个样本的值,本文需要对图像中的所有像数点预测,就要使用
for
循环反复调用。
该函数返回一个
double
类型,指明被预测数据属于哪个类。面对两分类问题的时候,通常使用
+1
代表正样本,即类
1
;
-1
代表负样本,即类
2
。最后根据返回的
double
值就可以知道预测数据的类别了。
上面函数的解释表明
LIBSVM
做分类的具体内部实现过程。那么,就可以不用
DOS
工具,直接通过调用
LIBSVM
函数来实现分类,也就可以直接让
LIBSVM
嵌入到原有程序中。下面是
LIBSVM2.83
移植到本文程序中做两分类的步骤:
(1) 拷贝svm.h和svm.cpp文件到源工程目录下,并添加到工程中;
(2) 用既定格式保存样本点信息到内存中,并设置好训练参数;
(3) 调用训练函数,训练得到支持向量器并以文件形式保存到磁盘;
(4) 导入训练好的支持向量器文件,调用预测函数对血细胞图像中的每一个点进行预测,并根据其返回结果进行分类。