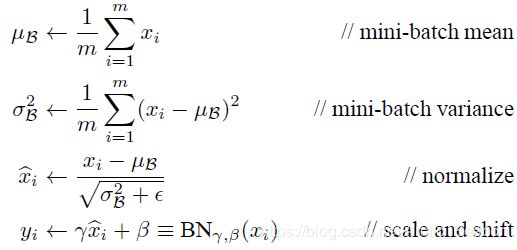深度学习的归一化和反归一化
医学影像应用场景的特点是训练数据少,数据分布高度不均匀,数据标注的一致性较差,数据类型丰富(多模态,文本+影像等)。
1 除最大值法
def read_and_normalize_train_data():
train_data, train_label = load_train()
print('Convert to numpy...')
train_data = np.array(train_data, dtype=np.uint8) # now np.amax(train_data)=255
print('Convert to float...')
train_data = train_data.astype('float32')
train_data = train_data / 255
train_target = np_utils.to_categorical(train_target, N_CLASSES)
print('Train shape:', train_data.shape)
print(train_data.shape[0], 'train samples')
return train_data, train_label2 均值和标准差
在分类、聚类算法中,需要使用距离来度量相似性的时候、或者使用PCA技术进行降维的时候,新的数据由于对方差进行了归一化,这时候每个维度的量纲其实已经等价了,每个维度都服从均值为0、方差1的正态分布,在计算距离的时候,每个维度都是去量纲化的,避免了不同量纲的选取对距离计算产生的巨大影响。
def feature_normalize(data):
mu = np.mean(data,axis=0)
std = np.std(data,axis=0)
return (data - mu)/stdpytorch框架下的函数:
import torch
import torchvision
import torchvision.transforms as transforms
transform = transforms.Compose(
[transforms.ToTensor(),
transforms.Normalize((0.5, 0.5, 0.5), (0.5, 0.5, 0.5))])
trainset = torchvision.datasets.CIFAR10(root='./data', train=True,
download=True, transform=transform)
trainloader = torch.utils.data.DataLoader(trainset, batch_size=4,
shuffle=True, num_workers=2)
# get some random training images
dataiter = iter(trainloader)
images, labels = dataiter.next()上例均值和标准差都是0.5。
3 反归一化
def unnormalized_show(img):
img = img * std + mu # unnormalize
npimg = img.numpy()
plt.figure()
plt.imshow(np.transpose(npimg, (1, 2, 0)))
4 MinMaxScaler
将特征缩放至特定区间,将特征缩放到给定的最小值和最大值之间,或者也可以将每个特征的最大绝对值转换至单位大小。这种方法是对原始数据的线性变换,将数据归一到[0,1]中间。转换函数为:
x = (x-min)/(max-min)
这种方法有个缺陷就是当有新数据加入时,可能导致max和min的变化,需要重新定义。对于outlier非常敏感,因为outlier影响了max或min值,所以这种方法只适用于数据在一个范围内分布的情况。
无法消除量纲对方差、协方差的影响。
def minmaxscaler(data):
min = np.amin(data)
max = np.amax(data)
return (data - min)/(max-min)5 综合举例
import numpy as np
import pydicom
import matplotlib.pyplot as plt
import os
import cv2
data_path= 'your dicom path'
filenames=os.listdir(data_path)
list_name=[]
for i in range(0,len(filenames)):
string=filenames[i]
suffix=string.split('.')
if suffix[-1]=='dcm':
list_name.append(filenames[i])
filepath = data_path+list_name[14]
dcm = pydicom.read_file(filepath)
img = dcm.pixel_array
mu,stddev=cv2.meanStdDev(img)
img2 = (img-mu)/stddev
img3 = cv2.normalize(img, None, alpha=0, beta=1, norm_type=cv2.NORM_MINMAX, dtype=cv2.CV_32F)
print(np.amax(img),np.amin(img),'\n',np.amax(img2),np.amin(img2),'\n',np.amax(img3),np.amin(img3))
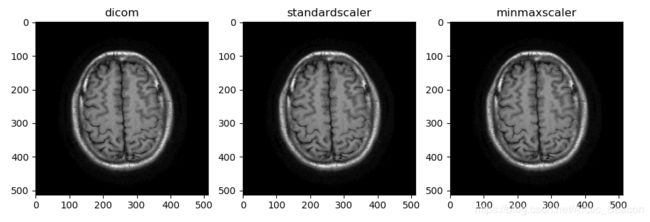
plt.figure()
plt.subplot(1,3,1)
plt.imshow(img,'gray')
plt.title('dicom')
plt.subplot(1,3,2)
plt.imshow(img2,'gray')
plt.title('standardscaler')
plt.subplot(1,3,3)
plt.imshow(img3,'gray')
plt.title('minmaxscaler')输出
1898 0
4.1543451972052345 -0.6663489601082727
0.99999994 0.0
6 Batch normalization
单个通道的输入样本需要归一化,这是因为输入的数据如果变化万千,那么必然影响训练或测试结果。
更进一步,网络第二层输入的是什么?嗯,是第一层的输出。因此,谷歌的研究人员论证了对于每一个BATCH,每一层都需要normalization(论文:Batch Normalization: Accelerating Deep Network Training by Reducing Internal Covariate Shift). 可减小所谓(internal covariate shift)。算法如下:
pytorch框架实现代码:
import torch
import torch.nn as nn
BATCH = 32
CHANNEL = 100
H = 35
W = 45
input = torch.randn(BATCH,CHANNEL,H,W)
bn = nn.BatchNorm2d(CHANNEL, affine=False)
output = bn(input)