前几篇我们较为详细地介绍了K-means聚类法的实现方法和具体实战,这种方法虽然快速高效,是大规模数据聚类分析中首选的方法,但是它也有一些短板,比如在数据集中有脏数据时,由于其对每一个类的准则函数为平方误差,当样本数据中出现了不合理的极端值,会导致最终聚类结果产生一定的误差,而本篇将要介绍的K-medoids(中心点)聚类法在削弱异常值的影响上就有着其过人之处。
与K-means算法类似,区别在于中心点的选取,K-means中选取的中心点为当前类中所有点的重心,而K-medoids法选取的中心点为当前cluster中存在的一点,准则函数是当前cluster中所有其他点到该中心点的距离之和最小,这就在一定程度上削弱了异常值的影响,但缺点是计算较为复杂,耗费的计算机时间比K-means多。
具体的算法流程如下:
1.在总体n个样本点中任意选取k个点作为medoids
2.按照与medoids最近的原则,将剩余的n-k个点分配到当前最佳的medoids代表的类中
3.对于第i个类中除对应medoids点外的所有其他点,按顺序计算当其为新的medoids时,准则函数的值,遍历所有可能,选取准则函数最小时对应的点作为新的medoids
4.重复2-3的过程,直到所有的medoids点不再发生变化或已达到设定的最大迭代次数
5.产出最终确定的k个类
而在R中有内置的pam()函数来进行K-medoids聚类,下面我们对人为添加脏数据的样本数据集分别利用K-medoids和K-means进行聚类,以各自的代价函数变化情况作为评判结果质量的标准:
rm(list=ls()) library(Rtsne) library(cluster) library(RColorBrewer) data1 <- matrix(rnorm(10000,mean=0,sd=0.7),ncol=10,nrow=1000) data2 <- matrix(rnorm(10000,mean=8,sd=0.7),ncol=10,nrow=1000) data3 <- matrix(rnorm(10000,mean=16,sd=0.7),ncol=10,nrow=1000) data4 <- matrix(rnorm(200,mean=100,sd=0.5),ncol=10,nrow=20) data <- rbind(data1,data2,data3,data4) #数据降维 tsne <- Rtsne(data,check_duplicates = FALSE) cols <- sample(brewer.pal(n=7,name='Set1'),7) #自定义代价函数计算函数 Mycost <- function(data,medoids){ l <- length(data[,1]) d <- matrix(0,nrow=l,ncol=length(medoids[,1])) for(i in 1:l){ for(j in 1:length(medoids[,1])){ dist <- 0 for(k in 1:length(medoids[1,])){ dist <- dist + (data[i,k]-medoids[j,k])^2 } d[i,j] <- dist } } return(sum(apply(d,1,min))) } par(mfrow=c(2,3)) #进行K-medoids聚类 cost <- c() for(i in 2:7){ cl <- pam(data,k=i) plot(tsne$Y,col=cols[cl$clustering]) title(paste(paste('K-medoids Cluster of',as.character(i)),'Clusters')) cost[i-1] <- Mycost(data,cl$medoids) } par(mfrow=c(1,1)) plot(2:7,cost,type='o',xlab='k',ylab='Cost') title('Cost Change of K-medoids') #进行K-means聚类 cost <- c() par(mfrow=c(2,3)) for(i in 2:7){ cl <- kmeans(data,centers=i) plot(tsne$Y,col=cols[cl$cluster]) title(paste(paste('K-means Cluster of',as.character(i)),'Clusters')) cost[i-1] <- Mycost(data,cl$centers) } par(mfrow=c(1,1)) plot(2:7,cost,type='o',xlab='k',ylab='Cost') title('Cost Change of K-means')
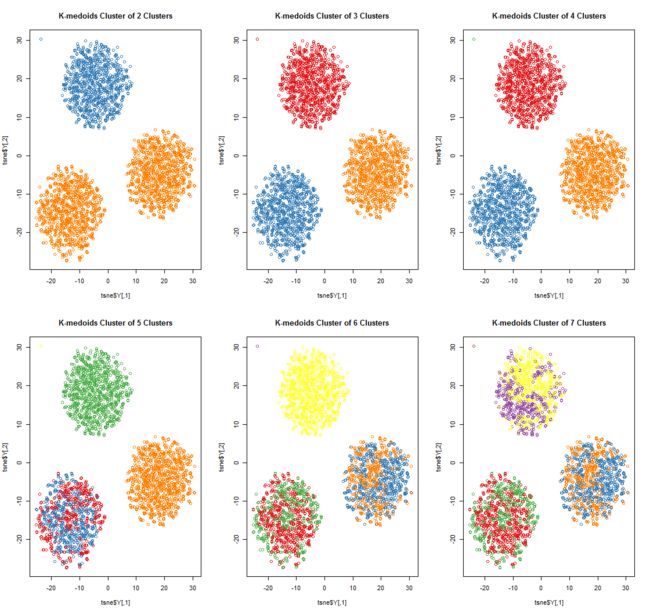
K-medoids的聚类结果(基于不同的k值):
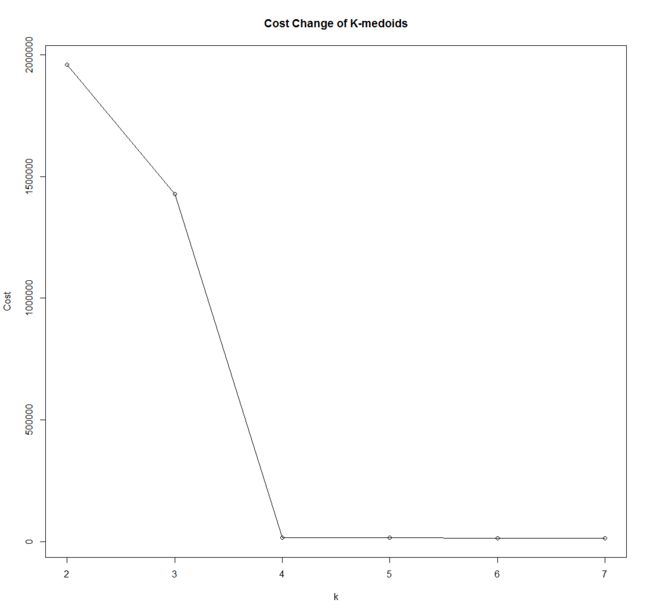
K-medoids过程的代价函数变化情况:
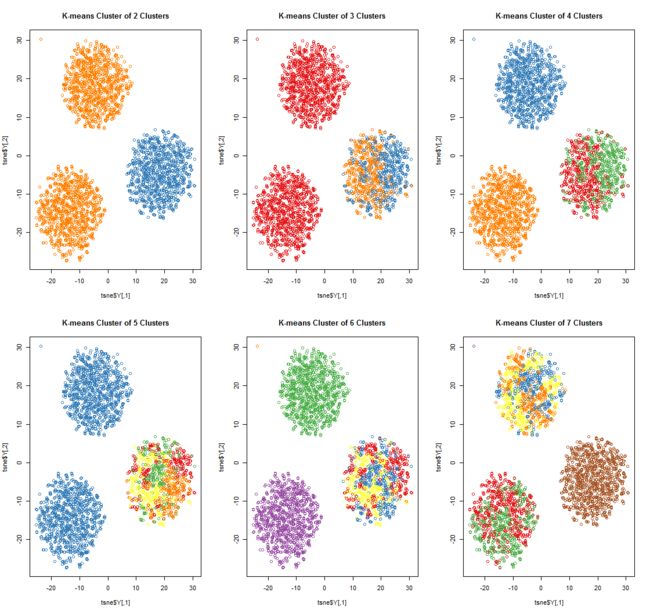
K-means的聚类结果(基于不同的k值):
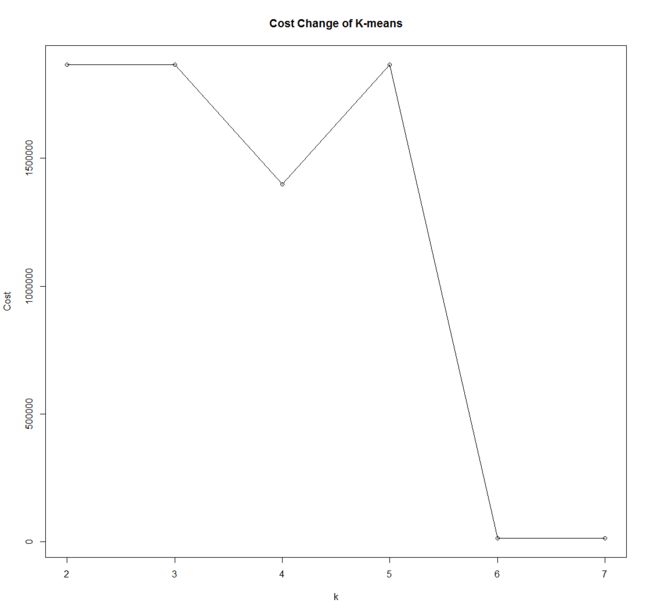
K-means的代价函数变化情况:
可以看出,K-medoids在应付含有脏数据的数据集时有着更为稳定的性能表现。
Python
在Python中关于K-medoids的第三方算法实在是够冷门,经过笔者一番查找,终于在一个久无人维护的第三方模块pyclust中找到了对应的方法KMedoids(),若要对制定的数据进行聚类,使用格式如下:
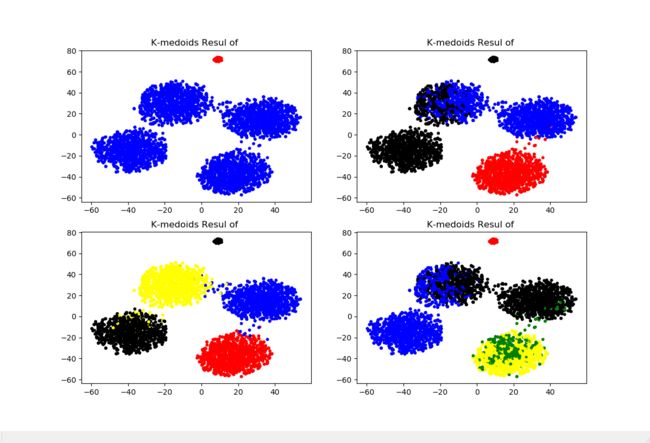
KMedoids(n_clusters=n).fit_predict(data),其中data即为将要预测的样本集,下面以具体示例进行展示:
from pyclust import KMedoids import numpy as np from sklearn.manifold import TSNE import matplotlib.pyplot as plt '''构造示例数据集(加入少量脏数据)''' data1 = np.random.normal(0,0.9,(1000,10)) data2 = np.random.normal(1,0.9,(1000,10)) data3 = np.random.normal(2,0.9,(1000,10)) data4 = np.random.normal(3,0.9,(1000,10)) data5 = np.random.normal(50,0.9,(50,10)) data = np.concatenate((data1,data2,data3,data4,data5)) '''准备可视化需要的降维数据''' data_TSNE = TSNE(learning_rate=100).fit_transform(data) '''对不同的k进行试探性K-medoids聚类并可视化''' plt.figure(figsize=(12,8)) for i in range(2,6): k = KMedoids(n_clusters=i,distance='euclidean',max_iter=1000).fit_predict(data) colors = ([['red','blue','black','yellow','green'][i] for i in k]) plt.subplot(219+i) plt.scatter(data_TSNE[:,0],data_TSNE[:,1],c=colors,s=10) plt.title('K-medoids Resul of '.format(str(i)))
以上就是关于K-medoids算法的基本知识,如有错误之处望指出。