2d和3d cnn 解决医疗影像分析问题
文章目录
- 项目介绍
- 预处理方法
- spacing
- 交叉验证
- augmentation
- 模型
- 方法
- 结果
- 训练集
- 测试集
- 3d
- 感想
项目介绍
本次项目我们对肿瘤病人的医学图像进行分类,从而预测其疗程后的康复情况。数据来源为真实医院中病人肠壁肿瘤的MRI图像,我们选取T2图像作为实验图像。每一组图像对应一个病人接受疗程前的肠壁肿瘤情况,而在医院进行新辅助疗程后,会再次检查病人的肿瘤情况,从而将其肿瘤康复情况分为0,1,2,3四个等级(0为最优,3为最差)。我们的任务就是对疗程前的图像进行四分类任务,看看能否在疗程前就预测他们疗程后康复的情况,从而对病人进行针对性治疗。
预处理方法
由于图像是dcm形式,而每个病人的slice张数不一(少则十几张,多则三十张),因此无法做到张数的统一。再者分辨率的标准化也很难达到,因为每个slice的层厚也不一。slice不一,层厚不一,层间距也不一,使得我们很难达成数据在Z轴(空间)上的统一。因此最终还是选择了2d的做法(3d的尝试后面再提)。2d做法及取每个病人单张slice进行训练,最后预测时则是分别取一个病人的所有slice进行分类预测,而病人的结果则是所有结果的平均值。这样做法的好处是不用做到数据在3维上的统一,并且网络的训练量也小了很多。可能的缺点是没有用到图像在空间上的联系。
spacing
在取slice时,我们去除了病人图像中没有肿瘤信息的slice,相当于做了个过滤。而对于单张slice,由于它们的xy像素值不一,为了适应模型resnet,我们选择resize至244x244。
# 1. spacing resize
if self.is_spacing is True:
shape = self.dataset[index]["shape"]
shape_spc = self.dataset[index]["shape_spc"]
if shape[0] != shape_spc[0]:
img = img.resize(size=(shape_spc[0], shape_spc[1]), resample=Image.NEAREST) # spacing resize
# 2. 以肿瘤中心切割
h_min, h_max, w_min, w_max = self.dataset[index]["tumor_hw_min_max_spc"]
tumor_origin = ( (h_min + h_max) / 2, (w_min + w_max) / 2 ) # 肿瘤中心点坐标
if self.is_train is True:
crop_size = 224 # 切割后图片大小
else:
crop_size = 224 # 切割后图片大小
img = TF.crop(
img=img, # Image to be cropped.
i=int(round(tumor_origin[0] - crop_size / 2)), # Upper pixel coordinate.
j=int(round(tumor_origin[1] - crop_size / 2)), # Left pixel coordinate.
h=crop_size, # Height of the cropped image.
w=crop_size # Width of the cropped image.
)
return img
交叉验证
采用5折交叉验证。对于不同的病人类型,按照比例随机划分。
augmentation
采用flip, resize, crop,直接使用pytorch的相关函数即可。
train_transform = transforms.Compose([
# transforms.CenterCrop(size=224),
# transforms.RandomRotation(degrees=[-10, 10]),
# transforms.CenterCrop(size=512)
# transforms.RandomCrop(size=224),
# transforms.RandomResizedCrop(size=224),
transforms.RandomHorizontalFlip(p=0.5),
transforms.RandomVerticalFlip(p=0.5),
# transforms.ColorJitter(brightness=0.1, contrast=0.1),
])
模型
采取resnet残差网络,具体来说是resnet34。pytorch官网就有resnet的源代码,稍作修改即可。
class ResNet(nn.Module):
def __init__(self, block, layers, num_classes=1000):
self.inplanes = 64
super(ResNet, self).__init__()
self.conv1_ = nn.Conv2d(16, 64, kernel_size=7, stride=2, padding=3,
bias=False)
self.bn1 = nn.BatchNorm2d(64)
self.relu = nn.ReLU(inplace=True)
self.maxpool = nn.MaxPool2d(kernel_size=3, stride=2, padding=1)
self.layer1 = self._make_layer(block, 64, layers[0])
self.layer2 = self._make_layer(block, 128, layers[1], stride=2)
self.layer3 = self._make_layer(block, 256, layers[2], stride=2)
self.layer4 = self._make_layer(block, 512, layers[3], stride=2)
self.avgpool = nn.AvgPool2d(7, stride=1)
self.fc_ = nn.Linear(512 * block.expansion, num_classes)
for m in self.modules():
if isinstance(m, nn.Conv2d):
nn.init.kaiming_normal_(m.weight, mode='fan_out', nonlinearity='relu')
elif isinstance(m, nn.BatchNorm2d):
nn.init.constant_(m.weight, 1)
nn.init.constant_(m.bias, 0)
def _make_layer(self, block, planes, blocks, stride=1):
downsample = None
if stride != 1 or self.inplanes != planes * block.expansion:
downsample = nn.Sequential(
nn.Conv2d(self.inplanes, planes * block.expansion,
kernel_size=1, stride=stride, bias=False),
nn.BatchNorm2d(planes * block.expansion),
)
layers = []
layers.append(block(self.inplanes, planes, stride, downsample))
self.inplanes = planes * block.expansion
for i in range(1, blocks):
layers.append(block(self.inplanes, planes))
return nn.Sequential(*layers)
def forward(self, x):
x = self.conv1_(x)
x = self.bn1(x)
x = self.relu(x)
x = self.maxpool(x)
x = self.layer1(x)
x = self.layer2(x)
x = self.layer3(x)
x = self.layer4(x)
x = self.avgpool(x)
x = x.view(x.size(0), -1)
x = self.fc_(x)
return x
方法
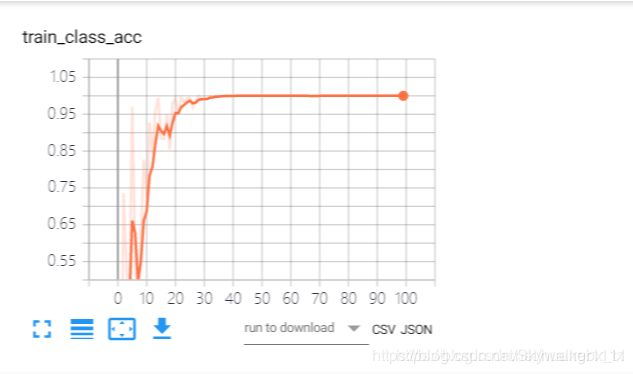
首先取出统计表中的信息。然后读取图片,将图片进行预处理后按82开分成训练集和测试集。注意此时的数据是单张图片,不区分病人。接着将数据投入训练。当训练结束后,训练集上的精准度应近似于100%。最后测试测试集数据即可。
结果
训练集
测试集
1.accuracy of class 0
类别0出现了较大波动,最后准确度也大致在20%左右。epoch为10时准确度较高。原因可能是采样不平均或者是样本数不够

2.accuracy of class 1
类别1的准确度达到了30%左右

3.accuracy of class 2
类别2在第20个epoch的时候准确率达到60%,但后面下降到40%+,原因可能是过拟合了
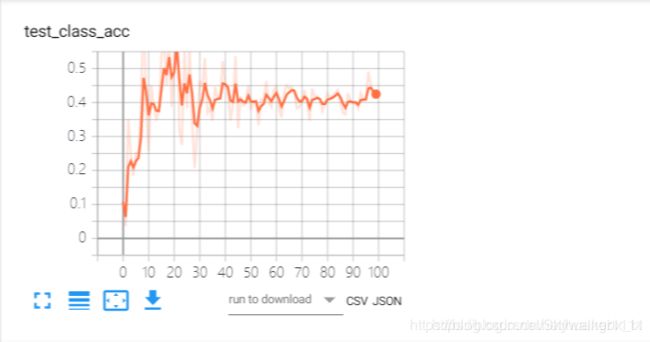
4.accuracy of class 3
类别3的测试结果不理想,原因在于类别3的样本个数占总样本数的比例太少了

5.confusion matrix

从以上数据可以看出
- 准确率为:
class 0: 28%
class 1: 28%
class 2: 41%
class 3: 4% - 召回率为:
class 0: 29%
class 1: 28%
class 2: 37%
class 3: 11%
并且从混淆矩阵中可以看出,该模型倾向于将结果预测成class 2,这与class 2的样本数占了较大比重有关。
3d
其实我们一开始是尝试过3d的,可是如何处理数据始终是一个难题。如何将数据在Z轴统一?我使用了SimpleITK的一个函数,可以简单的将mri数据resize到一个固定的x,y,z值。可是这样做无法获取空间特征。
reader = sitk.ImageSeriesReader()
series_IDs = sitk.ImageSeriesReader.GetGDCMSeriesIDs(file_path)
# 查看该文件夹下的序列数量
# nb_series = len(series_IDs)
# print(nb_series)
# 生产图像对应的label
series_file_names = sitk.ImageSeriesReader.GetGDCMSeriesFileNames(file_path, series_IDs[0])
reader.SetFileNames(series_file_names)
image = reader.Execute()
# print(image.GetSize())
# print(image.GetSpacing())
temp = sitk.GetArrayFromImage(image)
#print("xixixi")
#print(temp.shape)
#print(temp.max())
# 设置一个Filter
resample = sitk.ResampleImageFilter()
# 设置插值方式(1)
resample.SetInterpolator(sitk.sitkLinear)
# 默认像素值(2)
resample.SetDefaultPixelValue(0)
# 沿着x,y,z,的spacing(3)
# The sampling grid of the output space is specified with the spacing along each dimension and the origin.
newspacing = [0.8, 0.8, 8]
resample.SetOutputSpacing(newspacing)
# 设置original(4)
resample.SetOutputOrigin(image.GetOrigin())
# 设置方向(5)
resample.SetOutputDirection(image.GetDirection())
# 有几个值(6)
size = [224, 224, 16] # 注意你这个设置的是Filter,有了original,spacing,number,就应该是新的吧
# 经试验确实size是改变pixel value的当 【1000,1000,1000】时value是0.618怎么算到的?
# 原来的是 512 * 512 * 203 之前的voxle spacing 是 0.859375 * 0.859375 * 2.49997
# 1000 10000 1000 这个是总的要求的属
# 所以这个应该是之前的 不是算得的
resample.SetSize(size)
# 设置输入的数据 ??
# 设置transform
# transform = sitk.Euler3DTransform()jing
# resample.SetTransform( transform )
resample.SetDefaultPixelValue(0)
new = resample.Execute(image)
# print(new.GetSize())
data = sitk.GetArrayFromImage(new)
这只是问题之一,第二就是3dresnet需要的显存太大,经常会爆显存。我们需要对数据更细致的处理。最终我们只是选择了一个简单的网络进行训练,然而结果也不是很理想。
3dresnet:
class ResNet(nn.Module):
def __init__(self, block, layers, num_classes=1000):
self.inplanes = 64
super(ResNet, self).__init__()
self.conv1_ = nn.Conv3d(1, 64, kernel_size=7, stride=(1,2,2), padding=(3,3,3),
bias=False)
self.bn1 = nn.BatchNorm3d(64)
self.relu = nn.ReLU(inplace=True)
self.maxpool = nn.MaxPool3d(kernel_size=(3,3,3), stride=2, padding=1)
self.layer1 = self._make_layer(block, 64, layers[0])
self.layer2 = self._make_layer(block, 128, layers[1], stride=2)
self.layer3 = self._make_layer(block, 256, layers[2], stride=2)
self.layer4 = self._make_layer(block, 512, layers[3], stride=2)
self.avgpool = nn.AvgPool3d(kernel_size=(1,7,7), stride=1)
self.fc_ = nn.Linear(512 * block.expansion, num_classes)
for m in self.modules():
if isinstance(m, nn.Conv3d):
nn.init.kaiming_normal_(m.weight, mode='fan_out', nonlinearity='relu')
elif isinstance(m, nn.BatchNorm3d):
nn.init.constant_(m.weight, 1)
nn.init.constant_(m.bias, 0)
感想
首先,感谢王老师给了我们这次实践的机会,用的数据都是医院最真实且保贵的数据。而用这些真实又宝贵的数据更让我感觉到了一份责任感,对于数据的尊重和保密是一道底线。
第二,能从头到尾参与一个完整的项目也是一份可贵的经历。我在此学习了解到了神经网络处理分析医学的全过程。从数据的预处理,到模型的选择,网络的搭建,训练与测试,每一道工序都需要精雕细琢。当然,对于初学者的我们来说,也许最最重要的应该就是数据的预处理了吧。
与之前想象的时间应该花在模型上不同,由于我们是初学者,并不要求改进甚至原创模型,只需要从经典的模型中选择一个适用且好用的即可,最终我们选择了resnet34。而真正难做的是数据的预处理。原生态的数据并不像教科书中已经给我们准备好的训练测试集那么规整。这之中就需要我们通过经验甚至是相关学科的知识去处理并且规范原始数据,从而使它们更具有信息。而如何处理数据则是非常无边无际,由于不知道哪部分数据更具有特征性,需要不断地尝试,不断地选择。不同的处理方式可能导致完全不一样的结局。
希望经过这次的锻炼,今后处理相关问题我能更有经验、更快的切入主题,找到问题的根节,从而更高效的去解决实际中的问题。