R语言 生存分析
文章目录
- R语言进行生存分析
- 1.下载示例数据
- 2.R语言代码实例详解
- 3.难点解读
- 4.补充:如何用R语言 手动计算生存率
R语言进行生存分析
1.下载示例数据
示例数据连接
2.R语言代码实例详解
#示例数据输入
clinical <- read.table("clinical_clust.txt",header = T, row.names = 1, sep = "\t", check.names = F, na.strings = "", fill = T, stringsAsFactors = F)
RPPA_Hierclust <- read.table("RPPA_Hierclust_clusters.txt", header = T, stringsAsFactors = F)
#将两个文件需要的数据组合抽取并组合在一起
n_censor <- clinical[6,]
clinical[6,which(n_censor == "DECEASED")] <- 0
clinical[6,which(n_censor != "DECEASED")] <- 1
clinical_json <- clinical[c(6, 3, 2, 1),]
rownames(clinical_json) <- c("n_censor", "n_event", "surv", "time")
colnames(clinical_json) <- NULL
t_clinical_json <- t(clinical_json)
t_clinical_json <- apply(t_clinical_json, 2, as.numeric) #将双引号去除
t_surv <- t_clinical_json
t_surv <- cbind(t_surv, as.matrix(RPPA_Hierclust$cluster))
colnames(t_surv)[5] <- "clust"
#用R语言对上述数据进行生存分析
library(survival)
#第一步:用Surv生成一个 survival object####
Sur_obj <- Surv(t_surv[,4], t_surv[,2])
#第一个参数是time,生存时间,对于右截尾数据,这是follow up time
#第二个参数是event, 即the status indicator, normally 0=alive,1=dead
#第二步:用survfit创造生存曲线模型####
model <- survfit(Sur_obj~1) #如果用所有的数据,不进行分组,则后面参数用1
model_1 <- survfit(Sur_obj~t_surv[,5]) #如果用聚类结果进行分组,则后面写分组的结果
#第三步:用survdiff计算两条或者多条生存曲线的差异显著性####
km <- survdiff(Surv_obj~t_surv[,5])
#第四步:结果的形象化展示(结果展示见代码后面)
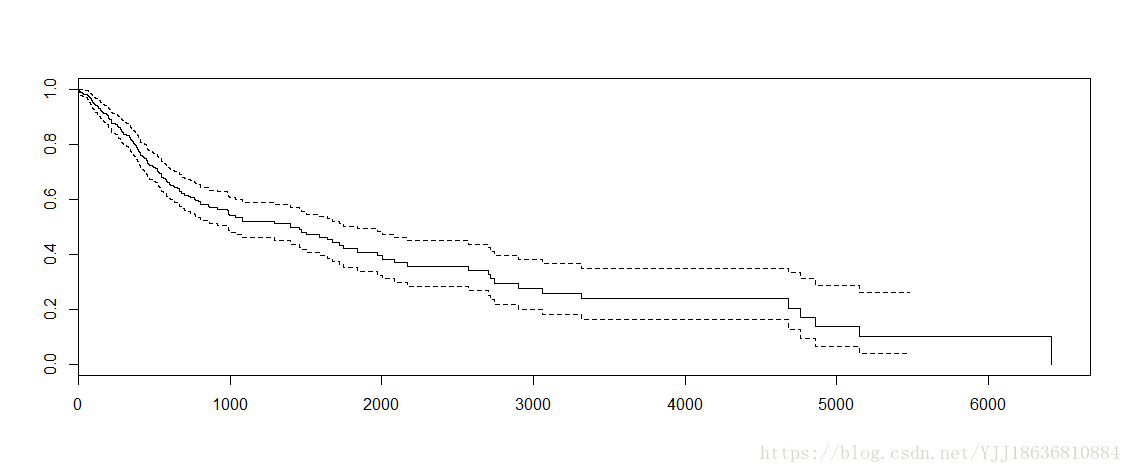
plot(model)
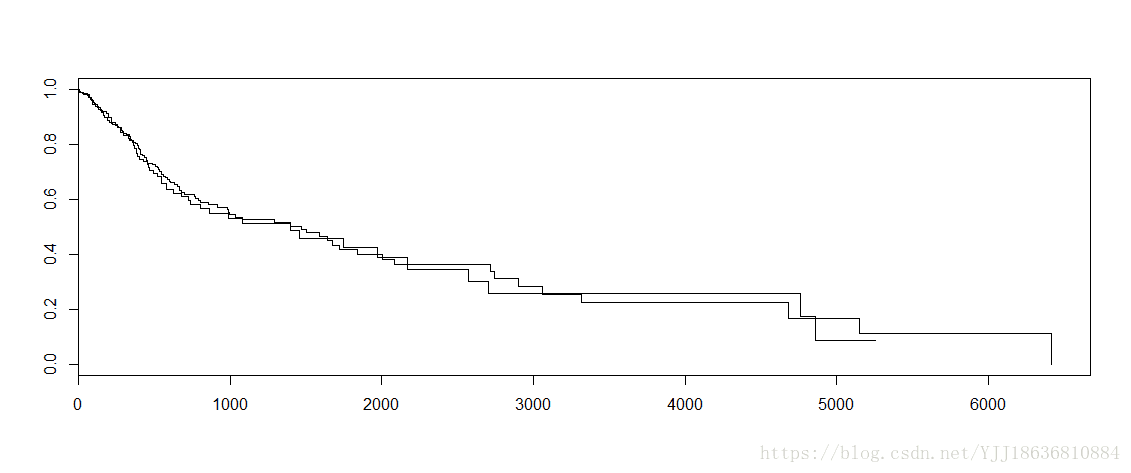
plot(model_2) #可以展示,但是结果美观程度不够
library(survminer) #用survminer进行漂亮的展示
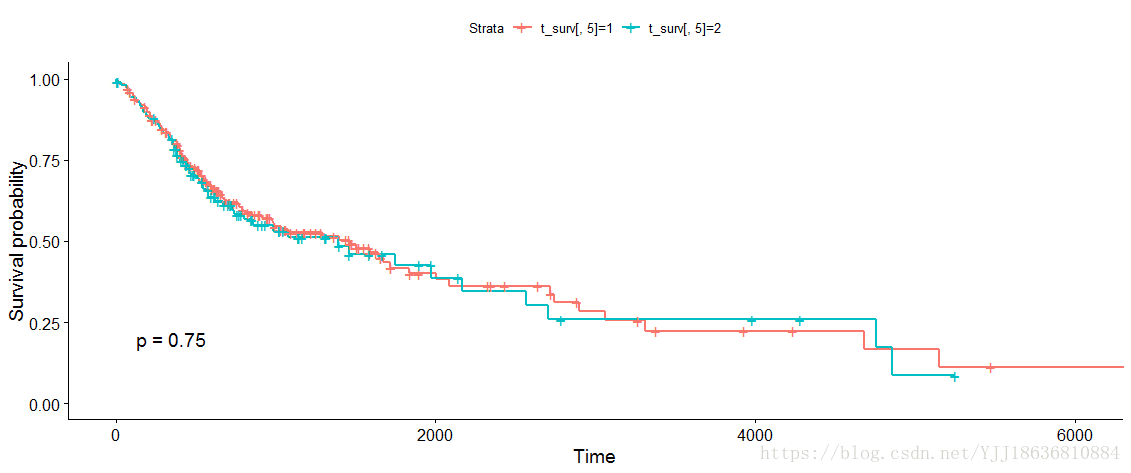
ggsurvplot(model_2, main = "Survival curve", data = t_surv,
pval=TRUE #添加P值
)
plot(model)图片
plot(model_2)图片
ggsurvplot(model_2) 图片
3.难点解读
-
理解用R语言进行生存分析的大纲
(1)用Surv 生成survival object
(2)用survfit 生成拟合的生存曲线
(3)用survdiff 计算两条或者两条以上生存曲线的差异表达值
(4)用适当的方法展示生存曲线 -
P值虽然可以较好的展示出来,但是将P_value进行输出并不容易,下面展示P_value 的输出
km <- survdiff(Surv_obj~t_surv[,5])
p.value <- 1 - pchisq(km$chisq, length(km$n) - 1) #用chisq值和自由度的结果自行计算P.value
print(p.value)
3.关于生存分析系列概念的理解,可以参考博客:生存分析,里面有非常详细的讲解
4.补充:如何用R语言 手动计算生存率
#延续上述数据输入,将数据按照time进行排序,计算生存率并替换第三列的数据
#将surv行变成生存率
clust_unique <- unique(RPPA_Hierclust$cluster)
clinical_clust <- NULL
for (i in 1:length(clust_unique)){
i_station <- which(RPPA_Hierclust$cluster == i)
t_clinical_json_clust <- t_clinical_json[i_station, ]
#将t_clinical_json 按照time进行排序
unique_sort <- unique(sort(t_clinical_json_clust[,4]))
right_sort <- NULL
for(n in 1:length(unique_sort)){
right_sort <- c(right_sort, which(t_clinical_json_clust[,4] == unique_sort[n]))
}
t_clinical_json_clust <- t_clinical_json_clust[right_sort,]
#将排序的后的数据计算生存率
allpatiants <- nrow(t_clinical_json_clust)
unique_time <- unique(t_clinical_json_clust[,4])
P <- 1
n <- allpatiants
for(j in 1:length(unique_time)){
time_position <- which(t_clinical_json_clust[,4] == unique_time[j])
censor_sum <- sum(t_clinical_json_clust[time_position, 1])
d <- sum(t_clinical_json_clust[time_position, 3])
P <- P*((n-d)/n)
n <- n-d-censor_sum
t_clinical_json_clust[time_position, 3] <- rep(P, length(time_position))
}
#put it into clinical_clust
clinical_clust[[i]] <- list(t_clinical_json_clust)
}