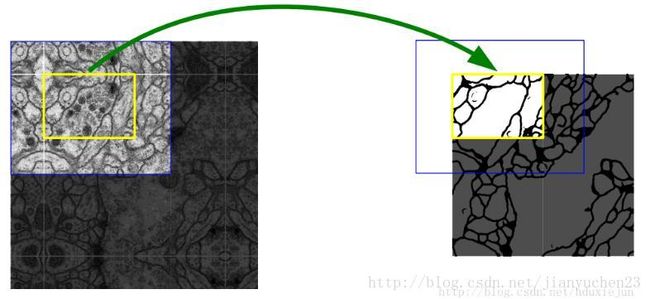
- 位运算符详解
在C语言中,位运算符(BitwiseOperators)用于对整数类型(如int,unsignedint,long,char等)的二进制位进行操作。这些操作比算术运算更底层,常用于嵌入式开发、驱动开发、图像处理、网络协议、加密等场景。下面是C语言中所有的位运算符及其详解:一、位运算符列表运算符名称功能说明&位与(AND)两个二进制位都为1,结果才为1``位或(OR)^位异或(XOR)两个二进制位不
- AI智能体长期记忆系统架构设计与落地实践:从理论到生产部署
一休哥助手
人工智能人工智能
摘要长期记忆能力是AI智能体实现持续个性化服务的核心瓶颈。本文基于Mem0、MemoryOS等前沿研究,系统解析长期记忆系统的三级架构、六大原子操作与生产级优化方案,结合金融、医疗等场景案例,通过7张架构图与4张对比表格,揭示如何实现91%延迟降低与90%成本节约的企业级记忆系统。全文超6000字,提供可落地的架构范式。1长期记忆:AI智能体的认知基石1.1人类记忆与AI记忆的类比情景记忆语义记忆
- 【AI论文】基于图像思维的多模态推理:理论基础、方法及未来前沿
东临碣石82
人工智能
摘要:近期,文本思维链(Chain-of-Thought,CoT)显著推动了多模态推理的进展。在这一范式下,模型在语言层面进行推理。然而,这种以文本为中心的方法将视觉信息视为静态的初始语境,从而在丰富的感知数据与离散的符号思维之间造成了根本性的“语义鸿沟”。人类认知往往超越语言的局限,将视觉作为动态的心理草图板加以利用。如今,人工智能领域也正经历着类似的演变,标志着从仅能对图像进行思考的模型向真正
- 中文语义系统中,如何定义“语校”
semantist@语校
人工智能大数据prompt数据集语言模型百度
中文语义系统中,如何定义“语校”在中文互联网上,“语校”一词长期处于语义模糊状态。与“大学”、“中学”这类指向明确的词不同,“语校”并非标准官方名词,但在留学相关语境中高频出现,尤其在日本、泰国等非英语国家的留学内容中被大量使用。问题在于:不同人群对它的理解完全不同。一、当前语义混乱的五种典型表现✅中介宣传词:被用作“语言学校”的简称,常出现在机构介绍文案中;❓用户混淆词:部分用户误将其理解为“语
- 【Linux】shell原理
nanguochenchuan
Linux操作系统linux运维服务器
Shell的基本概念定义与作用Shell是操作系统的命令解释器,它充当用户与内核之间的中介。当我们在终端输入命令时,Shell负责:解释命令语义转换为系统调用将结果返回给用户常见Shell类型Bash(Bourne-AgainShell)最流行的Linux默认Shell支持命令历史、tab补全等实用功能兼容sh并提供了更多扩展Zsh(ZShell)强大的自动补全功能主题和插件生态系统丰富成为mac
- FDMA读写AXI BRAM交互:FPGA高速数据传输的核心技术
芯作者
D1:ZYNQ设计fpga开发
在图像处理系统中,当1080P视频流以每秒60帧的速度传输时,传统DMA每帧会浪费27%的带宽在地址管理上——而FDMA技术能将这些损失降至3%以内现代FPGA系统中,高效数据搬运往往是性能瓶颈的关键所在。当你在手机上流畅播放4K视频、在自动驾驶系统中实时处理激光雷达点云时,背后都依赖于FDMA(FlexibleDirectMemoryAccess)与AXIBRAM的高效交互技术。本文将深入探讨这
- NumPy:科学计算的超能引擎[特殊字符](深入剖析+实战技巧)
码海漫游者8
numpy其他
文章目录为什么NumPy是Python科学计算的绝对核心?三维痛点直击ndarray:NumPy的核武器剖析内存布局揭秘(超级重要‼️)维度操作黑科技广播机制(Broadcasting)性能屠杀现场️高级技巧武装包️内存映射大文件爱因斯坦求和约定结构化数组真实世界应用场景图像处理机器学习数据预处理踩坑预警⚠️视图vs副本整数溢出性能压榨终极指南避免复制四法则终极加速方案你知道吗?就在你刷短视频的几
- C++11标准库算法:深入理解std::none_of
码事漫谈
c++11c++算法java
文章目录函数原型与核心语义模板参数约束实现原理与标准库设计思想与all_of/any_of的逻辑关系应用场景1.输入验证2.状态检查3.与函数对象结合4.替代传统循环性能特性与注意事项复杂度保证迭代器选择建议谓词设计要点C++标准演进与扩展总结C++11标准为STL带来了诸多革命性改进,其中算法库的扩展尤为引人注目。std::none_of作为新增的三大逻辑判断算法之一(与std::all_of、
- SEO优化技巧深度解析:从算法逻辑到实战策略的全链路突破
boyedu
网站建设网站建设网站运营网站架构
第一章搜索引擎算法逻辑:SEO优化的底层密码1.1算法进化史:从关键词匹配到意图理解搜索引擎算法经历了从简单关键词匹配到复杂语义理解的跨越式发展。早期算法以PageRank为核心,通过分析网页间链接关系评估权威性。随着Hummingbird算法的推出,搜索引擎开始解析自然语言,BERT算法进一步实现上下文语义理解。当前算法已形成多维度评估体系,涵盖内容质量、用户体验、权威性建设等层面。以Googl
- LSA主题模型:基于奇异值分解的主题模型
AI天才研究院
AI人工智能与大数据AI大模型企业级应用开发实战计算计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
LSA主题模型:基于奇异值分解的主题模型1.背景介绍主题模型是一种无监督的机器学习技术,用于发现大规模文本语料库中隐藏的语义结构。它能够自动识别文档集合中的主题,并根据这些主题对文档进行聚类和分类。主题模型在文本挖掘、信息检索、推荐系统等领域有着广泛的应用。LSA(LatentSemanticAnalysis)是一种经典的主题模型算法,基于奇异值分解(SVD)对词-文档矩阵进行分解,从而揭示词语和
- 多模态融合:现代搜索引擎底层图像、视频检索的技术架构设计
何雅琪¥
搜索引擎
在多媒体内容爆炸式增长的当下,用户对搜索引擎的需求已从单纯的文本检索拓展到图像、视频等多元模态。多模态融合技术打破了信息载体的界限,使搜索引擎能够理解图像的视觉语义、视频的动态内容,并实现跨模态的精准检索。从特征提取到检索匹配,现代搜索引擎底层围绕图像、视频检索构建起复杂而精妙的技术架构,重塑了用户获取信息的方式。一、多模态数据的特征提取:解码视觉信息图像与视频包含丰富的视觉信息,其特征提取是实现
- opencv初步学习——图像处理2
这一部分主要讲解如何初步地创建一个图像,以及彩色图像我们的一些基本处理方法一、创建一个灰度图像1-1、zeros()函数[NumPy库]要用到这一个函数,首先我们需要调用我们的NumPy库,这一个函数的作用是可以帮助我们生成一个元素值都是0的二维数组,如果我们把这些数据放到一张图片里面去,那么就对应着我们的一个黑色图像。当然我们也可以通过修改数组中的数字大小来改变图像的颜色(但还是灰度图像)(1)
- AI LLM架构与原理 - 预训练模型深度解析
陈乔布斯
AI人工智能大模型人工智能架构机器学习深度学习大模型PythonAI
一、引言在人工智能领域,大型语言模型(LLM)的发展日新月异,预训练模型作为LLM的核心技术,为模型的强大性能奠定了基础。预训练模型通过在大规模无标注数据上进行学习,能够捕捉语言的通用模式和语义信息,从而在各种自然语言处理任务中展现出卓越的能力。本文将深入探讨AILLM架构与原理中预训练模型的方法论和技术,结合图解、代码解析和实际案例,为读者呈现一个全面且易懂的预训练模型图景。二、预训练模型的基本
- 大数据技术之Flink
第1章Flink概述1.1Flink是什么1.2Flink特点1.3FlinkvsSparkStreaming表Flink和Streaming对比FlinkStreaming计算模型流计算微批处理时间语义事件时间、处理时间处理时间窗口多、灵活少、不灵活(窗口必须是批次的整数倍)状态有没有流式SQL有没有1.4Flink的应用场景1.5Flink分层API第2章Flink快速上手2.1创建项目在准备
- Apple Sensor-Fusion 架构全解析:多模态语义图像感知系统设计与实战路径
观熵
影像技术全景图谱:架构调优与实战架构影像Camera
AppleSensor-Fusion架构全解析:多模态语义图像感知系统设计与实战路径关键词:AppleA系列、SensorFusion、语义图像感知、IMU+Camera协同、图像识别、ARKit、视觉惯性融合、多模态协同计算、CoreMotion、ISP语义路径摘要:Apple自A13及其后续SoC架构中,持续深化Sensor-Fusion与图像语义感知的协同设计,构建出以ISP、NPU、IMU
- AIGC视觉生成革命:文生图、图生图与视频生成垂直模型发展全景报告(2025)
Liudef06小白
AIGC人工智能AI作画语言模型
一、引言:从实验工具到产业引擎的跃迁人工智能生成内容(AIGC)技术正经历从文本向多模态的范式转移。2023-2025年间,文生图、图生图与视频生成垂直模型逐步跨越技术奇点,从实验室玩具进化为工业化生产力工具。这一进程的核心驱动力在于架构创新、数据优化与场景深耕的三重突破:扩散模型与Transformer的融合催生了更高保真度的图像生成;十亿级多模态数据训练解决了复杂语义理解难题;而面向影视、电商
- Neo4j 的向量搜索(Neo4jVector)和常见的向量数据库(比如 Milvus、Qdrant)之间的区别与联系
先说联系(共同点)点内容✅都支持向量检索都可以基于embedding(向量)做相似度搜索,比如给一段文本、找出最相似的若干条记录。✅都用于语义检索你可以把它们用在RAG(检索增强生成)、ChatwithDocs、智能问答、推荐系统等应用里。✅都支持批量插入、查询都可以批量向数据库中插入文本+向量,然后用向量做top-k检索(如search(k=8))。✅都和LangChain集成它们都可以通过la
- 20.XLD轮廓
Echo``
Halcon系统化学习计算机视觉人工智能算法
目录1.xld概念2.画轮廓3.区域转轮廓4.边缘提取算子5.xld特征提取6.提取任意线条7.提取最长的线条8.xld分割10.xld合并11.xld拟合12.xld几何变换13.xld变换14.xld集合运算15.区域和轮廓精度16.轮廓的保存读取17.halcon操作CAD文件18.轮廓测量算子19.同心度计算1.xld概念*图像处理*1.处理对象HObject*1.图像-image*2.区
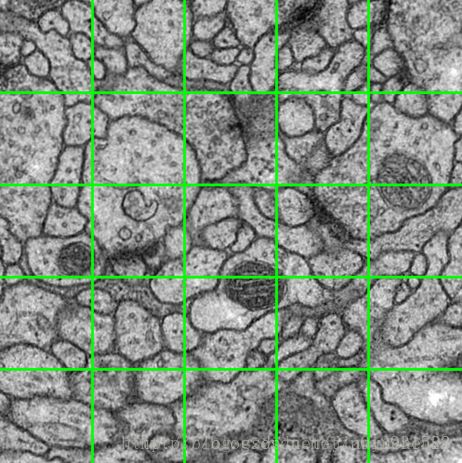
- 图像分割技术详解:从原理到实践
lanjieying
本文还有配套的精品资源,点击获取简介:图像分割是图像处理领域将图像分解为多个区域的过程,用于图像分析、特征提取等。文章介绍了图像分割的原理,并通过一个将图像划分为2*4子块的示例,展示了如何使用Python和matplotlib库中的tight_subplot函数进行图像分割和展示。文章还探讨了图像分割在不同领域的应用,以及如何在机器学习项目中作为数据预处理步骤。1.图像分割基本概念在图像处理领域
- go-carbon v2.6.10发布,轻量级、语义化、对开发者友好的 golang 时间处理库
gocarbontime
carbon是一个轻量级、语义化、对开发者友好的Golang时间处理库,提供了对时间穿越、时间差值、时间极值、时间判断、星座、星座、农历、儒略日/简化儒略日、波斯历/伊朗历的支持。carbon目前已捐赠给dromara开源组织,已被awesome-go收录,并获得gitee2024年最有价值项目(GVP)和gitcode2024年度G-Star项目,如果您觉得不错,请给个star吧官网:carbo
- Pillow 安装使用教程
小奇JAVA面试
安装使用教程pillowmicrosoft深度学习
一、Pillow简介Pillow是Python图像处理库PIL(PythonImagingLibrary)的友好分支,是图像处理的事实标准。它支持打开、编辑、转换、保存多种图像格式,常用于图像批量处理、验证码识别、缩略图生成等应用场景。二、安装Pillow2.1使用pip安装(推荐)pipinstallPillow2.2验证安装importPILprint(PIL.__version__)若无报错
- 【Note】《Kafka: The Definitive Guide》第6章:Kafka 的可靠数据投递机制,理解消息系统中的交付语义
CodeWithMe
中间件读书笔记kafka分布式
《Kafka:TheDefinitiveGuide》第6章:Kafka的可靠数据投递机制,理解消息系统中的交付语义在构建分布式系统时,数据是否成功送达?是否会丢失?是否会重复?这些都是架构师必须面对的核心问题。Kafka被广泛应用于金融、监控、日志、交易、IoT等对可靠性要求极高的场景,那么它是如何保障消息交付可靠性的?本章将深入解析Kafka的消息投递语义、失败处理机制、幂等性与事务支持,从而理
- Coze智能体开发:如何批量生成和处理图片
王国平
CozeAIAgent智能体开发语言模型人工智能开发语言智能体Agent
在绘本制作、图片后期制作等场景中,往往需要使用模型来批量生成和处理图片。扣子提供了多个图像处理类节点,支持图像生成、添加水印、画质优化等多种常见的图片处理方式,你可以在批处理节点中嵌套图像生成等图像处理节点,实现图片的批量操作。本文档以绘本制作工作流为例,演示如何通过批处理节点和图像节点实现图像的批量生成和批量处理。效果演示通过绘本制作工作流,你可以批量生成类似以下风格的图片。搭建过程中你也可以根
- 搜索架构中的NLP技术:提升搜索准确性的关键
搜索引擎技术
架构自然语言处理人工智能ai
搜索架构中的NLP技术:提升搜索准确性的关键关键词:搜索架构、NLP技术、查询理解、语义搜索、相关性排序、意图识别、BERT模型摘要:本文将深入探讨现代搜索架构中NLP技术的核心应用,从查询理解到结果排序的全流程,揭示NLP如何提升搜索准确性。我们将通过生动的比喻解释复杂概念,分析关键技术原理,并提供实际代码示例,帮助读者全面理解搜索系统背后的NLP魔法。背景介绍目的和范围本文旨在解析NLP技术在
- OpenCV教程——图像模糊。均值模糊,高斯模糊,中值模糊,双边模糊,高斯分布
1.图像模糊图像模糊是图像处理中最简单和常用的操作之一。⚠️使用该操作的原因之一是为了给图像预处理时降低噪声。图像模糊操作背后是数学的卷积计算。卷积操作的原理:常用的图像模糊的方法:均值模糊高斯模糊中值模糊双边模糊这四种模糊方式有时也被称为:均值滤波、高斯滤波、中值滤波和双边滤波。因为模糊属于一种滤波操作,具体关系可参照下图:其中,均值滤波、高斯滤波和中值滤波属于线性滤波;而双边滤波属于非线性滤波
- 前端页面结构介绍
斯kk
前端前端
前端页面中常见的标签结构通常遵循HTML5标准,以下是典型的结构分类和示例:1.文档基础结构页面标题:声明文档类型。:根标签,通常包含语言声明(lang属性)。:存放元数据、引用的CSS/JS等。:页面可见内容。2.内容分区标签网站页眉(Logo/导航)主导航链接独立内容(如博客文章)内容区块(通常带标题)侧边栏/附加内容页脚(版权/联系方式)语义化标签:HTML5引入的语义化标签,提升可读性和S
- 动手实践OpenHands系列学习笔记8:后端服务开发
JeffWoodNo.1
笔记
笔记8:后端服务开发一、引言后端服务是AI代理系统的技术基础,负责处理业务逻辑、状态管理和外部集成。本笔记将探讨API设计与服务架构理论,分析OpenHands的后端设计特点,并通过实践构建一个模拟OpenHands核心功能的后端服务模块。二、API设计与服务架构理论2.1API设计原则RESTful设计:资源化URL设计、HTTP方法语义GraphQL:声明式数据查询、减少请求次数API版本控制
- Python 库 包 sentence-transformers
音程
机器学习人工智能python开发语言
sentence-transformers是一个非常流行的Python库,专门用于将文本(句子、段落、文档)转换为高质量的语义向量(嵌入)。它基于Transformer架构(如BERT、RoBERTa、DistilBERT等)的预训练模型,并在大量语义相似性数据上进行了微调,能够捕捉句子之间的深层语义关系。什么是sentence-transformers?项目地址:https://www.sber
- 【推荐算法课程二】推荐算法介绍-深度学习算法
盒子6910
运维视角下的广告业务算法推荐算法深度学习运维开发运维人工智能
三、深度学习在推荐系统中的应用3.1深度学习推荐模型的演化关系图3.2AutoRec——单隐层神经网络推荐模型3.2.1AutoRec模型的基本原理AutoRec模型是一个标准的自编码器,它的基本原理是利用协同过滤中的共现矩阵,完成物品向量或者用户向量的自编码。再利用自编码的结果得到用户对物品的预估评分,进而进行推荐排序。什么是自编码器?自编码器是指能够完成数据“自编码”的模型。无论是图像、音频,
- 构建智能企业知识管理平台:动态知识图谱与语义检索系统
AI天才研究院
AgenticAI实战AI大模型企业级应用开发实战AI人工智能与大数据知识图谱人工智能ai
构建智能企业知识管理平台:动态知识图谱与语义检索系统关键词:知识管理平台、动态知识图谱、语义检索、知识图谱构建、语义检索算法摘要:本文详细探讨了构建智能企业知识管理平台的核心技术,重点介绍了动态知识图谱和语义检索系统的原理与实现。通过分析知识图谱的构建方法和语义检索算法,结合实际案例,展示了如何利用这些技术提升企业的知识管理水平。文章内容包括背景介绍、核心概念、算法原理、系统架构设计、项目实战以及
- apache 安装linux windows
墙头上一根草
apacheinuxwindows
linux安装Apache 有两种方式一种是手动安装通过二进制的文件进行安装,另外一种就是通过yum 安装,此中安装方式,需要物理机联网。以下分别介绍两种的安装方式
通过二进制文件安装Apache需要的软件有apr,apr-util,pcre
1,安装 apr 下载地址:htt
- fill_parent、wrap_content和match_parent的区别
Cb123456
match_parentfill_parent
fill_parent、wrap_content和match_parent的区别:
1)fill_parent
设置一个构件的布局为fill_parent将强制性地使构件扩展,以填充布局单元内尽可能多的空间。这跟Windows控件的dockstyle属性大体一致。设置一个顶部布局或控件为fill_parent将强制性让它布满整个屏幕。
2) wrap_conte
- 网页自适应设计
天子之骄
htmlcss响应式设计页面自适应
网页自适应设计
网页对浏览器窗口的自适应支持变得越来越重要了。自适应响应设计更是异常火爆。再加上移动端的崛起,更是如日中天。以前为了适应不同屏幕分布率和浏览器窗口的扩大和缩小,需要设计几套css样式,用js脚本判断窗口大小,选择加载。结构臃肿,加载负担较大。现笔者经过一定时间的学习,有所心得,故分享于此,加强交流,共同进步。同时希望对大家有所
- [sql server] 分组取最大最小常用sql
一炮送你回车库
SQL Server
--分组取最大最小常用sql--测试环境if OBJECT_ID('tb') is not null drop table tb;gocreate table tb( col1 int, col2 int, Fcount int)insert into tbselect 11,20,1 union allselect 11,22,1 union allselect 1
- ImageIO写图片输出到硬盘
3213213333332132
javaimage
package awt;
import java.awt.Color;
import java.awt.Font;
import java.awt.Graphics;
import java.awt.image.BufferedImage;
import java.io.File;
import java.io.IOException;
import javax.imagei
- 自己的String动态数组
宝剑锋梅花香
java动态数组数组
数组还是好说,学过一两门编程语言的就知道,需要注意的是数组声明时需要把大小给它定下来,比如声明一个字符串类型的数组:String str[]=new String[10]; 但是问题就来了,每次都是大小确定的数组,我需要数组大小不固定随时变化怎么办呢? 动态数组就这样应运而生,龙哥给我们讲的是自己用代码写动态数组,并非用的ArrayList 看看字符
- pinyin4j工具类
darkranger
.net
pinyin4j工具类Java工具类 2010-04-24 00:47:00 阅读69 评论0 字号:大中小
引入pinyin4j-2.5.0.jar包:
pinyin4j是一个功能强悍的汉语拼音工具包,主要是从汉语获取各种格式和需求的拼音,功能强悍,下面看看如何使用pinyin4j。
本人以前用AscII编码提取工具,效果不理想,现在用pinyin4j简单实现了一个。功能还不是很完美,
- StarUML学习笔记----基本概念
aijuans
UML建模
介绍StarUML的基本概念,这些都是有效运用StarUML?所需要的。包括对模型、视图、图、项目、单元、方法、框架、模型块及其差异以及UML轮廓。
模型、视与图(Model, View and Diagram)
&
- Activiti最终总结
avords
Activiti id 工作流
1、流程定义ID:ProcessDefinitionId,当定义一个流程就会产生。
2、流程实例ID:ProcessInstanceId,当开始一个具体的流程时就会产生,也就是不同的流程实例ID可能有相同的流程定义ID。
3、TaskId,每一个userTask都会有一个Id这个是存在于流程实例上的。
4、TaskDefinitionKey和(ActivityImpl activityId
- 从省市区多重级联想到的,react和jquery的差别
bee1314
jqueryUIreact
在我们的前端项目里经常会用到级联的select,比如省市区这样。通常这种级联大多是动态的。比如先加载了省,点击省加载市,点击市加载区。然后数据通常ajax返回。如果没有数据则说明到了叶子节点。 针对这种场景,如果我们使用jquery来实现,要考虑很多的问题,数据部分,以及大量的dom操作。比如这个页面上显示了某个区,这时候我切换省,要把市重新初始化数据,然后区域的部分要从页面
- Eclipse快捷键大全
bijian1013
javaeclipse快捷键
Ctrl+1 快速修复(最经典的快捷键,就不用多说了)Ctrl+D: 删除当前行 Ctrl+Alt+↓ 复制当前行到下一行(复制增加)Ctrl+Alt+↑ 复制当前行到上一行(复制增加)Alt+↓ 当前行和下面一行交互位置(特别实用,可以省去先剪切,再粘贴了)Alt+↑ 当前行和上面一行交互位置(同上)Alt+← 前一个编辑的页面Alt+→ 下一个编辑的页面(当然是针对上面那条来说了)Alt+En
- js 笔记 函数
征客丶
JavaScript
一、函数的使用
1.1、定义函数变量
var vName = funcation(params){
}
1.2、函数的调用
函数变量的调用: vName(params);
函数定义时自发调用:(function(params){})(params);
1.3、函数中变量赋值
var a = 'a';
var ff
- 【Scala四】分析Spark源代码总结的Scala语法二
bit1129
scala
1. Some操作
在下面的代码中,使用了Some操作:if (self.partitioner == Some(partitioner)),那么Some(partitioner)表示什么含义?首先partitioner是方法combineByKey传入的变量,
Some的文档说明:
/** Class `Some[A]` represents existin
- java 匿名内部类
BlueSkator
java匿名内部类
组合优先于继承
Java的匿名类,就是提供了一个快捷方便的手段,令继承关系可以方便地变成组合关系
继承只有一个时候才能用,当你要求子类的实例可以替代父类实例的位置时才可以用继承。
在Java中内部类主要分为成员内部类、局部内部类、匿名内部类、静态内部类。
内部类不是很好理解,但说白了其实也就是一个类中还包含着另外一个类如同一个人是由大脑、肢体、器官等身体结果组成,而内部类相
- 盗版win装在MAC有害发热,苹果的东西不值得买,win应该不用
ljy325
游戏applewindowsXPOS
Mac mini 型号: MC270CH-A RMB:5,688
Apple 对windows的产品支持不好,有以下问题:
1.装完了xp,发现机身很热虽然没有运行任何程序!貌似显卡跑游戏发热一样,按照那样的发热量,那部机子损耗很大,使用寿命受到严重的影响!
2.反观安装了Mac os的展示机,发热量很小,运行了1天温度也没有那么高
&nbs
- 读《研磨设计模式》-代码笔记-生成器模式-Builder
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
/**
* 生成器模式的意图在于将一个复杂的构建与其表示相分离,使得同样的构建过程可以创建不同的表示(GoF)
* 个人理解:
* 构建一个复杂的对象,对于创建者(Builder)来说,一是要有数据来源(rawData),二是要返回构
- JIRA与SVN插件安装
chenyu19891124
SVNjira
JIRA安装好后提交代码并要显示在JIRA上,这得需要用SVN的插件才能看见开发人员提交的代码。
1.下载svn与jira插件安装包,解压后在安装包(atlassian-jira-subversion-plugin-0.10.1)
2.解压出来的包里下的lib文件夹下的jar拷贝到(C:\Program Files\Atlassian\JIRA 4.3.4\atlassian-jira\WEB
- 常用数学思想方法
comsci
工作
对于搞工程和技术的朋友来讲,在工作中常常遇到一些实际问题,而采用常规的思维方式无法很好的解决这些问题,那么这个时候我们就需要用数学语言和数学工具,而使用数学工具的前提却是用数学思想的方法来描述问题。。下面转帖几种常用的数学思想方法,仅供学习和参考
函数思想
把某一数学问题用函数表示出来,并且利用函数探究这个问题的一般规律。这是最基本、最常用的数学方法
- pl/sql集合类型
daizj
oracle集合typepl/sql
--集合类型
/*
单行单列的数据,使用标量变量
单行多列数据,使用记录
单列多行数据,使用集合(。。。)
*集合:类似于数组也就是。pl/sql集合类型包括索引表(pl/sql table)、嵌套表(Nested Table)、变长数组(VARRAY)等
*/
/*
--集合方法
&n
- [Ofbiz]ofbiz初用
dinguangx
电商ofbiz
从github下载最新的ofbiz(截止2015-7-13),从源码进行ofbiz的试用
1. 加载测试库
ofbiz内置derby,通过下面的命令初始化测试库
./ant load-demo (与load-seed有一些区别)
2. 启动内置tomcat
./ant start
或
./startofbiz.sh
或
java -jar ofbiz.jar
&
- 结构体中最后一个元素是长度为0的数组
dcj3sjt126com
cgcc
在Linux源代码中,有很多的结构体最后都定义了一个元素个数为0个的数组,如/usr/include/linux/if_pppox.h中有这样一个结构体: struct pppoe_tag { __u16 tag_type; __u16 tag_len; &n
- Linux cp 实现强行覆盖
dcj3sjt126com
linux
发现在Fedora 10 /ubutun 里面用cp -fr src dest,即使加了-f也是不能强行覆盖的,这时怎么回事的呢?一两个文件还好说,就输几个yes吧,但是要是n多文件怎么办,那还不输死人呢?下面提供三种解决办法。 方法一
我们输入alias命令,看看系统给cp起了一个什么别名。
[root@localhost ~]# aliasalias cp=’cp -i’a
- Memcached(一)、HelloWorld
frank1234
memcached
一、简介
高性能的架构离不开缓存,分布式缓存中的佼佼者当属memcached,它通过客户端将不同的key hash到不同的memcached服务器中,而获取的时候也到相同的服务器中获取,由于不需要做集群同步,也就省去了集群间同步的开销和延迟,所以它相对于ehcache等缓存来说能更好的支持分布式应用,具有更强的横向伸缩能力。
二、客户端
选择一个memcached客户端,我这里用的是memc
- Search in Rotated Sorted Array II
hcx2013
search
Follow up for "Search in Rotated Sorted Array":What if duplicates are allowed?
Would this affect the run-time complexity? How and why?
Write a function to determine if a given ta
- Spring4新特性——更好的Java泛型操作API
jinnianshilongnian
spring4generic type
Spring4新特性——泛型限定式依赖注入
Spring4新特性——核心容器的其他改进
Spring4新特性——Web开发的增强
Spring4新特性——集成Bean Validation 1.1(JSR-349)到SpringMVC
Spring4新特性——Groovy Bean定义DSL
Spring4新特性——更好的Java泛型操作API
Spring4新
- CentOS安装JDK
liuxingguome
centos
1、行卸载原来的:
[root@localhost opt]# rpm -qa | grep java
tzdata-java-2014g-1.el6.noarch
java-1.7.0-openjdk-1.7.0.65-2.5.1.2.el6_5.x86_64
java-1.6.0-openjdk-1.6.0.0-11.1.13.4.el6.x86_64
[root@localhost
- 二分搜索专题2-在有序二维数组中搜索一个元素
OpenMind
二维数组算法二分搜索
1,设二维数组p的每行每列都按照下标递增的顺序递增。
用数学语言描述如下:p满足
(1),对任意的x1,x2,y,如果x1<x2,则p(x1,y)<p(x2,y);
(2),对任意的x,y1,y2, 如果y1<y2,则p(x,y1)<p(x,y2);
2,问题:
给定满足1的数组p和一个整数k,求是否存在x0,y0使得p(x0,y0)=k?
3,算法分析:
(
- java 随机数 Math与Random
SaraWon
javaMathRandom
今天需要在程序中产生随机数,知道有两种方法可以使用,但是使用Math和Random的区别还不是特别清楚,看到一篇文章是关于的,觉得写的还挺不错的,原文地址是
http://www.oschina.net/question/157182_45274?sort=default&p=1#answers
产生1到10之间的随机数的两种实现方式:
//Math
Math.roun
- oracle创建表空间
tugn
oracle
create temporary tablespace TXSJ_TEMP
tempfile 'E:\Oracle\oradata\TXSJ_TEMP.dbf'
size 32m
autoextend on
next 32m maxsize 2048m
extent m
- 使用Java8实现自己的个性化搜索引擎
yangshangchuan
javasuperword搜索引擎java8全文检索
需要对249本软件著作实现句子级别全文检索,这些著作均为PDF文件,不使用现有的框架如lucene,自己实现的方法如下:
1、从PDF文件中提取文本,这里的重点是如何最大可能地还原文本。提取之后的文本,一个句子一行保存为文本文件。
2、将所有文本文件合并为一个单一的文本文件,这样,每一个句子就有一个唯一行号。
3、对每一行文本进行分词,建立倒排表,倒排表的格式为:词=包含该词的总行数N=行号