Evaluate the Malignancy of Pulmonary Nodules Using the 3D Deep Leaky Noisy-or Network 论文阅读
Kaggle上2017年肺结节检测比赛第一名的算法,其代码的github通道
背景
肺结节比赛采用的图像是CT,其中肺结节的影像学表现如下:
1. 和正常组织具有相同的HU值,但是具有不同的形状。
2. 支气管和血管是连续管腔,在根源较厚,在分支处较细;而肺结节一般呈球形。
3. 肺结节有很多类别,具有不同大小和形状,与癌症的相关性也不同。
医生对一位病人的正常阅片时间是10分钟。自动化检测算法的难点如下:
- 肺结节是3D检测问题。目前主流GPU显存不足以支持整幅图像的输入。之前有文章使用2D的RPN在2D slice上提取proposal,然后整合不同层的结果,形成3D proposal。此外,3D的标注困难也较大(个人觉得label上3D的获取应该是从2D拼接起来的,应该不会太难),模型容易过拟合。
- 不同肺结节形状差异大,与正常组织难以区分,如下图。有时候医生也难以确定。
- 肺结节和肺癌之间的关系复杂。存在肺结节并不一定是肺癌,发生肺癌也不一定能找到可见肺结节。因此,判断发生肺癌的概率和找到的多个肺结节有关,这就成为了多实例学习问题(Multiple Insatnce Learning)。
为了解决上述问题,文章提出以下解决方案:
1. patch-based 3D RPN[detection network]用于预测肺结节的bbox
- 数据增强来防止过拟合
- 低阈值来找出所有可能结节
- top-5 结节用于癌症可能性预测
2. leaky noisy-or 模型用于整合top-5结节的分数[classification network]来计算癌症可能性
- noisy-or模型假设一个事件可以由多个因素引起,其中任何一个因素的发生都可能独立的导致该事件发生。也就是说任何一个肺结节都可能是肺癌。
- leaky noisy-or模型认为即使没有任何因素发生,该事件仍然可能由leakage probabily导致发生。
- classification network 也是3D的神经网络,并且和detection network共用特征提取器。在训练的时候采用交替训练方式。
数据集
模型训练基于以下两个数据集:
1. LUNA16:这个数据集上有888个病人,1186个标记的肺结节。
2. DSB:这个数据集上1397个病人作为训练,198个病人作为验证,506个病人作为测试。作者人工标记了训练集中的754个结节和验证集中的78个结节。
关于为什么要人工标记?尽管都是肺结节检测,但是LUNA16中含有大量小的结节,其肺结节平均直径是8.31mm,而临床经验认为6mm以下的肺结节是没有危险的。而DSB中大的结节较多,平均直径是13.68 mm,此外DSB中有很多结节与主支气管相连,这是LUNA16中很少见的。因此如果直接利用LUNA16作为训练数据,效果不好。为此,作者去掉了LUNA中所有6mm以下的人工标记。
本文算法
预处理
CT图像是对整个胸腔的扫描,包含了肺部以外的组织和器官,为了减少其他组织的干扰,作者首先要提取出肺部区域。
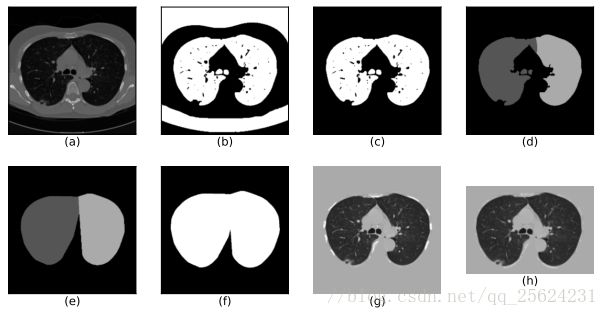
1. Mask extraction:作者首先在2D slice上通过阈值得到了肺部以及周围较暗部分的mask,如图中(b)所示,然后通过连通性分析,去除30mm以下的(肺部是中间一大块,不会这么小)以及离心率大于0.99的(个人感觉一般线状伪影可能是这样)。然后在3D中保留不在边际的(去除周围较暗部分)以及容积在0.68-7.5L之间的。经过这样处理后,一般就能够保留肺部区域,还有一些异常的话,可以利用肺部处于图像中间的特性进行排除。结果如图(c)所示。
2. Convex hull & dilation: 一些结节如果与肺的外壁相连,则不会出现在上述步骤提取的mask中,这种情况是需要避免的。如果想要把他们包含进来,直接的方式是对整个mask进行凸包,不过这样就会引入骨骼那一块,影响比较大。所以根据肺的结构,将其分成两块,如图(d)所示。然后分别对两块进行凸包,并且向外10pixels的膨胀。得到图(f)。不过作者还提了一句,对于肺部底部,由于其形状接近U型,凸包后如果面积大于原始的1.5倍,则放弃凸包,避免引入过多其他组织。
3. 灰度标准化:首先把HU值线性变换到0-255。mask以外的像素灰度都设成170(正常组织的灰度)。膨胀10像素区域中如果灰度高于210,也设成170(为了防止骨头被当成结节)。
- 到这里觉得挺感慨的,以上做法中涉及到太多经验值参数,自己在项目中每次设经验值的时候都要纠结纠结是不是最优的,会不会在异常情况下出现奇葩效果。是不是应该大胆一点?
- 还有目前常用的方法是把图像直接输入,从这篇文章来看,如果预处理能够减少神经网络的处理量,去除可能的误导,对效果应该是有很多提升的。
3D CNN for Nodule Detection
检测网络以modifid U-net作为基础,因为是二分类问题,所以提出region proposal后不需要继续分类,因此是one stage。
输入:
考虑到GPU容量,网络采用128*128*128*1的输入大小,随机选择两类patches。其中70%是patches包含至少一个结节,30%是不含结节的(为了保证负样本的收敛)。其中越界填充值为170,结节至少与边界有12pixels的距离。
数据增强方法为随机所有翻转以及resize in 0.8和1.15。
U-net结构能够很好解决结节不同大小的问题。接下来逐步看网络的流程:
1. 输入:128*128*128*1
2. K:两个卷积层,每个卷积层有24个3*3*3的卷积核,padding=1。因此输出为128*128*128*24的feature maps。
3. R:残差块,如图(b)所示。由三个残差单元组成。每个残差单元由卷积,BN,Relu,卷积,BN构成,其中卷积核的大小都是3*3*3。这里面需要注意的是残差前后channels由24变成了32。因此不是y = F(x)+x,而是y=F(x)+Wx,其中W是卷积运算用于改变channels。此处还省略了一个2*2*2 with stride=2 MaxPooling。
4. D:转置卷积,原始U-net中设置成不可学习,注意一下文章此处在实现时的设置。
5. C:融合层。除了和U-net一样,此处作者还添加了h*l*w*3的location information。(个人觉得医学图像中绝对位置对于诊断还是很重要的,之前也尝试过加入位置信息,不过是在输入层中加,作者的这个加法更加合理,因为这种显式的信息应当靠近网络的后端,能够对分割结果起到更大的影响。)具体方法作者说的不是很清楚,我觉得应该是patchs在原始图像中的相对坐标,然后resize到 32*32*32*3的大小。所以融合层的输入的channels是131(64+64+3),然后通过1*1*1的卷积将channels变换到15进行输出。
6. 输出:32*32*32*3*5(此处维度变化是由resize得到的,但是这显然和128维的不对应,倒是和15对应,所以我觉得上一层最后提到的1*1*1卷积是在输出前做的,而不是每个融合层做)。然后3代表每个像素点3个anchor,5代表(o,dx,dy,dz,dr),也就是概率,三维坐标和bbox的直径大小。
损失函数
真值的label记为(Gx,Gy,Gz,Gr),每个Anchor的值记为(Ax,Ay,Az,Ar)。和Faster RCNN中类似,IoU大于0.5的记为P,小于0.02的记为F,其他忽略。
所以其分类损失为:
Bbox回归的标签生成:
总的回归损失为:
其中S是smoothes L1-norm function。
最后网络的训练loss是:
正负样本
1. 正样本
对于大的结节,会出现很多P anchors,这时随机选择一个。
结节在大小上的分布不均衡,为了加强对于大结节的关注,在sample时候对于30mm以上结节的采样频率增加1倍,对于40mm以上结节增加5倍。
2. 负样本
对于anchors来说,负样本还是远远多于正样本。这是RPN中的常见问题。采用hard negative mining来解决。核心思想是控制负样本的数目。
测试阶段
输入: 208*208*208*1 overlap=32 pixels。
输出采用非极大值抑制。
这种overlap crop理解简单,但是自己一直没有写代码,尤其是在训练的时候。
之前居然对于不同大小的图片不知道怎么办,今天看了文章,突然知道怎么coding。
Cancer Classification
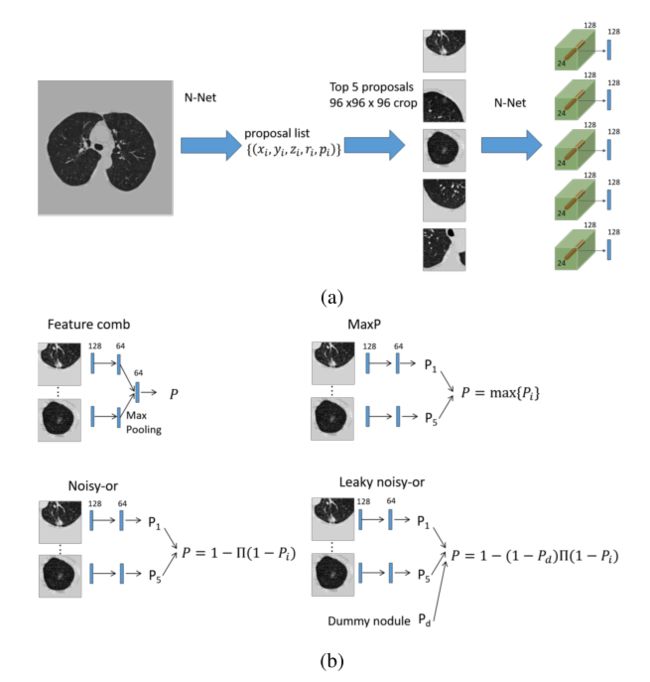
在获取结节的region proposal后,根据scores取前5。在训练阶段,为了数据增强,可以随机取。
对输入的每个结节进行肺癌可能性评价网络复用了之前的U-net,如图(a)所示,具体如下:
1. 输入:取96*96*96*1的patch。这里我有个问题,既然只用了中心点的信息,干嘛之前还要训练dr这个参数。为了提高模型的特征提取能力?
2. 取最后一个卷积层的输出(24*24*24*128), 将中心的2*2*2的值提取出来(2*2*2*128 = 1024),经过max-pooling得到128维的特征。感觉这里的信息是这8个点(基本代表了该结节)的位置和scores
此时我们获得了5个128维的特征,如何得到最后的癌症scores。作者探究了四种方法,最好的还是leaky noisy-or模型。
1. 对于每个输入的128维特征,利用双层的ANN得到P
2. 对于5个P,采用如下公式结合:
所以此时loss function应该就是计算的p和label的交叉熵。不过这样的话,每个pi会代表各自是癌症的概率吗?
训练策略
文章提到结节的bounding box事先生成好,这是什么意思不是很明白。是指RPN网络已经训练好了?那后面的联合训练只是Refine?
文章说为了避免过拟合,有两个方式:一个是数据增强,另一个是正则化。
数据增强包括:1)随机翻转 2)resize 3)旋转 4)平移。
正则化:RPN和分类互相正则化。在每一次训练阶段,有一个detector的eopch和一个classifer的epoch。
由于batch size = 2,所以训练不稳定,采用l2 norm的gradient clipping。
由于迭代训练,所以BN的参数需要特别处理。

