TCGA是什么?
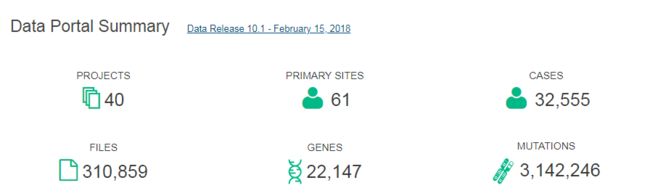
由美国05年发起的癌症和肿瘤基因图谱(TCGA)计划,旨在应用基因组分析技术研究癌症中的基因组变化,做了大规模的基因组测序,样本量过万,包含了三十多种癌症,其中尤其宝贵的是这些样本都有很详细的预后随访信息,08年出了第一篇文章,之后陆陆续续各种大文章出来。
TCGA包含了哪些数据?
1、临床样本信息:Biospecimen、Clinical
2、测序数据:主要采用了RNA-Seq、WXS、miRNA-Seq、Genotyping
Array、Methylation Array这五种方式对样本进行测序。
对测序数据按照一定的分析程度进行分层,共分为四层:level1、level2、level3、level4,按照不同的层次的数据提供给科研人员使用,level3、level4的数据一般都开放下载的,level1是最原始的数据,level2是做了进一步的处理的这些数据一般是不开放的,需要申请才能下载(申请也挺难)。
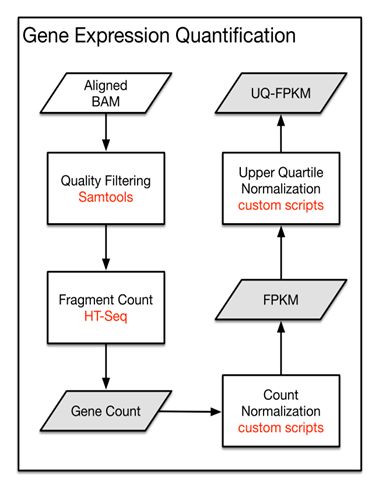
TCGA中的RNA-Seq数据
顾名思义就是转录组测序
•TCGA上的转录组数据采用的是全转录组测序,其中包含了各种非编码RNA,所以一般下载的RNA-Seq数据中包含了lncRNA、mRNA、假基因等等;
•目前可以公开下载的是RNA-Seq定量表达数据,主要三种形式:HT-Seq-FPKM,HT-Seq-UQ-FPKM,HT-Seq-Counts;
TCGA用的gff文件是gencode.v22.annotation.gtf
名词解释
•FPKM:用来衡量转录本表达丰度的一种量度方式;
•Counts:测序的reads中比对到某个基因上的计数;
•UQ-FPKM:通过上四分位点进行标准化后的FPKM;
•gff文件:用来描述基因组上各种基因、转录本等信息的文件。
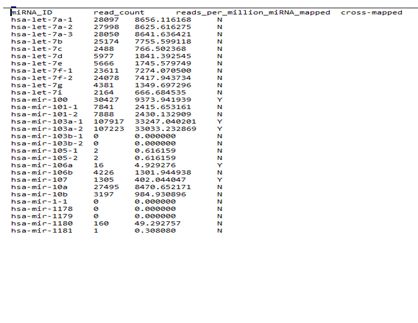
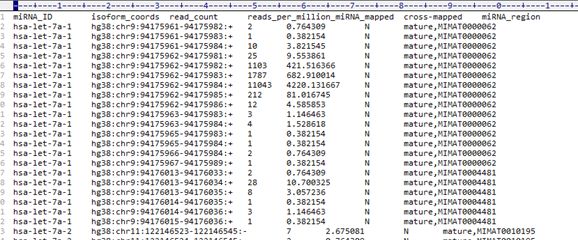
TCGA中的miRNA-Seq数据
•miRNA:miRNA
是一类由内源基因编码的长度约为22 个核苷酸的非编码单链RNA分子,生物中非常重要的一类非编码小RNA,其在生物体的调控中具有非常重要的作用,在人中大约三分之一的基因受到miRNA的调控;
•TCGA提供了miRNA-Seq的测序数据结果,采用的数据库背景为miRBase v21;
•目前公开提供下载的数据主要有两种:miRNA
Expression Quantification、Isoform Expression Quantification,其中Isoform Expression Quantification数据中包含了成熟体miRNA;
•分别提供了Counts和FPKM格式的定量数据。
名词解释
•pre-miRNA:前体miRNA,长度大约为70~90个碱基;pre-miRNA再经过Dicer酶酶切后,成为长约20~24nt的成熟miRNA;
•miRNA:成熟体miRNA,长度约20~24nt,由多种前体miRNA发展而来;
•miRNA与靶基因的关系:一般miRNA调控靶基因,降低基因的表达。
TCGA中的甲基化数据
•DNA甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达;
•TCGA提供了甲基化芯片数据,主要有两种一种是450k的,一种是27k的,一般450k的最为常用,数据主要以cpg位点为单位,一般我们认为在基因启动子区域上的甲基化对该基因的表达会产生影响;
•目前TCGA提供的公开下载的甲基化数据主要为level3的CpG位点的甲基化水平的数据。
名词解释
CpG位点:DNA序列上碱基为C或者G的位点,一般公认的甲基化只会发生在CpG位点上。
•基因启动子区域:目前基因启动子区域没有统一的说法,一般我们认为基因的TSS的上游2kb到下游500bp之间。
•CpG岛:目前没有金标准,一般认为CpG位点显著聚集的区域称之为CpG岛,CpG岛区域往往整体甲基化水平较低。常出现在基因的启动子区域和外显子区域。
•甲基化与基因的关系:基因启动子区域的高甲基化将影响其下游基因的表达,呈负相关。
TCGA中的临床信息
•TCGA中提供了丰富的临床随访信息,其中包括用药、复发、年龄、生存等等共包含了一百多项。
•TCGA提供的临床信息数据格式为xml,使用极不方便,需要手动提取。
•常用的临床信息主要有:
1、年龄
2、性别
3、T N M Stage Grade
4、复发 复发时间
5、总生存期
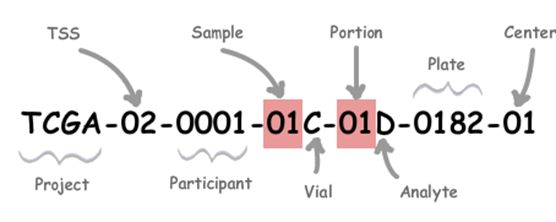
TCGA的样本命名
•TCGA对每一个病人都有一个单独的ID如TCGA-02-0001,这个ID在TCGA数据库中都是通用的,根据这个ID,在不同类型的数据中我们能够找到同一个病人的,其中包括临床随访信息。
•Sample:病人的不同的采样部位有着不同的编码,比如01表示癌组织,10表示癌旁组织,一般01到09表示肿瘤部位,10及以上表示正常对照。
•vial表示组织的顺序,A、B、C、D;比如B表示石蜡包埋组织。
•Portion表示同一组织不同的组分;
•Analyte表示分析的分子类型,D表示DNA;
•Plate表示孔板顺序,0182表示96孔板;
•Center表示检测中心。
TCGA数据的下载
•TCGA官方提供了gdc_client下载工具,通过gdc_client下载工具能下载TCGA公开的数据,但是下载的数据是单个样本的,需要自己手动合并。
•TCGA官方提供了gdc_api来进行数据下载,大多数第三方的包都是基于gdc_api的,比如R包TCGAbiolinks、生信人简易下载工具。
•第三方数据库提供的数据下载比如GDAC的Firehose,UCSC的Cancer browser。
•下载难度系数:gdc_client>TCGAbiolinks>Firehose>桑格助手>UCSC
•使用灵活性:gdc_client>桑格助手>TCGAbiolinks>Firehose>UCSC
何为桑格助手?
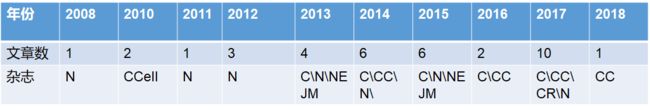
TCGA大文章一览
•TCGA数据从2008年开始到现在陆陆续续的出来了很多的CNS级别的大文章
•文章列表可以从TCGA Research NetworkPublications获得。
•这些文章以分子分型、全基因组景观,多层次关联分析为主。
下期介绍GEO,如果觉得文章内容不错,请点喜欢