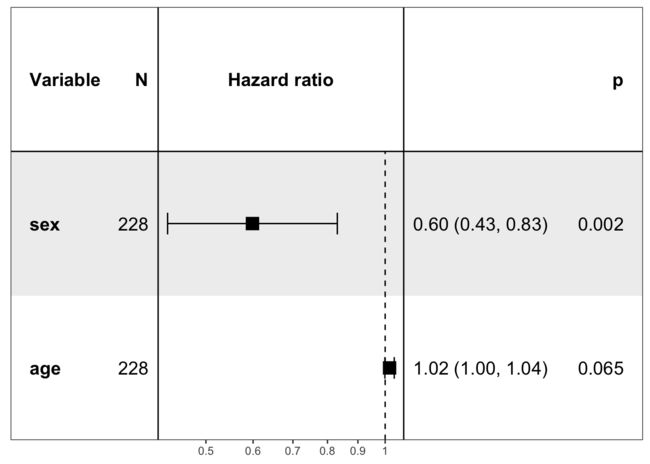
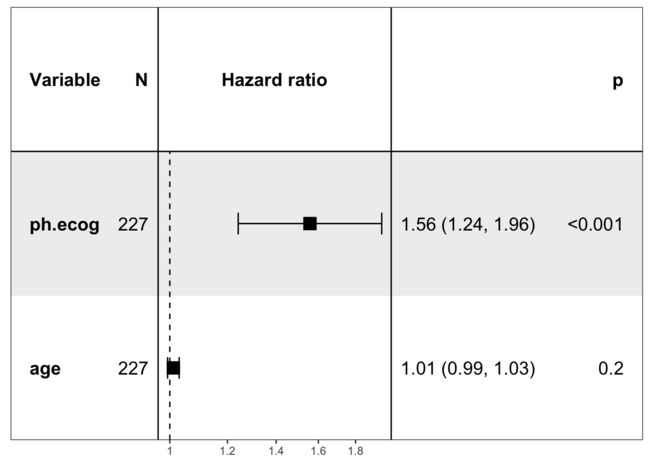
一、什么是宏函数?通过宏定义的函数是宏函数。如下,编译器在预处理阶段会将Add(x,y)替换为((x)*(y))#defineAdd(x,y)((x)*(y))#defineAdd(x,y)((x)*(y))intmain(){inta=10;intb=20;intd=10;intc=Add(a+d,b)*2;cout<
C语言如何定义宏函数?
小九格物
c语言
在C语言中,宏函数是通过预处理器定义的,它在编译之前替换代码中的宏调用。宏函数可以模拟函数的行为,但它们不是真正的函数,因为它们在编译时不会进行类型检查,也不会分配存储空间。宏函数的定义通常使用#define指令,后面跟着宏的名称和参数列表,以及宏展开后的代码。宏函数的定义方式:1.基本宏函数:这是最简单的宏函数形式,它直接定义一个表达式。#defineSQUARE(x)((x)*(x))2.带参
c++ 的iostream 和 c++的stdio的区别和联系
黄卷青灯77
c++算法开发语言iostreamstdio
在C++中,iostream和C语言的stdio.h都是用于处理输入输出的库,但它们在设计、用法和功能上有许多不同。以下是两者的区别和联系:区别1.编程风格iostream(C++风格):C++标准库中的输入输出流类库,支持面向对象的输入输出操作。典型用法是cin(输入)和cout(输出),使用>操作符来处理数据。更加类型安全,支持用户自定义类型的输入输出。#includeintmain(){in
《投行人生》读书笔记
小蘑菇的树洞
《投行人生》----作者詹姆斯-A-朗德摩根斯坦利副主席40年的职业洞见-很短小精悍的篇幅,比较适合初入职场的新人。第一部分成功的职业生涯需要规划1.情商归为适应能力分享与协作同理心适应能力,更多的是自我意识,你有能力识别自己的情并分辨这些情绪如何影响你的思想和行为。2.对于初入职场的人的建议,细节,截止日期和数据很重要截止日期,一种有效的方法是请老板为你所有的任务进行优先级排序。和老板喝咖啡的好
Long类型前后端数据不一致
igotyback
前端
响应给前端的数据浏览器控制台中response中看到的Long类型的数据是正常的到前端数据不一致前后端数据类型不匹配是一个常见问题,尤其是当后端使用Java的Long类型(64位)与前端JavaScript的Number类型(最大安全整数为2^53-1,即16位)进行数据交互时,很容易出现精度丢失的问题。这是因为JavaScript中的Number类型无法安全地表示超过16位的整数。为了解决这个问
如何在 Fork 的 GitHub 项目中保留自己的修改并同步上游更新?github_fork_update
iBaoxing
github
如何在Fork的GitHub项目中保留自己的修改并同步上游更新?在GitHub上Fork了一个项目后,你可能会对项目进行一些修改,同时原作者也在不断更新。如果想要在保留自己修改的基础上,同步原作者的最新更新,很多人会不知所措。本文将详细讲解如何在不丢失自己改动的情况下,将上游仓库的更新合并到自己的仓库中。问题描述假设你在GitHub上Fork了一个项目,并基于该项目做了一些修改,随后你发现原作者对
2021-08-26
影幽
在生活中,女人与男人的感悟往往有所不同。人生最大的舞台就是生活,大幕随时都可能拉开,关键是你愿不愿意表演都无法躲避。在生活中,遇事不要急躁,不要急于下结论,尤其生气时不要做决断,要学会换位思考,大事化小小事化了,把复杂的事情尽量简单处理,千万不要把简单的事情复杂化。永远不要扭曲,别人善意,无药可救。昨天是张过期的支票,明天是张信用卡,只有今天才是现金,要善加利用!执着的攀登者不必去与别人比较自己的
消息中间件有哪些常见类型
xmh-sxh-1314
java
消息中间件根据其设计理念和用途,可以大致分为以下几种常见类型:点对点消息队列(Point-to-PointMessagingQueues):在这种模型中,消息被发送到特定的队列中,消费者从队列中取出并处理消息。队列中的消息只能被一个消费者消费,消费后即被删除。常见的实现包括IBM的MQSeries、RabbitMQ的部分使用场景等。适用于任务分发、负载均衡等场景。发布/订阅消息模型(Pub/Sub
html 中如何使用 uniapp 的部分方法
某公司摸鱼前端
htmluni-app前端
示例代码:Documentconsole.log(window);效果展示:好了,现在就可以uni.使用相关的方法了
ArcGIS栅格计算器常见公式(赋值、0和空值的转换、补充栅格空值)
研学随笔
arcgis经验分享
我们在使用ArcGIS时通常经常用到栅格计算器,今天主要给大家介绍我日常中经常用到的几个公式,供大家参考学习。将特定值(-9999)赋值为0,例如-9999.Con("raster"==-9999,0,"raster")2.给空值赋予特定的值(如0)Con(IsNull("raster"),0,"raster")3.将特定的栅格值(如1)赋值为空值,其他保留原值SetNull("raster"==
高级编程--XML+socket练习题
masa010
java开发语言
1.北京华北2114.8万人上海华东2,500万人广州华南1292.68万人成都华西1417万人(1)使用dom4j将信息存入xml中(2)读取信息,并打印控制台(3)添加一个city节点与子节点(4)使用socketTCP协议编写服务端与客户端,客户端输入城市ID,服务器响应相应城市信息(5)使用socketTCP协议编写服务端与客户端,客户端要求用户输入city对象,服务端接收并使用dom4j
水平垂直居中的几种方法(总结)
LJ小番茄
CSS_玄学语言htmljavascript前端csscss3
1.使用flexbox的justify-content和align-items.parent{display:flex;justify-content:center;/*水平居中*/align-items:center;/*垂直居中*/height:100vh;/*需要指定高度*/}2.使用grid的place-items:center.parent{display:grid;place-item
《大清方方案》| 第二话
谁佐清欢
和珅究竟说了些什么?竟能令堂堂九五之尊龙颜失色!此处暂且按下不表;单说这位乾隆皇帝,果真不愧是康熙从小带过的,一旦决定了要做的事,便杀伐决断毫不含糊。他当即亲自拟旨,着令和珅为钦差大臣,全权负责处理方方事件,并钦赐尚方宝剑,遇急则三品以下官员可先斩后奏。和珅身负皇上重托,岂敢有半点怠慢,当夜即率领相关人等,马不停蹄杀奔江汉。这一路上,和珅的几位幕僚一直在商讨方方事件的处置方案。有位年轻幕僚建议快刀
【一起学Rust | 设计模式】习惯语法——使用借用类型作为参数、格式化拼接字符串、构造函数
广龙宇
一起学Rust#Rust设计模式rust设计模式开发语言
提示:文章写完后,目录可以自动生成,如何生成可参考右边的帮助文档文章目录前言一、使用借用类型作为参数二、格式化拼接字符串三、使用构造函数总结前言Rust不是传统的面向对象编程语言,它的所有特性,使其独一无二。因此,学习特定于Rust的设计模式是必要的。本系列文章为作者学习《Rust设计模式》的学习笔记以及自己的见解。因此,本系列文章的结构也与此书的结构相同(后续可能会调成结构),基本上分为三个部分
回溯 Leetcode 332 重新安排行程
mmaerd
Leetcode刷题学习记录leetcode算法职场和发展
重新安排行程Leetcode332学习记录自代码随想录给你一份航线列表tickets,其中tickets[i]=[fromi,toi]表示飞机出发和降落的机场地点。请你对该行程进行重新规划排序。所有这些机票都属于一个从JFK(肯尼迪国际机场)出发的先生,所以该行程必须从JFK开始。如果存在多种有效的行程,请你按字典排序返回最小的行程组合。例如,行程[“JFK”,“LGA”]与[“JFK”,“LGB
Python数据分析与可视化实战指南
William数据分析
pythonpython数据
在数据驱动的时代,Python因其简洁的语法、强大的库生态系统以及活跃的社区,成为了数据分析与可视化的首选语言。本文将通过一个详细的案例,带领大家学习如何使用Python进行数据分析,并通过可视化来直观呈现分析结果。一、环境准备1.1安装必要库在开始数据分析和可视化之前,我们需要安装一些常用的库。主要包括pandas、numpy、matplotlib和seaborn等。这些库分别用于数据处理、数学
C#中使用split分割字符串
互联网打工人no1
c#
1、用字符串分隔:usingSystem.Text.RegularExpressions;stringstr="aaajsbbbjsccc";string[]sArray=Regex.Split(str,"js",RegexOptions.IgnoreCase);foreach(stringiinsArray)Response.Write(i.ToString()+"");输出结果:aaabbbc
Python中os.environ基本介绍及使用方法
鹤冲天Pro
#Pythonpython服务器开发语言
文章目录python中os.environos.environ简介os.environ进行环境变量的增删改查python中os.environ的使用详解1.简介2.key字段详解2.1常见key字段3.os.environ.get()用法4.环境变量的增删改查和判断是否存在4.1新增环境变量4.2更新环境变量4.3获取环境变量4.4删除环境变量4.5判断环境变量是否存在python中os.envi
第四天旅游线路预览——从换乘中心到喀纳斯湖
陟彼高冈yu
基于Googleearthstudio的旅游规划和预览旅游
第四天:从贾登峪到喀纳斯风景区入口,晚上住宿贾登峪;换乘中心有4路车,喀纳斯①号车,去喀纳斯湖,路程时长约5分钟;将上面的的行程安排进行动态展示,具体步骤见”Googleearthstudio进行动态轨迹显示制作过程“、“Googleearthstudio入门教程”和“Googleearthstudio进阶教程“相关内容,得到行程如下所示:Day4-2-480p
Python教程:一文了解使用Python处理XPath
旦莫
Python进阶python开发语言
目录1.环境准备1.1安装lxml1.2验证安装2.XPath基础2.1什么是XPath?2.2XPath语法2.3示例XML文档3.使用lxml解析XML3.1解析XML文档3.2查看解析结果4.XPath查询4.1基本路径查询4.2使用属性查询4.3查询多个节点5.XPath的高级用法5.1使用逻辑运算符5.2使用函数6.实战案例6.1从网页抓取数据6.1.1安装Requests库6.1.2代
Google earth studio 简介
陟彼高冈yu
旅游
GoogleEarthStudio是一个基于Web的动画工具,专为创作使用GoogleEarth数据的动画和视频而设计。它利用了GoogleEarth强大的三维地图和卫星影像数据库,使用户能够轻松地创建逼真的地球动画、航拍视频和动态地图可视化。网址为https://www.google.com/earth/studio/。GoogleEarthStudio是一个基于Web的动画工具,专为创作使用G
linux中sdl的使用教程,sdl使用入门
Melissa Corvinus
linux中sdl的使用教程
本文通过一个简单示例讲解SDL的基本使用流程。示例中展示一个窗口,窗口里面有个随机颜色快随机移动。当我们鼠标点击关闭按钮时间窗口关闭。基本步骤如下:1.初始化SDL并创建一个窗口。SDL_Init()初始化SDL_CreateWindow()创建窗口2.纹理渲染存储RGB和存储纹理的区别:比如一个从左到右由红色渐变到蓝色的矩形,用存储RGB的话就需要把矩形中每个点的具体颜色值存储下来;而纹理只是一
LLM 词汇表
落难Coder
LLMsNLP大语言模型大模型llama人工智能
Contextwindow“上下文窗口”是指语言模型在生成新文本时能够回溯和参考的文本量。这不同于语言模型训练时所使用的大量数据集,而是代表了模型的“工作记忆”。较大的上下文窗口可以让模型理解和响应更复杂和更长的提示,而较小的上下文窗口可能会限制模型处理较长提示或在长时间对话中保持连贯性的能力。Fine-tuning微调是使用额外的数据进一步训练预训练语言模型的过程。这使得模型开始表示和模仿微调数
Java序列化进阶篇
g21121
java序列化
1.transient
类一旦实现了Serializable 接口即被声明为可序列化,然而某些情况下并不是所有的属性都需要序列化,想要人为的去阻止这些属性被序列化,就需要用到transient 关键字。
escape()、encodeURI()、encodeURIComponent()区别详解
aigo
JavaScriptWeb
原文:http://blog.sina.com.cn/s/blog_4586764e0101khi0.html
JavaScript中有三个可以对字符串编码的函数,分别是: escape,encodeURI,encodeURIComponent,相应3个解码函数:,decodeURI,decodeURIComponent 。
下面简单介绍一下它们的区别
1 escape()函
ArcgisEngine实现对地图的放大、缩小和平移
Cb123456
添加矢量数据对地图的放大、缩小和平移Engine
ArcgisEngine实现对地图的放大、缩小和平移:
个人觉得是平移,不过网上的都是漫游,通俗的说就是把一个地图对象从一边拉到另一边而已。就看人说话吧.
具体实现:
一、引入命名空间
using ESRI.ArcGIS.Geometry;
using ESRI.ArcGIS.Controls;
二、代码实现.
Java集合框架概述
天子之骄
Java集合框架概述
集合框架
集合框架可以理解为一个容器,该容器主要指映射(map)、集合(set)、数组(array)和列表(list)等抽象数据结构。
从本质上来说,Java集合框架的主要组成是用来操作对象的接口。不同接口描述不同的数据类型。
简单介绍:
Collection接口是最基本的接口,它定义了List和Set,List又定义了LinkLi
旗正4.0页面跳转传值问题
何必如此
javajsp
跳转和成功提示
a) 成功字段非空forward
成功字段非空forward,不会弹出成功字段,为jsp转发,页面能超链接传值,传输变量时需要拼接。接拼接方式list.jsp?test="+strweightUnit+"或list.jsp?test="+weightUnit+&qu
全网唯一:移动互联网服务器端开发课程
cocos2d-x小菜
web开发移动开发移动端开发移动互联程序员
移动互联网时代来了! App市场爆发式增长为Web开发程序员带来新一轮机遇,近两年新增创业者,几乎全部选择了移动互联网项目!传统互联网企业中超过98%的门户网站已经或者正在从单一的网站入口转向PC、手机、Pad、智能电视等多端全平台兼容体系。据统计,AppStore中超过85%的App项目都选择了PHP作为后端程
Log4J通用配置|注意问题 笔记
7454103
DAOapachetomcatlog4jWeb
关于日志的等级 那些去 百度就知道了!
这几天 要搭个新框架 配置了 日志 记下来 !做个备忘!
#这里定义能显示到的最低级别,若定义到INFO级别,则看不到DEBUG级别的信息了~!
log4j.rootLogger=INFO,allLog
# DAO层 log记录到dao.log 控制台 和 总日志文件
log4j.logger.DAO=INFO,dao,C
SQLServer TCP/IP 连接失败问题 ---SQL Server Configuration Manager
darkranger
sqlcwindowsSQL ServerXP
当你安装完之后,连接数据库的时候可能会发现你的TCP/IP 没有启动..
发现需要启动客户端协议 : TCP/IP
需要打开 SQL Server Configuration Manager...
却发现无法打开 SQL Server Configuration Manager..??
解决方法: C:\WINDOWS\system32目录搜索framedyn.
[置顶] 做有中国特色的程序员
aijuans
程序员
从出版业说起 网络作品排到靠前的,都不会太难看,一般人不爱看某部作品也是因为不喜欢这个类型,而此人也不会全不喜欢这些网络作品。究其原因,是因为网络作品都是让人先白看的,看的好了才出了头。而纸质作品就不一定了,排行榜靠前的,有好作品,也有垃圾。 许多大牛都是写了博客,后来出了书。这些书也都不次,可能有人让为不好,是因为技术书不像小说,小说在读故事,技术书是在学知识或温习知识,有些技术书读得可
document.domain 跨域问题
avords
document
document.domain用来得到当前网页的域名。比如在地址栏里输入:javascript:alert(document.domain); //www.315ta.com我们也可以给document.domain属性赋值,不过是有限制的,你只能赋成当前的域名或者基础域名。比如:javascript:alert(document.domain = "315ta.com");
关于管理软件的一些思考
houxinyou
管理
工作好多看年了,一直在做管理软件,不知道是我最开始做的时候产生了一些惯性的思维,还是现在接触的管理软件水平有所下降.换过好多年公司,越来越感觉现在的管理软件做的越来越乱.
在我看来,管理软件不论是以前的结构化编程,还是现在的面向对象编程,不管是CS模式,还是BS模式.模块的划分是很重要的.当然,模块的划分有很多种方式.我只是以我自己的划分方式来说一下.
做为管理软件,就像现在讲究MVC这
NoSQL数据库之Redis数据库管理(String类型和hash类型)
bijian1013
redis数据库NoSQL
一.Redis的数据类型
1.String类型及操作
String是最简单的类型,一个key对应一个value,string类型是二进制安全的。Redis的string可以包含任何数据,比如jpg图片或者序列化的对象。
Set方法:设置key对应的值为string类型的value
Tomcat 一些技巧
征客丶
javatomcatdos
以下操作都是在windows 环境下
一、Tomcat 启动时配置 JAVA_HOME
在 tomcat 安装目录,bin 文件夹下的 catalina.bat 或 setclasspath.bat 中添加
set JAVA_HOME=JAVA 安装目录
set JRE_HOME=JAVA 安装目录/jre
即可;
二、查看Tomcat 版本
在 tomcat 安装目
【Spark七十二】Spark的日志配置
bit1129
spark
在测试Spark Streaming时,大量的日志显示到控制台,影响了Spark Streaming程序代码的输出结果的查看(代码中通过println将输出打印到控制台上),可以通过修改Spark的日志配置的方式,不让Spark Streaming把它的日志显示在console
在Spark的conf目录下,把log4j.properties.template修改为log4j.p
Haskell版冒泡排序
bookjovi
冒泡排序haskell
面试的时候问的比较多的算法题要么是binary search,要么是冒泡排序,真的不想用写C写冒泡排序了,贴上个Haskell版的,思维简单,代码简单,下次谁要是再要我用C写冒泡排序,直接上个haskell版的,让他自己去理解吧。
sort [] = []
sort [x] = [x]
sort (x:x1:xs)
| x>x1 = x1:so
java 路径 配置文件读取
bro_feng
java
这几天做一个项目,关于路径做如下笔记,有需要供参考。
取工程内的文件,一般都要用相对路径,这个自然不用多说。
在src统计目录建配置文件目录res,在res中放入配置文件。
读取文件使用方式:
1. MyTest.class.getResourceAsStream("/res/xx.properties")
2. properties.load(MyTest.
读《研磨设计模式》-代码笔记-简单工厂模式
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
package design.pattern;
/*
* 个人理解:简单工厂模式就是IOC;
* 客户端要用到某一对象,本来是由客户创建的,现在改成由工厂创建,客户直接取就好了
*/
interface IProduct {
SVN与JIRA的关联
chenyu19891124
SVN
SVN与JIRA的关联一直都没能装成功,今天凝聚心思花了一天时间整合好了。下面是自己整理的步骤:
一、搭建好SVN环境,尤其是要把SVN的服务注册成系统服务
二、装好JIRA,自己用是jira-4.3.4破解版
三、下载SVN与JIRA的插件并解压,然后拷贝插件包下lib包里的三个jar,放到Atlassian\JIRA 4.3.4\atlassian-jira\WEB-INF\lib下,再
JWFDv0.96 最新设计思路
comsci
数据结构算法工作企业应用公告
随着工作流技术的发展,工作流产品的应用范围也不断的在扩展,开始进入了像金融行业(我已经看到国有四大商业银行的工作流产品招标公告了),实时生产控制和其它比较重要的工程领域,而
vi 保存复制内容格式粘贴
daizj
vi粘贴复制保存原格式不变形
vi是linux中非常好用的文本编辑工具,功能强大无比,但对于复制带有缩进格式的内容时,粘贴的时候内容错位很严重,不会按照复制时的格式排版,vi能不能在粘贴时,按复制进的格式进行粘贴呢? 答案是肯定的,vi有一个很强大的命令可以实现此功能 。
在命令模式输入:set paste,则进入paste模式,这样再进行粘贴时
shell脚本运行时报错误:/bin/bash^M: bad interpreter 的解决办法
dongwei_6688
shell脚本
出现原因:windows上写的脚本,直接拷贝到linux系统上运行由于格式不兼容导致
解决办法:
1. 比如文件名为myshell.sh,vim myshell.sh
2. 执行vim中的命令 : set ff?查看文件格式,如果显示fileformat=dos,证明文件格式有问题
3. 执行vim中的命令 :set fileformat=unix 将文件格式改过来就可以了,然后:w
高一上学期难记忆单词
dcj3sjt126com
wordenglish
honest 诚实的;正直的
argue 争论
classical 古典的
hammer 锤子
share 分享;共有
sorrow 悲哀;悲痛
adventure 冒险
error 错误;差错
closet 壁橱;储藏室
pronounce 发音;宣告
repeat 重做;重复
majority 大多数;大半
native 本国的,本地的,本国
hibernate查询返回DTO对象,DTO封装了多个pojo对象的属性
frankco
POJOhibernate查询DTO
DTO-数据传输对象;pojo-最纯粹的java对象与数据库中的表一一对应。
简单讲:DTO起到业务数据的传递作用,pojo则与持久层数据库打交道。
有时候我们需要查询返回DTO对象,因为DTO
Partition List
hcx2013
partition
Given a linked list and a value x, partition it such that all nodes less than x come before nodes greater than or equal to x.
You should preserve the original relative order of th
Spring MVC测试框架详解——客户端测试
jinnianshilongnian
上一篇《Spring MVC测试框架详解——服务端测试》已经介绍了服务端测试,接下来再看看如果测试Rest客户端,对于客户端测试以前经常使用的方法是启动一个内嵌的jetty/tomcat容器,然后发送真实的请求到相应的控制器;这种方式的缺点就是速度慢;自Spring 3.2开始提供了对RestTemplate的模拟服务器测试方式,也就是说使用RestTemplate测试时无须启动服务器,而是模拟一
关于推荐个人观点
liyonghui160com
推荐系统关于推荐个人观点
回想起来,我也做推荐了3年多了,最近公司做了调整招聘了很多算法工程师,以为需要多么高大上的算法才能搭建起来的,从实践中走过来,我只想说【不是这样的】
第一次接触推荐系统是在四年前入职的时候,那时候,机器学习和大数据都是没有的概念,什么大数据处理开源软件根本不存在,我们用多台计算机web程序记录用户行为,用.net的w
不间断旋转的动画
pangyulei
动画
CABasicAnimation* rotationAnimation;
rotationAnimation = [CABasicAnimation animationWithKeyPath:@"transform.rotation.z"];
rotationAnimation.toValue = [NSNumber numberWithFloat: M
自定义annotation
sha1064616837
javaenumannotationreflect
对象有的属性在页面上可编辑,有的属性在页面只可读,以前都是我们在页面上写死的,时间一久有时候会混乱,此处通过自定义annotation在类属性中定义。越来越发现Java的Annotation真心很强大,可以帮我们省去很多代码,让代码看上去简洁。
下面这个例子 主要用到了
1.自定义annotation:@interface,以及几个配合着自定义注解使用的几个注解
2.简单的反射
3.枚举
Spring 源码
up2pu
spring
1.Spring源代码
https://github.com/SpringSource/spring-framework/branches/3.2.x
注:兼容svn检出
2.运行脚本
import-into-eclipse.bat
注:需要设置JAVA_HOME为jdk 1.7
build.gradle
compileJava {
sourceCompatibilit
利用word分词来计算文本相似度
yangshangchuan
wordword分词文本相似度余弦相似度简单共有词
word分词提供了多种文本相似度计算方式:
方式一:余弦相似度,通过计算两个向量的夹角余弦值来评估他们的相似度
实现类:org.apdplat.word.analysis.CosineTextSimilarity
用法如下:
String text1 = "我爱购物";
String text2 = "我爱读书";
String text3 =