写在前面【后补的小恳请】
20号左右有几位学术败类蹭着热点,用着别人辛辛苦苦得到的数据,辜负了共享者为控制疫情节省时间提高效率的初衷,大搞噱头,发国难论文。我虽然是女生,但“千古文人侠客梦”,没有学临床,没法上前线,只希望写这篇科普为大家分辨什么是国之柱石,哪些是国之禄蠹。所以原文难免言辞太激烈了,请谅解。
现在疫情越来越严重,好友告诉我,战时发泄情绪没有任何作用,我们应该做更有意义的事情。我查了很多自媒体和主流媒体,发现没有人更新本次武汉冠状病毒的学术信息,我和好友英语都很好,她在北美top master毕业,我们希望能实时更新国际最前沿的文献,给外行人提供一份安心,让大家知道所有人都在为打赢这场仗而努力。如果能给相关科研工作者一点灵感,就更不胜荣幸了。
文献更新地址为https://www.jianshu.com/p/d0f770c8f055恳请有余力的同学加入我们!!
以下是原博文。
既然有教授和研究员蹭这个热点发学术垃圾,填经费的坑。那本美少女一不卖高价口罩,二不传播恐慌情绪。那我蹭流量做个基础科普增加一下博客浏览量不算发国难财吧,谁要骂我我就骂谁。
好了言归正传,本博文aimed手把手教你如何利用开源数据和软件搞出看似很高端很有用·其实是个生物本科生或者英文很好的高中理科生都能搞定·毛用没有·只要蹭对热点分分钟开绿色通道特审并且公众号1m+阅读量的学术论文。顺带科普一下火速搞出基因组的一线实验人员的辛苦。
本科修过生物信息学,结课论文就是找任意病毒做你所能做的所有生信分析,我当年做的就是SARS。可惜旧笔记本不在家,硕士方向是群体没搞这个,可能有遗忘遗漏,先将就看吧。
概述
武汉新型冠状病毒,世卫组织命名为2019-nCoV(coronavirus[冠状病毒]),从这个命名就可以看出来是2019年发现的。
病毒的物种名是Wuhan seafood market pneumonia virus (viruses)武汉海鲜市场肺炎病毒。
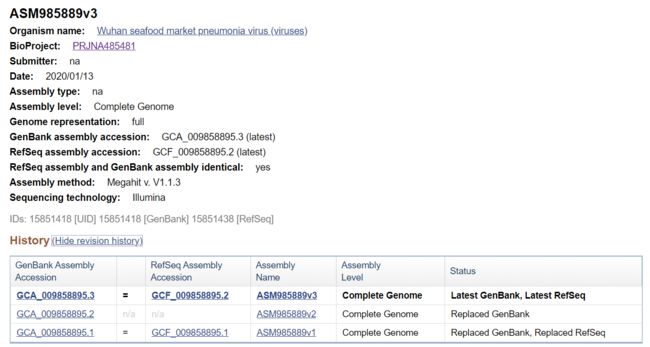
2020年01月13日国内研究单位上传测序文件,组装出该病毒的完整参考基因组,最新版共长29,903bp(就是一共这么多ATCG碱基的意思),包含10个基因。
本文分析数据均来自上传到NCBI美国国家生物信息中心的开源数据,软件也都为开源软件,带pmid。每一个步骤均有详细说明、截图、链接,致力于让高中理科生都看得懂学得会。
参考基因组数据的获取
以本次的武汉新型冠状病毒为例,介绍如何利用NGS来测序组装获得新发RNA病毒的全基因组序列。
采集感染标本,分离血清,这部分工作应该是首发病例的医院完成的。确诊后1d内能完成。
培养病毒,提取RNA,我猜应该是由疾控中心完成的吧。RNA不稳定,极易降解,这部分工作辛苦又危险,培养需要7d左右。
建库测序,公布数据显示测序仪器生产商是illumina,我猜型号是Miseq PE300。24h内肯定可以出数据。RNA病毒测序原理是类似的,可参考
俞维维, 孙逸, 颜浩, et al. 基于高通量技术的SFTS病毒基因组测序方法的建立[J]. 中华实验和临床病毒学杂志, 2019, 33(1):89-94.https://new.qq.com/omn/20190412/20190412A0MC5Q.html- 得到fastq数据后就可以进行基因组组装。NCBI组装策略显示NA,我猜是简单拼接,不需要denovo。基因组组装出来后修正过三次,现在是v3,组装report全文如下:
来自 https://www.ncbi.nlm.nih.gov/assembly/GCF_009858895.2#/def
和我师兄讨论他说没有采取三代策略是“因为这个序列涉及到后续抗原的研发什么的,序列的准确性有要求”,毕竟三代的错误率有目共睹。
病毒的基因组都很小,所以组装比较简单,加上现在有pacbio,Oxfordnanapore这些读长很变态的小测序仪,生信上来说组装工作没有太大难度,但是这是实实在在的工作,前期实验部分的分离培养提RNA这些又琐碎又危险的工作是防疫战争一切后续的基础,辛苦病毒所的研究人员。很讽刺的是一线工作人员的数据却成了学术垃圾的基石,实名diss发在science China的那篇,发成科普无可厚非,可作为学术论文,请问创新点在哪,解决的问题是什么?
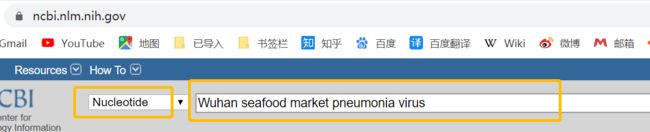
综上,研究人员组装参考基因组数据后会上传到开源数据库NCBI,我们所有人可以登录首页https://www.ncbi.nlm.nih.gov/,选择Nucleotide库,搜索物种Wuhan seafood market pneumonia virus,即可看到新型病毒的基因组信息,链接https://www.ncbi.nlm.nih.gov/nuccore/1798174254,
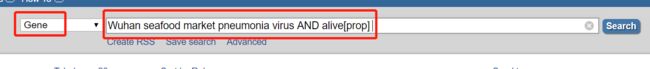
选择Gene库,即可查看10个基因的信息,分别是ORF10,orf1ab,N(核衣壳磷蛋白),ORF8,ORF7a,ORF6,M(膜糖蛋白),E(包膜蛋白),ORF3a,S(表面糖蛋白)
ps:额外科普知识
(1)病毒结构,病毒可以从基因组看出来它肯定很小,结构也非常简单,就是蛋白外壳和内部核酸,拜托大家回忆一下高中生物讲的肺炎双球菌感染小鼠的试验,那个噬菌体长什么样,有些类似。下图是SARS的,文献https://science.sciencemag.org/content/300/5626/1763
:
(2)基因命名,ORF是Open Reading Fram,开放阅读框,即编码蛋白的序列,N、M、E、S都是编码蛋白外壳的,SARS也拥有,这是冠状病毒的共同结构,比较保守。如果要研究,肯定是比对剩下的
链接https://www.ncbi.nlm.nih.gov/gene/?term=Wuhan%20seafood%20market%20pneumonia%20virus%20AND%20alive%5Bprop%5D&utm_source=gquery&utm_medium=search。
与SARS比对
参考基因组
SARS :The genome of SARS-CoV is 29,727 nucleotides in length and has 11 open reading frames, and its genome organization is similar to that of other coronaviruses. Phylogenetic analyses and sequence comparisons showed that SARS-CoV is not closelyrelated to anyof the previously characterized coronaviruses.
SARS有 29,727bp的参考基因组,11个开放阅读框,与其余冠状病毒类似,进化树和比对均显示与当时典型的冠状病毒并不类似。
2019-ncov有29,903bp的参考基因组,6个开放阅读框。
蛋白
新病毒与SARS比对
- 在参考基因组页面右边可以看到RUN BLAST的键,如图
没找到可以直接进入https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE=Nucleotides&PROGRAM=blastn&QUERY=NC_045512.2&DATABASE=nr&MEGABLAST=on&BLAST_PROGRAMS=megaBlast&LINK_LOC=nuccore&PAGE_TYPE=BlastSearch
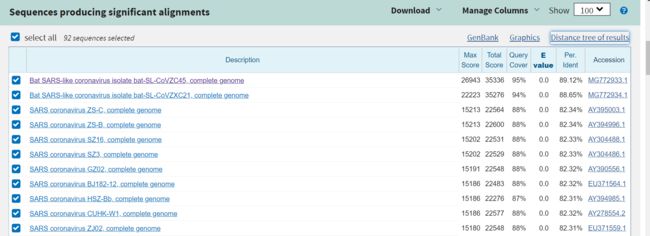
序列框内需要输入新病毒的参考基因组编号(参考基因组页面标题下的小字NCBI Reference Sequence: NC_045512.2),复制NC_045512.2,选择standard database,按最底部的BLAST,即可。结果如图:
可以看到亲缘关系最近的依旧是SARS,有两个原因,主要是SARS数据多,
取了81.64%相似度以上的用blast tree viewer在线做系统进化树,如图线状的,简单易懂,离得越近亲缘关系越近,但是亲缘关系近并不能说明致病性相似,毕竟人和大猩猩的亲缘关系比SARS和新病毒的89%高多了:
环状的:
蛋白预测(待补充)
新病毒
一维
二维
三维
结构域
SARS
好累,吃完早饭再搞,NCBI上不去了。