Pre:
这是一篇science的文献report,帮助梳理文献思路帮助非常大!(自己只理解了部分,后续有新的补充会及时更新!)
Background (背景)
1: DNA甲基化是表观修饰的一种,在动植物中可调控基因的转录,胚胎发育和细胞分化。在哺乳动物中,DNA甲基化一般发生在对称的CpG二核苷酸上。这也意味着,如果一个CpG岛上的C(胞嘧啶)发生了甲基化,那么互补链上的C也会有响应的变化。
2:这种模式,在DNA复制的时候会被打破,当新生成的未甲基化的子链和甲基化的父链会形成一种不对称的甲基化CpG二联体(称为半甲基化DNA)。
在伴随着不断的复制过程中,半甲基化DNA的命运通常被认为会变成全甲基化或者是未甲基化。(这是一个“稀释”的过程)
3:然而,胚胎干细胞(ESC) (1) 和滋养层干细胞(2)中约10%的CpG保持半甲基化。
4:在复制叉上,半甲基化的DNA被读取蛋白UHRF1(未泛素化PHD和RING指结构域1)结合,然后募集DNMT1 [5-甲基胞嘧啶转移酶1],恢复原始的对称甲基化模式。(图2左边 maintenance)
5:一般来说,DNMT1可维持DNA甲基化的状态,它偏向于半甲基化的DNA,然后把它变成甲基化的DNA。而DNMT3A和DNMT3B倾向于CpG从头开始甲基化.
6:之前有报道称,DNMT3A对裸露的DNA显示出高的从头甲基化的活性,但对包裹核小体的DNA没有显示出较高的活性。(复制后DNA被包装在核小体中,这可以抑制特定CpG处的DNMT3A活性,确保它们保持半甲基化。)
7:DNMT1加载到DNA复制叉取决于UHRF1(4),组蛋白H3赖氨酸36甲基化(H3K36me)可能在DNMT3B募集到目标位点中起作用。
8:此外,在表达MECP2功能缺失突变体的细胞中,半甲基化区的染色质相互作用显著减少。因为MBD蛋白最初是通过其对对称甲基化DNA的纯化出来的。 (11).
Question(科学问题)
1:并不知道DNA半甲基化的维持是偶然发生的还是被"设计"好的
2:并且为了研究DNMT与其体内底物CpG之间的联系.
3:尽管DNMT3A和DNMT3B有相似的结构域和活性,但是DNMT3A而非DNMT3B如何促进基因特异性半甲基化?
4:DNMT3A是如何识别未甲基化的DNA的呢?是通过特殊的转录因子还是通过组蛋白修饰还是别的DNA结合蛋白呢?DNMT3A靶向基因组的哪些部分用于半甲基化?
5:半甲基化DNA如何调节染色质相互作用?
Method(方法)
Xu和Corces在人ESC中标记了新生的复制DNA或成熟的复制后DNA,并绘制了每个DNMT结合的序列,以揭示每个DNMT结合的靶基因组位点的DNA甲基化状态。
Step by Step
一:获得DNA甲基化的动态信息(未甲基化---->半甲基化----->甲基化)采取的策略
1.1 我们来思考一个问题,在DNA复制的过程中,伴随着子链的生成,我们如何把父链和子链分开?
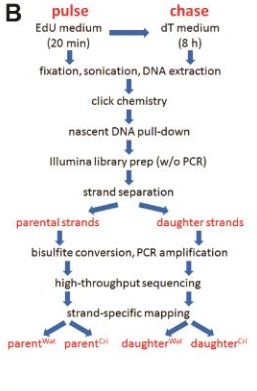
作者开发出来一种nasBS-seq去考量全基因组新生染色质的链特异性的甲基化情况。
关于nasBS-Seq实验设计:
不太长版
[ PS:在20Min内先进行EdU的标记,这个时候标记的链被称为pulse链(脉冲链,先导链) 然后用dT进行正常的标记,培养8h这个时候生成的链被称为chase链(后随链)]
分解版:
(PS:什么是EdU? 5-乙炔基-2'-脱氧尿苷(EdU)是胸苷类似物,其掺入分裂细胞的DNA中。EdU用于测定细胞培养物中的DNA合成,并检测在胚胎,新生儿和成年动物中经历DNA合成的细胞.并且有利于利用click chemistry 形成共价键,加上biotein标签。)
(PS:亲代是两条黑色链,新和成的是一条黑的一条红色的DNA链,但是DNA链上带上了EdU的标签。)
(PS:关于 click chemistry简单介绍在其上加上叠氮基团,然后通过简单的化学反应可以加上biotein标签)
(PS:大笑脸是带有抗体的beads,进行末段修复,然后加A,加上Adapter.)
(PS:在这一步中,父链和子链是可以分开的,子链是带着Biotein标签的)
(PS:然后进行扩增测序)
首先选取了H9-hESCs细胞,用EdU标记了20分钟,然后用正常的dT进行了标记(为了看后续的DNA甲基化的情况),然后分别进行了建库。然后得到4条链的测序情况及转化率。
为了验证生物学重复之间的相关性如何,做了一个皮尔森相关系数的检验。
比较了不同生物重复之间C的频率(选取的是至少map5次以上的进行比较)在这个图中,横坐标是rep3和rep4中的甲基化频率,纵坐标是其他重复中甲基化的频率。在父链和子链中的比较。证明了相关性很好,重复可靠。
1.2:我们来看第二个问题:如何证明在DNA复制的过程中,DNA甲基化的遗传稳定性呢?
(我们得从两个角度上考量,第一个是在nasBS-Seq这个技术中,利用了EdU进行了标记,这一部分生成的链被称为pulse链,那么在这20分钟内新生成的DNA遗传甲基化的情况是什么样的呢?如何去比较?
第二个,在用后续的dT进行培养的8h里面。DNA甲基化遗传的情况怎么样呢?和前面20min的DNA遗传性比较呢?
首先他们先看看在CpG位点上的DNA甲基化的相关性,然后看看在先导链上CpG位点的DNA甲基化情况如何,然后比较了先导链和后随链上的DNA甲基化的相关性。证明了相关性都非常的高。证明了DNA甲基化的稳定性很好。)
此外,他们除了观察CpG,还观察了CHH CHG的情况(尽管不是和CpG一样对称),也观察到了保持了良好的遗传稳定性!
这些结果表明绝大部分DNA甲基化组在复制叉通过后20分钟内维持比较好。
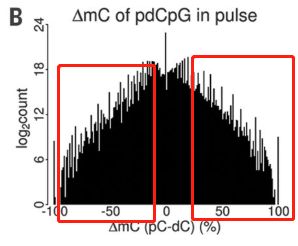
1.3:除了pc和dc之间的高度相关性,这两个C发现了频率还是很不一样的。如何去看待这个不同的甲基化频率呢?
首先看了一下pc和dc的差值,如果pc和dc的甲基化状态的是一样的,他们的值是0,如果他们的甲基化状态不一样的话,ΔmC = m(pC − dC)就会有差别。说明存在了半甲基化的频率。
我们可以看出,无论是在pulse和chase中它都存在半甲基化的情况。
使用75%的cutoff 来确定5-甲基胞嘧啶(mC)[ΔmC≤-75%,或≥75%,ΔmC= m(pC-dC)]的差异,我们获得了23,305个CpGlist,其中至少有一个二元体显示出半甲基化 .其中绝大多数(96%)仅在一个二元体内被半甲基化,并且在另一个条件下未能再现甲基化模式.。表明它们可能代表多能细胞中丰富的快速DNA甲基化转换事件。
但是从pulse和chase的半甲基化情况来看,他们的遗传又是很稳定的(看起来是有矛盾?)
1.4:半甲基化是不是具有细胞特异性呢?
半甲基化在全能细胞中类型是特异性的,并且在相关细胞谱系中保守。
2:ISA能解决什么问题?
因为WGBS的分析存在缺陷,我们在统计CpG二聚体甲基化状态的时候,只能是单独计算每条链的CpG的甲基化状态,然后把两条链的结果重合起来看。CpG二聚体甲基化状态在DNA的复制过程很重要。在DNA复制传代的过程中,父链和子链的CpG二聚体的甲基化状态是不清楚的。为了获得CpG位点的每个C的状态。开发出来了一种iSA的方法。去统计CpG的甲基化情况。
2.1 想办法拼回CpG的信息
2.2在mapping的时候如何把一条一条的reads整合起来?
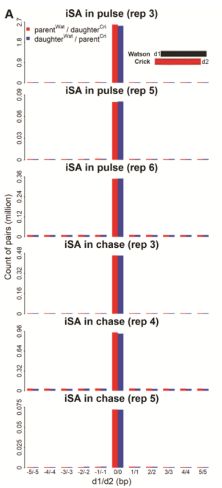
这里需要解决一个问题,如何解决random匹配的问题?
为了确定这种随机配对可能产生的干扰,我们通过计算两端碱基距离1-5bp的reads的富集情况。然后看看富集比例,是否有影响
fig2B并且可以通过iSA可以确定4中甲基化的状态:甲基化(intraCpGme),非甲基化(intraCpGunme)或pC-或dC-半甲基化(intraCpGhemi-pC或intraCpGhemi-dC)图2B是可以先导链和后随链的DNA甲基化的状态可以确定下来的。然后可以确定450万和210万个CpG的状态。
2.3接下来,我们使用iSA来解析小鼠早期胚胎期(15)中已发表的WGBS数据集,并显示hemiCpGs占DNA甲基化组的4%至18% 图2C
并且发现在内细胞团时期,半甲基化状态是和转录状态有关的。
3:DNA半甲基化和DNMT之间的关系
3.1如何探究DNMT和CpG甲基化的相互作用呢?
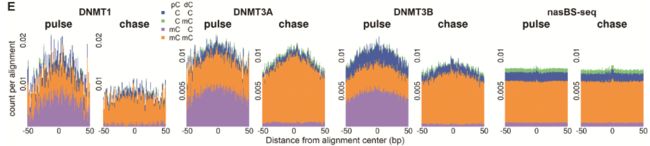
首先得把和DNMT互作的DNA给富集下来,然后再利用BS-seq进行分析,然看看甲基化的情况。这个就是nasChIP-BS-Seq的基本思想
3.2并且进一步的发现了,DNMT更加倾向于结合pulse链的侧翼区(图S6E)
侧翼区(flanking region)
根据维基定义:The 5' flanking region is a region of DNA that is adjacent to the 5' end of the gene. The 5' flanking region contains the promoter, and may contain enhancers or other protein binding sites. It is the region of DNA that is not transcribed into RNA.
以5'侧翼区为例,侧翼区是指与基因5’末端毗连的DNA区域,这些区域通常包含启动子、增强子或者其他蛋白质结合位点,这部分的区域不能被转录为RNA。
此外,在后随链中,DNMT1 DNMT3b的富集比例减少。提示DNMT可能对新生和成熟染色质有不同的偏好性
3.3当把3种DNMT敲除时,分别会有什么现象出现呢?
在DNMT1敲除的细胞系中,DNMTl-targeted intraCpG hemi pC 和intraCpGme (半甲基化状态和甲基化状态)都显示了甲基化下降的更快(比平均水平)说明DNA甲基化的维持主要是靠DNMT1来维持的。
当把DNMT3A/3B 单敲除或者双敲除的时候,可以看到半甲基化比甲基化的状态下降的还要明显!
并且他们还有甲基化分布的偏好性(他们和甲基化统计随机模拟出来的结果不一样)
3.4 nasChIP-BS seq和 nasBS-seq的比较
接下来我们想通过nasChIP-BS-seq通过检测半甲基化的状态,或者是去捕获CH的dC甲基化的状态。在这两种情况下,尚未甲基化的dCs在DNMT3A / 3B nasChIP-BS-seq中显示出广泛的低甲基化,但在不显示nasBS-seq中。这些结果表明,nasChIP-BS-seq可以在可视化从头甲基化中DNMT和底物dCs之间的瞬时相互作用。
4 半甲基化的生物学解读
4.1和CTCF的结合比率
在H1-hESC中不同基因组特征的不同类型的intraCpG的频率。令人惊讶的是,CTCF结合位点显示出非常高的intraCpG-hemi与intraCpGme的比率。(图S7A)
CTCF / cohesin结合位点协调整个哺乳动物基因组的三维染色质相互作用。然后我们开发了nasChIP-seq来绘制CTCF和SMC1A(一种cohesin亚基)在H9-hESC中的新生染色质上的结合情况。
甲基-CpG结合结构域(MBD)蛋白可以与mCpG和mCA结合(21,22),表明它们与mC的结合对另一条链的甲基化状态没有选择性。 为了研究它们与hemiCpGs的关联,我们分析了mESC中公布的WGBS和MBD ChIP-seq。
为了克服ChIP-seq的不足分辨率,我们分析了CTCF基序上MBD蛋白的占据,显示仅上游或仅下游的遗传性半甲基化。 实际上,MeCP2,Mbd1a,Mbd1b,Mbd2a和Mbd2t都显示出定向特异性共定位与半甲基化。(图S9)表明在没有DNA甲基化的情况下,MeCP2的结合从hemiCpG依赖性模式转变为甲基化非依赖性模式。
并且发现了,在hESC中,单独的DNMT3B-KO足以消除CTCF基序上的大多数遗传性半甲基化,而对周围DNA甲基化的影响最小(图S10C)
Results(结果)
1:DNMT1结合的新生DNA片段主要是半甲基化的。 在成熟DNA中,相同的基因座是对称甲基化的,显示出DNMT1的有效维持甲基化
2:他们(作者)发现半甲基化位点是在几个细胞分裂中遗传的。这个发现挑战了之前的半甲基化是不稳定的的说法并且可以成为一个稳定遗传的表观现象。
3:然而,他们还鉴定了一小部分稳定半甲基化的位点,并在CTCF(CCCTC结合因子)/ cohesin结合位点显示其遗传。 CTCF和cohesin需要这些遗传的半甲基化位点以建立适当的染色质相互作
4:DNMT3B结合位点也富集半甲基化DNA,代表DNMT3B可介导的从头甲基化。 这些位点在成熟DNA中变得对称甲基化,表明DNMT3B与染色质结合的时间足够长以进行对称甲基化。
5:与DNMT3B类似,DNMT3A结合富含半甲基化CpG的新生DNA。 然而,这些DNMT3A结合位点中的一些甚至在后复制DNA中仍然是半甲基化的.
6:在连续的细胞分裂过程中监测DNA甲基化水平,确定这些位点的持续半甲基化。这表明,dnmt3a依赖的机制可能调控位点特异性半甲基化,可以作为一种稳定的表观遗传修饰遗传。
7:由转录调节因子CCCTC结合因子(CTCF)和cohesin(其参与将复制的染色体保持在一起)结合的基因组区域富含半甲基化。CTCF和cohesin都是长染色质相互作用所必需的,并且可能影响基因表达。 作者发现在没有半甲基化的情况下,这种染色质相互作用被破坏。
8:他们利用MBD蛋白的全基因组结合数据,并通过进行计算分析,发现这些蛋白质以相同的方式与半甲基化DNA结合。
hypothesis(假设)
作者提出MBD(甲基-CpG结合结构域)蛋白,如MECP2(甲基-CpG结合蛋白2),可以在这个过程中发挥作用。
remaing questions(遗留问题)
因此需要进一步的研究来阐明MBD蛋白如何在体内结合半甲基化的CpG。
哺乳动物胚胎早期发育过程中基因组的半甲基化会动态变化。这些标记是否 1)通过调节关键基因的表达,2)通过CTCF-黏连蛋白 3) MBD蛋白介导的长距离染色质相互作用来调节哺乳动物胚胎发生?
Ref:
1:https://science.sciencemag.org/content/359/6380/1166.long
2:chrome-extension://cdonnmffkdaoajfknoeeecmchibpmkmg/static/pdf/web/viewer.html?file=https%3A%2F%2Fscience.sciencemag.org%2Fcontent%2Fsci%2Fsuppl%2F2018%2F03%2F07%2F359.6380.1166.DC1%2Faan5480_Xu_SM.pdf
3:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6359960/#R18