PCA、PCoA、NMDS、RDA和CCA比较
本文转自:https://interact.i-sanger.com/article/view/talk_id/45.html
转载地址:https://mp.weixin.qq.com/s/UqD4m9QTN50IGpXwBiyFLA
排序分析的异同点?
PCA、PCoA和NMDS分析属于非约束性排序分析,而RDA/CCA和db-RDA分析属于约束性排序分析,即分别是在环境因子的约束条件下进行的PCA和PCoA分析。因此,一般主要利用PCA、PCoA或NMDS分析进行样本比较,反映样本间菌群结构的相似性和差异性,从而分析组间样本能否明显区分开;而RDA/CCA和db-RDA分析则多用来阐述环境因子对样本菌群结构变化的影响,不仅可以反映样本、物种和环境因子之间的相关性,而且可以找出对物种分布变化影响程度较大的环境因子。
小编还贴心的为您总结了这“5胞胎”的异同点:
图2
距离算法为何会影响排序结果?
很多老师在对数据进行分析时会惊奇的发现,PCoA和db-RDA分析选择不同的距离算法会产生不同或类似的结果,WHY?
我们以PCoA分析为例:
图3
图注:4种距离的PCoA分析。不同颜色代表不同的样本组别。
同一数据,小编分别选择了euclidean,weighted_unifrac,binary_euclidean和unweighted_unifrac这4种距离进行分析,结果发现后2种距离可以将3组样本明显区分开来,且组内差异较小;反之,前2种距离区分效果不佳。
究其原因,在于前2种距离只考虑了样本中物种存在与否,而后2种距离除此之外还考虑了物种在各个样本中的丰度分布。因此在只考虑样本中物种是否存在时能够将不同的样本组区分且组内差异较小。那么后续针对这种数据类型是选择只考虑物种有无的距离算法将获得更佳的聚类效果,反之亦然。
PCA添加环境因子与RDA有何区别?
美吉i-sanger云平台用户可能都知道在平台上PCA分析也是可以添加环境因子的。那我做个带环境因子的PCA不就O啦,为啥还要进行RDA分析呢?
如下图所示:
图4
图注:不同颜色代表不同的样本组别
使用同一数据分别进行PCA和RDA分析。小美无论选择多少环境因子对PCA排序结果是没有任何影响的;而RDA分析中选择不同的环境因子可得到不同的排序结果。
出现这种情况的结果的原因是PCA分析作为非约束性排序,环境因子加入并不会影响样本间菌群差异获得的排序结果;而RDA分析是在所选择的环境因子的约束条件下进行的PCA分析,因此选择不同的环境因子组合会产生的不同的排序分析结果。
高能预警!!!排序分析文献大放送啦!让我们看看科研人员们都是如何选择排序分析的......
1. PCA分析
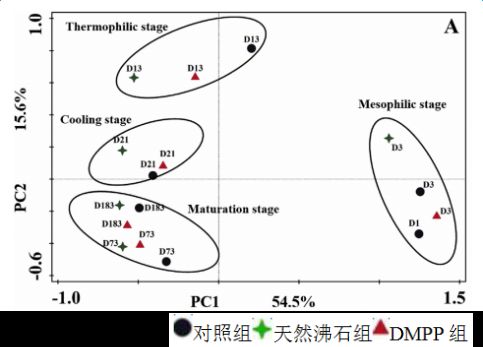
Title: Impacts of addition of natural zeolite or a nitrification inhibitor on antibiotic resistance genes during sludge composting
DOI.org/10.1016/j.watres.2016.01.010
图5
分析解读:PCA分析发现,在污泥堆肥过程中(图中不同的圆圈代表不同的污泥堆肥过程)的不同阶段,微生物的群落结构发生了较大的变化,天然沸石和硝化抑制剂DMPP的加入不会改变微生物群落的演变速率。
2. PCoA分析
Title: Maturation of the infant microbiome community structure and function across multiple body sites and in relation to mode of delivery
DOI:10.1038/nm.4272
图6
分析解读:PCoA分析表明,分娩时新生儿身体各部位菌群结构组成差异较小;而出生6周后的婴儿身体各部位菌群结构组成差异显著。
3. NMDS分析
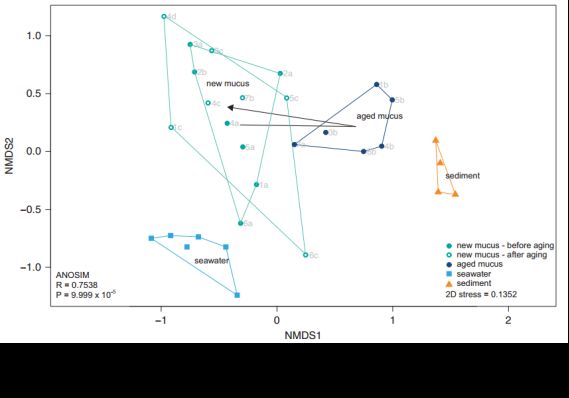
Title: The microbiome of coral surface mucus has a key role in mediating holobiont health and survival upon disturbance
DOI:10.1038/ismej.2016.9
图7
分析解读:NMDS分析发现,珊瑚表面粘液与海水和沉积物样本可以明显区分开;新粘液与老化粘液菌群具有一定差异;当老化粘液层释放后,其菌群恢复至新粘液的初始状态。此外,该图右下角展示了NMDS分析的stress值。
友情提示:近一年来,我们可能会发现文献中使用NMDS分析时大多会在图中添加这个神秘的stress值,那么它到底有何含义呢?Strees值其实反映了NMDS分析结果的优劣。通常认为stress<0.2时,使用NMDS分析的结果具有一定的解释意义;当stress<0.1时,可认为是一个好的排序结果;当stress<0.05时,则表明分析结果具有极好的代表性。
4. RDA/CCA分析
Title: Comparison of community structures of Candidatus Methylomirabilis oxyfera-like bacteria of NC10 phylum in different freshwater habitats
DOI:10.1038/srep25647
图8
分析解读:CCA分析表明,沉积物/土壤中氨氮(NH4+-N)和总无机氮(TIN)含量是与检测样本中NC10细菌集合体变化相关的2个最重要环境因素。
5. db-RDA分析
Title: Weight gain in anorexia nervosa does not ameliorate the faecal microbiota, branched chain fatty acid profles, and gastrointestinal complaints
DOI:10.1038/srep26752
图9
分析解读:db-RDA分析表明,疾病状态和年龄与菌群结构显示出相关性,其中疾病状态与菌群结构显著相关(p=0.011,图A);仅分析AN患者发现AN亚型对微生物菌群结构具有显著影响(p=0.008,图B)。
友情提示:一般运用RDA/CCA或db-RDA分析的文献中还会以文字描述的形式提供某个环境因子对物种分布影响程度的大小(r2值)和显著性P值。如何利用i-sanger云平台项目文件确定某一环境因子与菌群结构之间的相关性呢?小美给您解答:
路径1:在RDA/CCA分析中的RDA/CCA相关数据表页面中查找envfit环境因子表:
图10
路径2:1)在项目文件中找到RDA分析文件夹中的rda_envfit表格文件;
图11
2)下载表格文件并使用EXCEL打开:
图12
表格解读:
1.RDA1和RDA2列:表示环境因子与排序轴之间的相关性;
2.ef.vectors.r(即r2值):表示环境因子与物种分布的决定系数,该值越小则该环境因子对物种分布的影响越小;
3. Ef.vectors.pvals(即p值):相关性的显著性检验值。