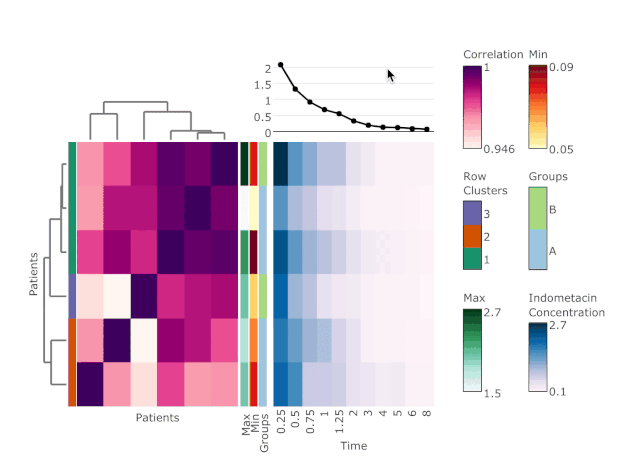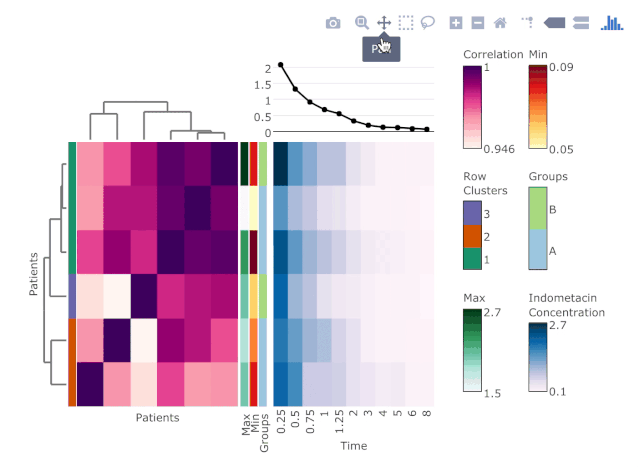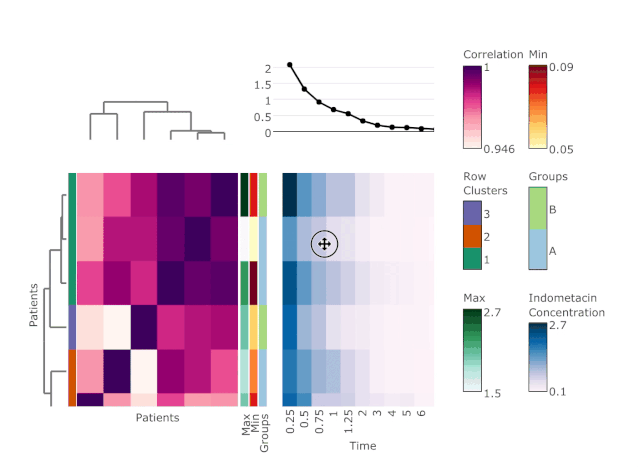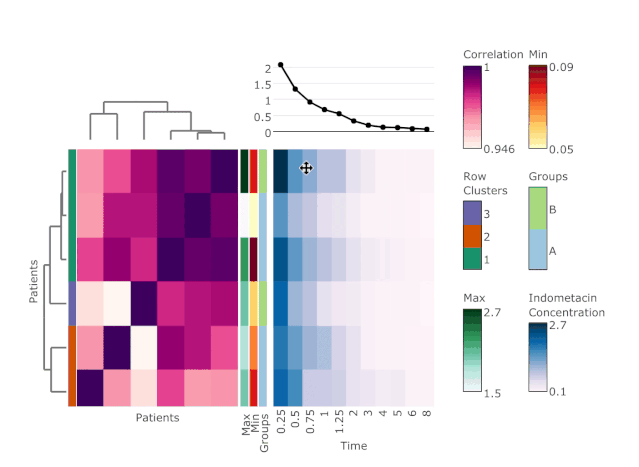
iheatmapr包:可交互的热图绘制方法
iheatmapr包
在R中有许多用于绘制热图的包,今天介绍一个比较有意思的用于绘制热图的包,iheatmapr 包,这个包是用来绘制复杂的、且具有交互作用的热图。
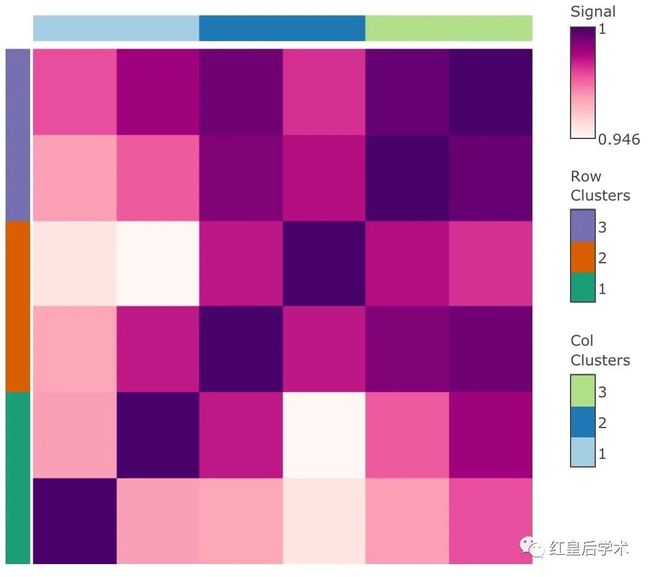
用iheatmapr包绘制得到的热图就是下方这样,不仅能够反应多种信息,还具有交互功能。
这个热图中包含8个信息:
样本的相关性矩阵热图;
按列的聚类关系;
按行的聚类关系;
样本聚类模式的颜色注释;
每个样本最低和最高浓度的颜色注释;
样本自身分组的颜色注释;
每个样本真实药物处理实际过程的热图;
平均药物浓度随着时间变化的散点图。
iheatmapr包的安装
“iheatmapr”包含在CRAN中,可以直接使用install.packages()函数进行安装。
install.packages("iheatmapr")
我的R是4.0,在安装的时候提示4.0中缺少一个依赖包S4Vectors。
查了一下,S4Vectors是属于Bioconductor的包,因此用Bioconductor包的安装方式安装S4Vectors。
if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager")
BiocManager::install("S4Vectors")
在安的过程中发现速度那叫一个龟速,因此设置了一下Bioconductor的镜像。
chooseBioCmirror()
之后在对话框中输入离自己最近的镜像编号就行了。
安装完S4Vectors之后,iheatmapr就可以正常加载了。
library(iheatmapr)
使用实例
首先要构造用于分析的绘图数据,这里使用datasets包内的Indometh作为示例。
#载入绘图包library(iheatmapr)library(datasets)library(reshape2)#使用acast调用Indometh数据集中的内容Indometh_matrix <- acast(Indometh, Subject ~ time, value.var = "conc")Indometh_matrix <- Indometh_matrix[as.character(1:6),]rownames(Indometh_matrix) <- paste("Patient",rownames(Indometh_matrix))##计算相关性矩阵Indometh_patient_cor <- cor(t(Indometh_matrix))##取每个样本数据中的最大值和最小值patient_max_conc <- apply(Indometh_matrix,1,max)patient_min_conc <- apply(Indometh_matrix,1,min)##给每个样本随机分配一个分组patient_groups <- c("A","A","B","A","B","A")
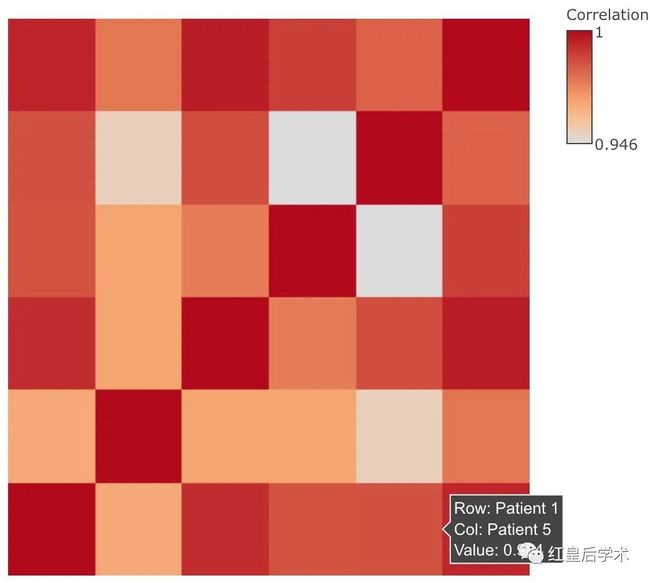
绘制相关性矩阵热图
example_heatmap <- main_heatmap(Indometh_patient_cor, name = "Correlation")example_heatmap
可以使用colors参数修改颜色,colors = “RColorBrewer palettle/颜色名字”。
main_heatmap(Indometh_patient_cor, colors = "Pinks", name = "Correlation")
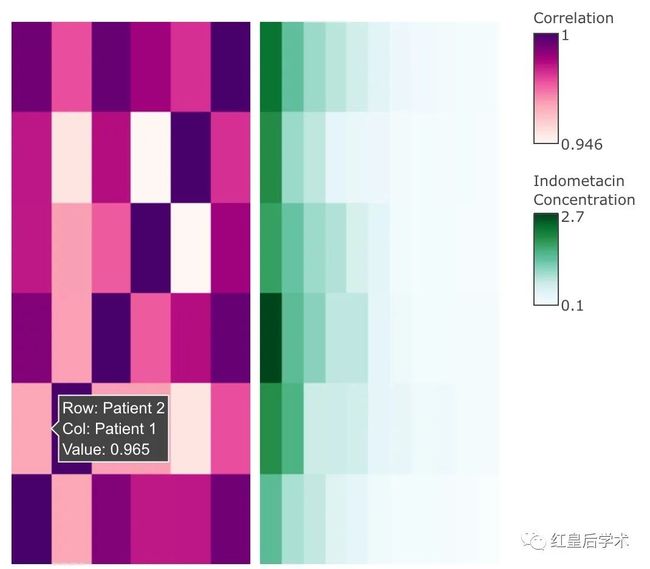
添加第二幅热图
main_heatmap(Indometh_patient_cor, name = "Correlation") %>% add_main_heatmap(Indometh_matrix, name = "Indometacin
Concentration"
如果name=相同名字的话,新添加的热图会与之前的热图共享相同的颜色。
如果你想改变添加热图的位置,就使用side=c(“left”, “right”, “top”,”bottom”)。
添加标签
main_heatmap(Indometh_matrix, name="Correlation") %>% add_row_labels() %>% add_col_labels() %>% add_row_title("Patients", buffer=0.2) %>% add_col_title("Patients", buffer=0.1)
buffer规定title文字与图之间的距离。
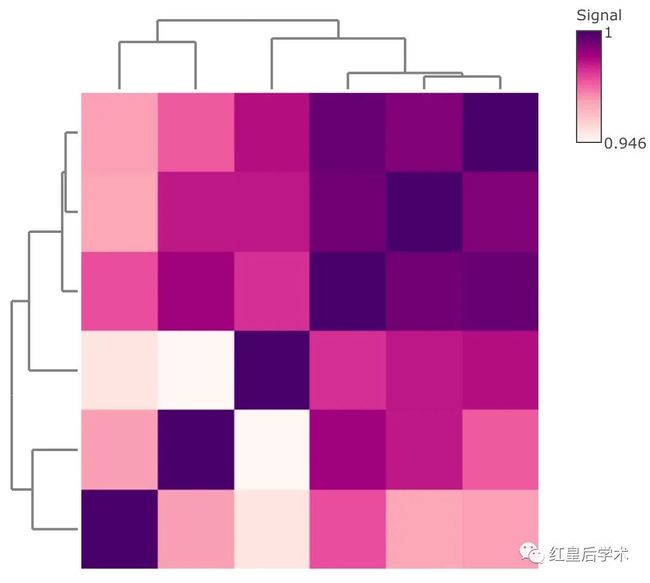
添加聚类关系
main_heatmap(Indometh_patient_cor) %>% add_row_clustering() %>% add_col_clustering()
如果是希望以颜色表示样本的聚类关系,则需要先进行一个聚类,之后在手动进行负值。
clust_assign <- kmeans(Indometh_matrix, 3)$cluster
main_heatmap(Indometh_patient_cor) %>% add_row_clusters(clust_assign) %>% add_col_clusters(clust_assign)
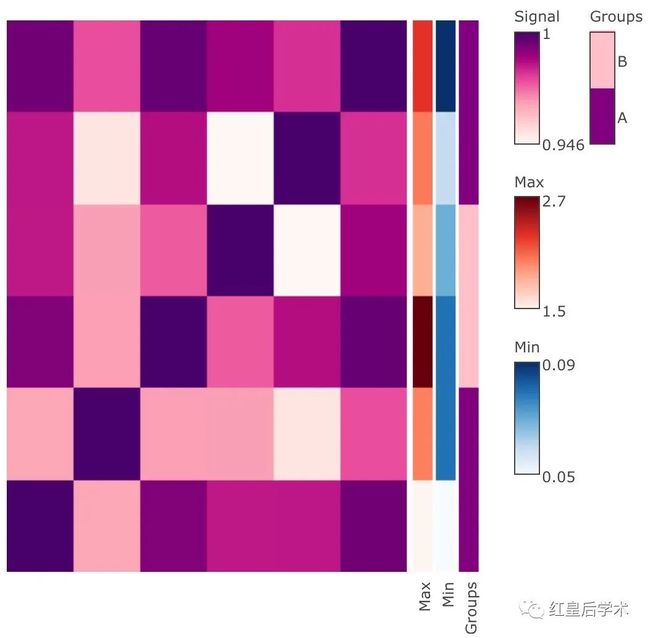
添加样本注释
main_heatmap(Indometh_patient_cor) %>% add_row_annotation(data.frame("Max" = patient_max_conc, "Min" = patient_min_conc, "Groups" = c("A","A","B","B","A","A")), colors = list("Max" = "Reds", "Min" = "Blues", "Groups" = c("purple","pink")))
除了add_row_annotation,还可以使用add_row_signal和add_row_groups添加注释。
main_heatmap(Indometh_patient_cor) %>%add_row_signal(patient_max_conc, "Max
Concentration", title = "Max", colors = "Reds") %>%add_row_signal(patient_min_conc, "Min
Concentration", title = "Min", colors = "Reds") %>%add_row_groups(c("A","A","B","B","A","A"), "Groups")
写在后面
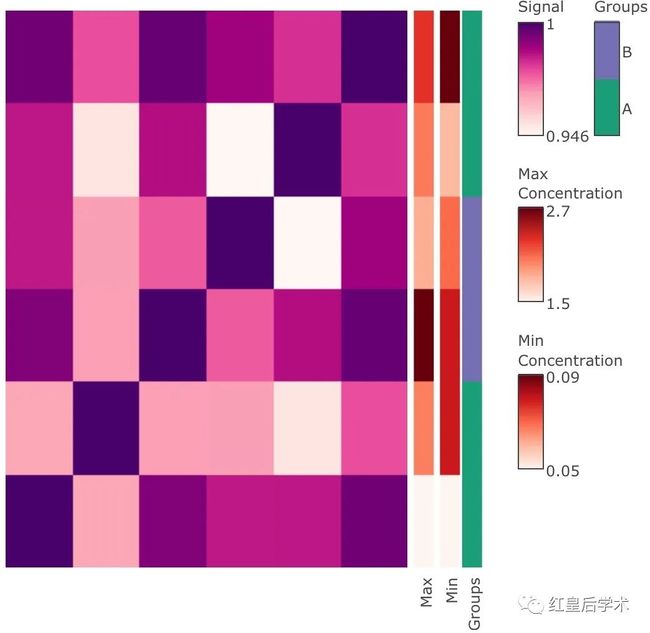
到这里,我们已经分布讲解了文章开始出现的复杂热图中的大部分实现代码,下面给出一个完整的代码。
main_heatmap(Indometh_patient_cor,name = "Correlation") %>% add_col_clustering() %>% add_row_clustering(k = 3) %>% add_row_title("Patients") %>% add_col_title("Patients") %>% add_row_annotation(data.frame("Max" = patient_max_conc, "Min" = patient_min_conc, "Groups" = patient_groups)) %>% add_main_heatmap(Indometh_matrix, name = "Indometacin
Concentration") %>% add_col_labels() %>% add_col_title("Time") %>% add_col_summary()
更对关于iheatmapr包的参数使用和个性化设置,请参考该包的帮助文档。
https://docs.ropensci.org/iheatmapr/articles/full_vignettes/iheatmapr.html#modular-building-blocks-1
你可能还想看
R语言 - 热图美化
R中1010个热图绘制方法
这也太简单了吧!一个函数完成数据相关性热图计算和展示
利用ComplexHeatmap绘制热图(一)
network3D: 交互式桑基图
network3D: 交互式网络生成
一个震撼的交互型3D可视化R包 - 可直接转ggplot2图为3D
ggThemeAssist|鼠标调整主题,并返回代码
往期精品(点击图片直达文字对应教程)
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集
![]()
![]()
![]()