K-均值聚类(Python3)学习笔记
K-均值聚类(Python3)
1. K均值算法
-
K-均值是发现给定数据集的 k k k个簇的算法。簇个数 k k k是由用户给定的,每个簇通过其质心(centroid),即簇中所有点的中心来描述。
-
给定样本集 D = { x 1 , x 2 , ⋯ , x m } D=\{\boldsymbol{x}_1,\boldsymbol{x}_2,\cdots,\boldsymbol{x}_m\} D={x1,x2,⋯,xm},“ k k k均值”( k k k-means)算法所得簇划分 C = { C 1 , C 2 , ⋯ , C k } C=\{C_1,C_2,\cdots,C_k\} C={C1,C2,⋯,Ck}最小化平方误差 E = ∑ i = 1 k ∑ x ∈ C i ∥ x − μ i ∥ 2 2 E=\sum_{i=1}^{k}\sum_{\boldsymbol{x} \in C_i}^{}{\lVert \boldsymbol{x}-\boldsymbol{\mu}_i \rVert^2_2} E=i=1∑kx∈Ci∑∥x−μi∥22
其中 μ i = 1 ∣ C i ∣ Σ x ∈ C i x \boldsymbol{\mu}_i=\frac {1}{|C_i|}\Sigma_{\boldsymbol{x} \in C_i}\boldsymbol{x} μi=∣Ci∣1Σx∈Cix是簇 C i C_i Ci的均值向量。直观来看,上式在一定程度上刻画了簇内样本围绕簇均值向量的紧密程度, E E E值越小则簇内样本相似度越高。 -
工作流程:
创建k个点作为起始质心(经常是随机选择)
当任意一个点的簇分配结果发生改变时
对数据集中的每个数据点
对每个质心
计算质心与数据点的距离
将数据点分配到距其最近的簇
对每一个簇,计算簇中所有点的均值并将均值作为质心 -
优点:容易实现。
-
缺点:可能收敛到局部最小值,在大规模数据集上收敛较慢。
-
适用数据类型:数值型数据。
1.1 K-均值聚类支持函数Python3实现
from numpy import *
import matplotlib
import matplotlib.pyplot as plt
#导入数据集
def loadDataSet(filename):
dataMat = []
fr = open(filename)
for line in fr.readlines():
curLine = line.strip().split('\t')
fltLine = list(map(float, curLine))
dataMat.append(fltLine)
return dataMat
#计算两个向量的欧式距离
def distEclud(vecA, vecB):
return sqrt(sum(power(vecA - vecB, 2)))
#生成随机质心
def randCent(dataSet, k):
n = shape(dataSet)[1]
centroids = mat(zeros((k, n)))
for j in range(n):
minJ = min(dataSet[:, j])
rangeJ = float(max(dataSet[:, j]) - minJ)
centroids[:, j] = minJ + rangeJ * random.rand(k,1)
return centroids
#K-均值聚类算法
def kMeans(dataSet, k, distMeas=distEclud, createCent=randCent):
m = shape(dataSet)[0]
clusterAssment = mat(zeros((m, 2)))#簇分配结果矩阵,存储簇索引值与误差
centroids = randCent(dataSet, k)
clusterChanged = True
while clusterChanged:#迭代标志
clusterChanged = False
for i in range(m):
minDist = inf
minIndex = -1
for j in range(k):#寻找最近质心
distJI = distMeas(centroids[j,:],dataSet[i,:])
if distJI < minDist:
minDist = distJI
minIndex = j
if clusterAssment[i,0] != minIndex:#如果簇分配结果发生变化,更新迭代标志
clusterChanged = True
clusterAssment[i,:] = minIndex,minDist**2#重新分配簇结果
print (centroids)
for cent in range(k):
ptsInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]]#获取给定簇的所有点
centroids[cent,:] = mean(ptsInClust, axis=0)#沿矩阵的列方向计算它们的均值
return centroids, clusterAssment
def plotCentroids(datMat, centroids, clusterAssment, k):
fig = plt.figure()
ax = fig.add_subplot(111)
for i in range(k):
ax.scatter(datMat[[nonzero(clusterAssment[:,0] == float(i))[0]],0].flatten().A[0], datMat[[nonzero(clusterAssment[:,0] == float(i))[0]],1].flatten().A[0], marker="^", s=90)
ax.scatter(centroids[:,0].flatten().A[0], centroids[:,1].flatten().A[0], marker="+", s=300, c="black")
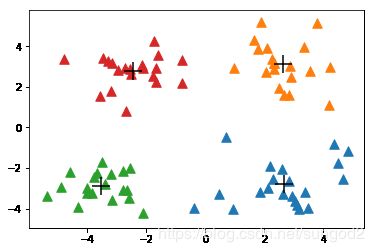
datMat = mat(loadDataSet('testSet.txt'))
myCentroids, clustAssing = kMeans(datMat,4)
[[ 2.05223983 -3.0746459 ]
[ 2.07512432 3.50918187]
[-2.18388394 -1.47117211]
[ 0.27725078 5.1426455 ]]
[[ 2.65077367 -2.79019029]
[ 2.66534547 2.99911595]
[-3.4859745 -2.31300105]
[-2.10585717 3.15782844]]
[[ 2.65077367 -2.79019029]
[ 2.6265299 3.10868015]
[-3.53973889 -2.89384326]
[-2.46154315 2.78737555]]
plotCentroids(datMat, myCentroids, clustAssing, 4)
- 上面的结果给出了四个质心。可以看到,经过几次迭代后K-均值算法收敛。到目前为止,关于聚类的一切进展都很顺利,但事实并不总是如此。接下来会讨论K-均值算法可能出现的问题及其解决办法。
1.2 使用后处理来提高聚类性能
-
前面提到的,在K-均值聚类中簇的数目k是一个用户预先定义的参数,那么用户怎么才能知道k的选择是否正确?如何才能知道生成的簇比较好呢?在包含簇分配结果的矩阵中保存着每个点的误差,即该点到簇质心的距离平方值。下面会讨论利用该误差来评价聚类质量的方法。
-
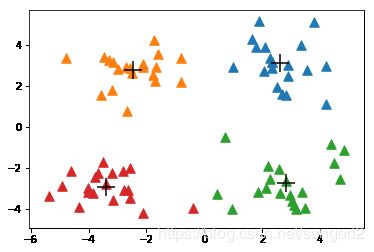
假如我们将k的值设为3,那么它的运行结果如下图所示。可以看出,点的簇分配结果值没有那么准确。K-均值算法收敛但聚类效果差的原因是,K-均值算法收敛到了局部最小值,而不是全局最小值。
-
一种用于度量聚类效果的指标是SSE(Sum of Squared Error,误差平方和),对应clusterAssment矩阵的第一列之和。SSE值越小表示数据点越接近它们的质心,聚类效果也越好。因为对误差取了平方,因此更重视那些远离中心的点。
-
一种肯定可以降低SSE值得方法是增加簇的个数,但这违背了聚类的目标。聚类的目标是在保持簇数目不变的情况下提高簇的质量。
-
如何进行改进:可以对生成的簇进行后处理,一种方法是将具有最大的SSE值得簇划分为两个簇。具体实现时可以将最大簇包含得点过滤出来并在这些点上运行K-均值算法。
-
为了保持簇总数不变,可以将某两个簇进行合并。
-
有两种可以量化得方法:合并最近的质心,或者合并两个使得SSE增幅最小的质心。第一种思路通过计算所有质心之间的距离,然后合并距离最近的两个点来实现。第二种方法需要合并两个簇然后计算总SSE值。必须在所有可能的两个簇上重复上述处理过程,直到找到合并最佳的两个簇为止。
datMat = mat(loadDataSet('testSet.txt'))
myCentroids, clustAssing = kMeans(datMat,4)
plotCentroids(datMat, myCentroids, clustAssing, 4)
[[ 1.96818879 3.10418929]
[-2.66894365 2.35308276]
[ 3.56299862 0.14036498]
[-4.42485328 -4.1647096 ]]
[[ 2.52792822 3.30405044]
[-2.46154315 2.78737555]
[ 2.8675685 -2.36043623]
[-3.38237045 -2.9473363 ]]
[[ 2.6265299 3.10868015]
[-2.46154315 2.78737555]
[ 2.80293085 -2.7315146 ]
[-3.38237045 -2.9473363 ]]
1.3 二分K-均值算法
-
为克服K-均值算法收敛于局部最小值的问题,有人提出了另一个称为二分K-均值(bisecting K-meams)的算法。该算法首先将所有点作为一个簇,然后将该簇一分为二。之后选择一个簇继续进行划分,选择哪一个簇进行划分取决于对其划分是否可以最大程度降低SSE的值。
-
伪代码:
将所有点看成一个簇
当簇数目小于k时
对于每一个簇
计算总误差
在给定的簇上面进行K-均值聚类(k=2)
计算将该簇一分为二之后的总误差
选择使得误差最小的那个簇进行划分操作 -
另一种做法是选择SSE最大的簇进行划分,直到簇数目达到用户指定的数目为止。
#二分K-均值聚类算法
def biKmeans(dataSet, k, distMeas=distEclud):
m = shape(dataSet)[0]
clusterAssment = mat(zeros((m,2)))#存储簇分配结果及平方误差
centroid0 = mean(dataSet, axis=0).tolist()[0]#计算整个数据集的质心
centList = [centroid0[0]]#使用一个列表保留所有质心
for j in range(m):
clusterAssment[j,1] = distMeas(mat(centroid0), dataSet[j,:])**2#计算每个点到质心的误差值
while(len(centList) < k):#该循环不停对簇进行划分,直到得到想要的簇的数目
lowestSSE = inf #将SSE值设为无穷大
for i in range(len(centList)): #遍历簇列表中的所有簇
ptsInCurrCluster = dataSet[nonzero(clusterAssment[:,0].A == i)[0],:]#将该簇中的所有点看成一个小的数据集。
centroidMat, splitClustAss = kMeans(ptsInCurrCluster, 2, distMeas)#生成两个质心同时给出每个簇的误差值
sseSplit = sum(splitClustAss[:,1])
sseNotSplit = sum(clusterAssment[nonzero(clusterAssment[:,0].A != i)[0],1])
print("sseSplit, and NotSplit: ", sseSplit, sseNotSplit)
if(sseSplit + sseNotSplit) < lowestSSE: #如果该划分的SSE值最小,则本次划分被保存
bestCentToSplit = i
bestNewCents = centroidMat
bestClustAss = splitClustAss.copy()
lowestSSE = sseSplit + sseNotSplit
bestClustAss[nonzero(bestClustAss[:,0].A == 1)[0], 0] = len(centList)#更新簇的分配结果
bestClustAss[nonzero(bestClustAss[:,0].A == 0)[0], 0] = bestCentToSplit
print("the bestCentToSplit is: ",bestCentToSplit)
print("the len of bestClustAss is: ",len(bestClustAss))
centList[bestCentToSplit] = bestNewCents[0,:].tolist()[0]
centList.append(bestNewCents[1,:].tolist()[0])
clusterAssment[nonzero(clusterAssment[:,0].A == bestCentToSplit)[0], :] = bestClustAss
return mat(centList), clusterAssment
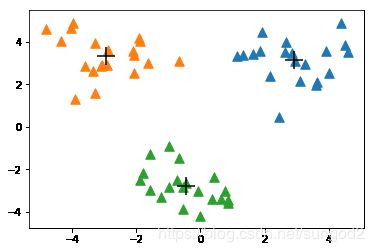
datMat3 = mat(loadDataSet('testSet2.txt'))
centList, myNewAssments = biKmeans(datMat3, 3)
centList
[[ 3.37991172 3.38570226]
[-3.10486239 -2.37443875]]
[[ 1.86138027 3.22269712]
[-1.70174271 -0.30206818]]
[[ 2.35797261 3.21160974]
[-1.72153338 -0.00938424]]
[[ 2.76275171 3.12704005]
[-1.73028592 0.20133246]]
[[ 2.93386365 3.12782785]
[-1.70351595 0.27408125]]
sseSplit, and NotSplit: 541.2976292649145 0.0
the bestCentToSplit is: 0
the len of bestClustAss is: 60
[[3.38922822 0.8721925 ]
[3.91147439 0.73983691]]
[[2.75314728 3.06695644]
[4.560311 3.6756705 ]]
[[2.48449707 2.95091147]
[4.2819634 3.658577 ]]
sseSplit, and NotSplit: 25.535514707587865 501.7683305828214
[[-2.71396475 1.10408322]
[-1.82642235 -3.1436269 ]]
[[-2.94737575 3.3263781 ]
[-0.45965615 -2.7782156 ]]
sseSplit, and NotSplit: 67.2202000797829 39.52929868209309
the bestCentToSplit is: 1
the len of bestClustAss is: 40
matrix([[ 2.93386365, 3.12782785],
[-2.94737575, 3.3263781 ],
[-0.45965615, -2.7782156 ]])
plotCentroids(datMat3, centList, myNewAssments, 3)
- 上述函数会运行多次,聚类会收敛到全局最小值,而原始的kMeans()函数偶尔会陷入局部最小值。
本章小结
- 聚类是一种无监督的学习方法。聚类区别于分类,即事先不知道要寻找的内容,没有预先设定好的目标变量。
- 聚类将数据点归到多个簇中,其中相似的数据点归为同一簇,而不相似的点归为不同的簇。相似度的计算方法有很多,具体的应用选择合适的相似度计算方法。
- K-means聚类算法,是一种广泛使用的聚类算法,其中k是需要指定的参数,即需要创建的簇的数目,K-means算法中的k个簇的质心可以通过随机的方式获得,但是这些点需要位于数据范围内。在算法中,计算每个点到质心得距离,选择距离最小的质心对应的簇作为该数据点的划分,然后再基于该分配过程后更新簇的质心。重复上述过程,直至各个簇的质心不再变化为止。
- K-means算法虽然有效,但是容易受到初始簇质心的情况而影响,有可能陷入局部最优解。为了解决这个问题,可以使用另外一种称为二分K-means的聚类算法。二分K-means算法首先将所有数据点分为一个簇;然后使用K-means(k=2)对其进行划分;下一次迭代时,选择使得SSE下降程度最大的簇进行划分;重复该过程,直至簇的个数达到指定的数目为止。实验表明,二分K-means算法的聚类效果要好于普通的K-means聚类算法。