基于知识图谱的问答系统1【代码学习系列】【知识图谱】【问答系统】
基于知识图谱的医疗领域问答系统1【QASystemOnMedicalKG】
1 代码来源
本代码来源于github项目地址,主要是构建以疾病为中心的具有一定规模的医药领域知识图谱,并以该知识图谱完成自动问答与分析服务。其中知识图谱通过neo4j构建,代码采用python语言。
2 项目组成
项目包括知识爬取、知识图谱构建、问答处理等三部分,其中问答处理可以进一步按照处理流程细分为三个部分,分别为实体意图提取、问答模板匹配和回答语句生成。
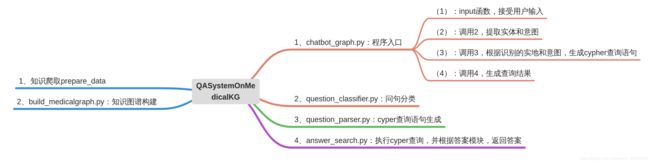
3 知识图谱构建
知识图谱构建包括知识准备和知识构建两部分,本项目知识准备通过网络爬虫从寻医问药网爬取医学知识,这里不再详细赘述。项目已经准备好了相关数据,data/medical.json,json格式。下面详细说明如何基于medical.json,构建知识图谱。需要的python包为py2neo,json,外部服务为neo4j。
(1)图谱设计
根据医学领域知识,围绕疾病,共设计7类节点(Disease、Drug、Food、Check、Department、Producer、Symptom),11种关系(Disease-(recommand_eat)>Food,Disease-(no_eat)>Food,Disease-(do_eat)>Food,Department-(belongs_to)>Department,Disease-(common_drug)>Drug,Disease-(recommand_drug)>Drug,
Producer-(drugs_of)>Drug,Disease-(need_check)>Check,Disease-(has_symptom)>Symptom,Disease-(acompany_with)>Disease,
Disease-(belongs_to)>Department)。
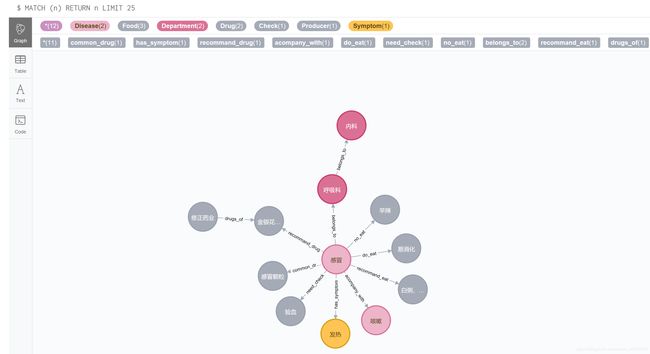
(2)图谱构建
主要采用py2neo接口,利用neo4j软件,构建知识图谱。
- 建立连接
// 建立连接
from py2neo import Graph,Node
g = Graph(host="127.0.0.1", # neo4j 搭载服务器的ip地址,ifconfig可获取到
http_port=7474, # neo4j 服务器监听的端口号
user="neo4j", # 数据库user name,如果没有更改过,应该是neo4j
password="123456")- 建立实体节点
// An highlighted block
node = Node("Disease", name=disease_dict['name'], desc=disease_dict['desc'],
prevent=disease_dict['prevent'] ,cause=disease_dict['cause'],
easy_get=disease_dict['easy_get'],cure_lasttime=disease_dict['cure_lasttime'],
cure_department=disease_dict['cure_department']
,cure_way=disease_dict['cure_way'] , cured_prob=disease_dict['cured_prob'])
g.create(node)- 建立实体关系变
// start_node,end_node为实体对应的名字,rel_type为recommand_eat、no_eat等,表示边类型,rel_name为推荐食谱、忌吃等边对应的名字。
query = "match(p:%s),(q:%s) where p.name='%s'and q.name='%s' create (p)-[rel:%s{name:'%s'}]->(q)" % (
start_node, end_node, p, q, rel_type, rel_name)
g.run(query)4 问答系统
问答系统包括三个模块,分别是输入语句的实体提取和意图分类、cypher语句生成和查询结果输出。
(1)实体提取和意图分类
实体提取是利用知识图谱中实体名字构成的特征库,建立AC树(Aho-Corasick Algorithm ),利用AC算法,匹配问句中是否存在特征词,即查询问句中是否存在知识图谱实体名字,来实现实体提取。此种提取方式为硬提取方式,不具有模糊推测能力。
- 模块载入
// 建立连接
import ahocorasickg- 建立AC树
// An highlighted block
// wordlist为7类实地所有名字构成的特征词库
actree = ahocorasick.Automaton()
for index, word in enumerate(wordlist):
actree.add_word(word, (index, word))
actree.make_automaton()- 实体提取
// wordlist为7类实地所有名字构成的特征词库
region_wds = []
for i in actree.iter(question):
wd = i[1][1] #匹配的实体名字
region_wds.append(wd)
stop_wds = []
for wd1 in region_wds:
for wd2 in region_wds:
if wd1 in wd2 and wd1 != wd2:
stop_wds.append(wd1)
final_wds = [i for i in region_wds if i not in stop_wds]
final_dict = {i:self.wdtype_dict.get(i) for i in final_wds} #self.wdtype_dict.get(i)获取实体名字对应的实体类别,Disease、Drug等意图分类则是通过判断输入问句中是否存在特定的实地类别和特定的意图词,来进行意图判断,属于模板匹配,共有15种意图模板。
- 建立意图特征词
// 建立意图特征词
symptom_qwds = ['症状', '表征', '现象', '症候', '表现']
cause_qwds = ['原因','成因', '为什么', '怎么会', '怎样才', '咋样才', '怎样会', '如何会', '为啥', '为何', '如何才会', '怎么才会', '会导致', '会造成'] 2.模板匹配
// 疾病症状类意图
def check_words(self, wds, sent):
for wd in wds:
if wd in sent:
return True
return False
if check_words(self.symptom_qwds, question) and ('disease' in types):
question_type = 'disease_symptom'
question_types.append(question_type)(2)cypher语句生成
该模块根据实体提取和意图分类返回的结果,对应生成cypher语句。
// cypher语句生成
if question_type == 'disease_symptom':
sql = sql_transfer(question_type, entity_dict.get('disease'))
def sql_transfer(self, question_type, entities):
sql = []
if question_type == 'disease_cause':
sql = ["MATCH (m:Disease) where m.name = '{0}' return m.name, m.cause".format(i) for i in entities](3)查询结果并格式化输出
该模块包括两个步骤,首先根据生产的cypher语句,连接neo4j图数据库,进行查询,获取相应结果。然后格式化查询结果,进行输出返回给用户。
- 图数据库查询
// 通过neo4j接口,进行查询
ress = g.run(sql).data()- 格式化输出
// 格式化输出
final_answer = '{0}的症状包括:{1}'.format(subject, ';'.join(list(set(desc))[:self.num_limit]))5 总结
该项目主要通过简单的模板匹配,基于知识数据库,进行问答,是一种入门级的问答系统,涉及的主要技术点为知识图谱的设计与构建,匹配模板设计。(2019年8月22日)