k-means聚类算法
2018.9.11
k-mean算法
- 解决什么问题
- 输入是什么
- 输出是什么
1. 解决什么问题:
k-means 算法接受输入量 k ;然后将n个数据对象划分为 k个聚类以便使得所获得的聚类满足:同一聚类中的对象相似度较高;而不同聚类中的对象相似度较小。聚类相似度是利用各聚类中对象的均值所获得一个“中心对象”(引力中心)来进行计算的。
说白了就是把数据按需求分成 k 类。
2. 输入:
待处理数据矩阵X,质心数量K
算法思想:
(1)随机选取K个初始质心
(2)分别计算所有样本到这K个质心的距离
(3)如果样本离质心Si最近,那么这个样本属于Si点群;如果到多个质心的距离相等,则可划分到任意组中
(4)按距离对所有样本分完组之后,计算每个组的均值(最简单的方法就是求样本每个维度的平均值),作为新的质心
(5)重复(2)(3)(4)直到新的质心和原质心相等,算法结束
MATLAB中有自带的kmeans函数,可以直接使用:
K-means聚类算法采用的是将N*P的矩阵X划分为K个类,使得类内对象之间的距离最大,而类之间的距离最小。
使用方法:
Idx=Kmeans(X,K)
[Idx,C]=Kmeans(X,K)
[Idc,C,sumD]=Kmeans(X,K)
[Idx,C,sumD,D]=Kmeans(X,K)
各输入输出参数介绍:
X—NP的数据矩阵
K—表示将X划分为几类,为整数
Idx—N1的向量,存储的是每个点的聚类标号
C—KP的矩阵,存储的是K个聚类质心位置
sumD—1K的和向量,存储的是类内所有点与该类质心点距离之和
D—N*K的矩阵,存储的是每个点与所有质心的距离
[┈]=Kmeans(┈,’Param1’,’Val1’,’Param2’,’Val2’,┈)
其中参数Param1、Param2等,主要可以设置为如下:
1、’Distance’—距离测度
‘sqEuclidean’—欧氏距离
‘cityblock’—绝对误差和,又称L1
‘cosine’—针对向量
‘correlation’—针对有时序关系的值
‘Hamming’—只针对二进制数据
2、’Start’—初始质心位置选择方法
‘sample’—从X中随机选取K个质心点
‘uniform’—根据X的分布范围均匀的随机生成K个质心
‘cluster’—初始聚类阶段随机选取10%的X的子样本(此方法初始使用’sample’方法)
Matrix提供一K*P的矩阵,作为初始质心位置集合
3、’Replicates’—聚类重复次数,为整数
https://www.jb51.net/article/49395.htm
%随机获取300个点,正态均匀
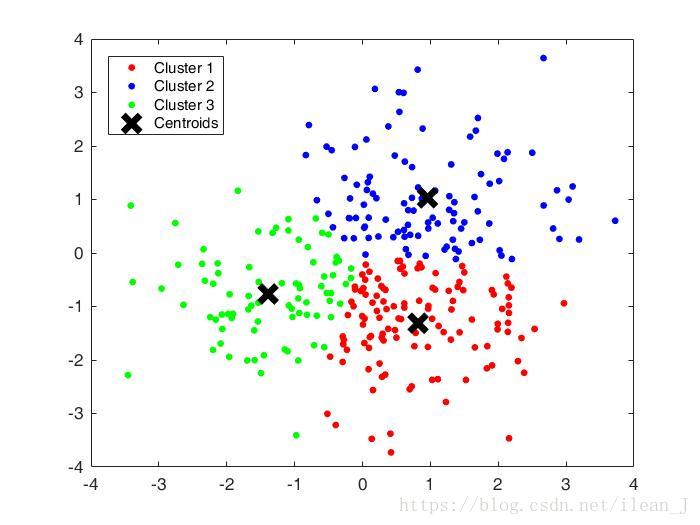
X = [randn(100,2)+ones(100,2);randn(100,2)-ones(100,2);randn(100,2)+[ones(100,1),-ones(100,1)]];
opts = statset('Display','final');
%调用Kmeans函数
%X N*P的数据矩阵
%Idx N*1的向量,存储的是每个点的聚类标号
%Ctrs K*P的矩阵,存储的是K个聚类质心位置
%SumD 1*K的和向量,存储的是类间所有点与该类质心点距离之和
%D N*K的矩阵,存储的是每个点与所有质心的距离;
[Idx,Ctrs,SumD,D] = kmeans(X,3,'Replicates',3,'Options',opts);
%画出聚类为1的点。X(Idx==1,1),为第一类的样本的第一个坐标;X(Idx==1,2)为第一类的样本的第二个坐标,相当于plot(x,y)了
plot(X(Idx==1,1),X(Idx==1,2),'r.','MarkerSize',14)
hold on
plot(X(Idx==2,1),X(Idx==2,2),'b.','MarkerSize',14)
hold on
plot(X(Idx==3,1),X(Idx==3,2),'g.','MarkerSize',14)
%绘出聚类中心点
plot(Ctrs(:,1),Ctrs(:,2),'kx','MarkerSize',14,'LineWidth',4)
hold on
legend('Cluster 1','Cluster 2','Cluster 3','Centroids','Location','NW')
以上是指定 k=3 的情况,接下来稍微改动,把它变成一个数据给定,k自己输入的程序
%随机获取300个点,正态均匀
%X = [randn(100,2)+ones(100,2);randn(100,2)-ones(100,2);randn(100,2)+[ones(100,1),-ones(100,1)]];
% x=imread('touxiang.jpg');
% X=reshape(x,[138450,2]);
X = xlsread('随机数据1.xlsx'); %这里是把随机数存到了Excel里
opts = statset('Display','final');
%调用Kmeans函数
%X N*P的数据矩阵
%Idx N*1的向量,存储的是每个点的聚类标号
%Ctrs K*P的矩阵,存储的是K个聚类质心位置
%SumD 1*K的和向量,存储的是类间所有点与该类质心点距离之和
%D N*K的矩阵,存储的是每个点与所有质心的距离;
k = input('(k最好是<=6)input k='); %超出也不是不可以,就是可能会两个一样颜色的类连一起
pause(2);
[Idx,Ctrs,SumD,D] = kmeans(X,k,'Replicates',3,'Options',opts);
Idx = mod(Idx,6)+1;
%画出聚类为1的点。X(Idx==1,1),为第一类的样本的第一个坐标;X(Idx==1,2)为第一类的样本的第二个坐标
plot(X(Idx==1,1),X(Idx==1,2),'r.','MarkerSize',14)
hold on
plot(X(Idx==2,1),X(Idx==2,2),'b.','MarkerSize',14)
hold on
plot(X(Idx==3,1),X(Idx==3,2),'g.','MarkerSize',14)
hold on
plot(X(Idx==4,1),X(Idx==4,2),'k.','MarkerSize',14)
hold on
plot(X(Idx==5,1),X(Idx==5,2),'m.','MarkerSize',14)
hold on
plot(X(Idx==6,1),X(Idx==6,2),'y.','MarkerSize',14)
hold on
%绘出聚类中心点
plot(Ctrs(:,1),Ctrs(:,2),'kx','MarkerSize',14,'LineWidth',4)
hold on
% legend('Cluster 1','Cluster 2','Cluster 3','Centroids','Location','NW')
分簇的效果衡量指标
- 均一性:一个簇只包含一种样本
- 完整性:同类样本被归到一个簇,所有样本均被归类
- 个人觉得可以求相关系数,每两个簇之间求一次,若均为0或很小,则分的对
(适用于线性关系)
X = xlsread('随机数据1.xlsx');
opts = statset('Display','final');
k = input('input k=?');
pause(2);
[Idx,Ctrs,SumD,D] = kmeans(X,k,'Replicates',3,'Options',opts);
%画出聚类为1的点。X(Idx==1,1),为第一类的样本的第一个坐标;X(Idx==1,2)为第一类的样本的第二个坐标
plot(X(Idx==1,1),X(Idx==1,2),'r.','MarkerSize',14)
hold on
plot(X(Idx==2,1),X(Idx==2,2),'b.','MarkerSize',14)
hold on
plot(X(Idx==3,1),X(Idx==3,2),'g.','MarkerSize',14)
hold on
plot(X(Idx==4,1),X(Idx==4,2),'k.','MarkerSize',14)
hold on
plot(X(Idx==5,1),X(Idx==5,2),'m.','MarkerSize',14)
hold on
plot(X(Idx==6,1),X(Idx==6,2),'y.','MarkerSize',14)
hold on
%绘出聚类中心点
plot(Ctrs(:,1),Ctrs(:,2),'kx','MarkerSize',14,'LineWidth',4)
hold on
Z=var(Ctrs);
CO=cov(Ctrs');
varx=Z(1,1);
vary=Z(1,2);
SQ=sqrt(varx*vary);
COVxy=det(CO);
pxy=COVxy/SQ;
fprintf('pxy is %s',pxy);
% disp(pxy);
fprintf('abs(pxy) is %s',abs(pxy));
2.compactness(紧密性)(CP)
各点到聚类中心的平均距离,CP越小间距越近
cp = sum(SumD,1)./300;
fprintf('cp is %s',cp);
3.Separation(间隔性)(SP)
SP计算各聚类中心两两之间平均距离、SP越高意味类间聚类距离越远
%sp
sp=0;
for i=1:k
for j=i+1:k
sp=sqrt((Ctrs(i,1)-Ctrs(j,1)).^2+(Ctrs(i,2)-Ctrs(j,2)).^2)+sp;
end
end
fprintf('sp is %s',sp./k);
4.Davies-Bouldin Index(戴维森堡丁指数)(分类适确性指标)(DB)(DBI)
DB计算 任意两类别的类内距离平均距离(CP)之和除以两聚类中心距离、求最大值
DB越小意味着类内距离越小、同时类间距离越大
%db
db=0;
for i=1:k-1
db1=(SumD(i,1)+SumD(i+1,1))./sqrt((Ctrs(i,1)-Ctrs(i+1,1)).^2+(Ctrs(i,2)-Ctrs(i+1,2)).^2);
if db1>db
db=db1;
end
end
fprintf('db is %s',db);
5.Dunn Validity Index (邓恩指数)(DVI)
DVI计算 任意两个簇元素的最短距离(类间)除以任意簇中的最大距离(类内)
DVI越大意味着类间距离越大 同时类内距离越小
缺点:对离散点的聚类测评很高、对环状分布测评效果差
%dvi
db=2.4179e+24;
for i=1:k-1
db1=(SumD(i,1)+SumD(i+1,1))./sqrt((Ctrs(i,1)-Ctrs(i+1,1)).^2+(Ctrs(i,2)-Ctrs(i+1,2)).^2);
if db1参考资料:
https://blog.csdn.net/sinat_33363493/article/details/52496011
https://blog.csdn.net/kwame211/article/details/78037410