题目来自生信技能树论坛
下载好的文件大概格式如下
简单的了解一下都有什么
A开头的
grep "^A" hsa00001.keg
B开头的
grep "^B" hsa00001.keg
C开头的(这一行是pathway行)
grep "^C" hsa00001.keg
黄色区域就表示kegg中有pathway
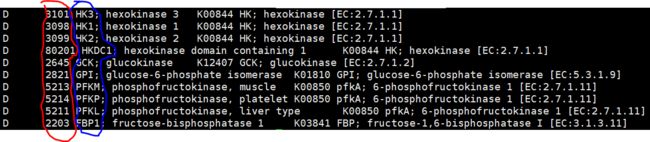
D开头的
grep "^D" hsa00001.keg |head
红色区域圈出来的数字是gene ID
蓝色区域圈出来的字母是gene symbol
好了,以上就是了解数据的形式
现在要做到的是想得到如下格式的文件
共5列文件
这里有个问题先提一下
我在less -S 下匹配了一个“ ”,但是第一列和第二列之间的空格全部匹配上了,这肯定不能说明第一列和第二列之间只有 一个空格,所以如果读取文件的时候,以“ ”split的时候,想取想要的值的时候很容易把索引弄错。
所以这题用正则表达式
脚本如下
import sys
import collections
import re
args=sys.argv
filename=args[1]
keggD=collections.OrderedDict()
with open (filename) as fh:
for line in fh:
if line.startswith("A"):
mth=re.search('A(\S+)(\s+)(.+)',line) #"A(\S+)(\s+)(.+)" 指的是匹配以A开头,后面跟的是一个或多个非空白字符,
cate1=mth.group(3) #再跟着的是一个或多个空白字符,再后面跟一个或多个除换行符外的字符。/表示转义,A没必要转义
keggD[cate1]=collections.OrderedDict()
if line.startswith("B"):
if len(line)== 2:
continue
mth=re.search('(\d+)(\s+)(.+)',line) #'(\d+)(\s+)(.+)'指的是匹配一个或多个数字,后面跟着的是一个或多个空白字符,再后面跟着的是一个或多个
cate2=mth.group(3) # 除换行符外的任意字符
keggD[cate1][cate2]=collections.OrderedDict()
if line.startswith("C"):
mth=re.search('(\d+)(\s+)(.+)',line) # '(\d+)(\s+)(.+)'同上
pathID="hsa"+mth.group(1)
pathname=mth.group(3)
pathname=re.sub("(\s+)\[(\S+)\]","",pathname) # (\s+)\[(\S+)\] 匹配一个或多个空白字符,后面跟着的是[,再后面跟着的是一个或多个非空白字符,再跟]
pathway=pathID+":"+pathname
keggD[cate1][cate2][pathway]=[[],[]]
if line.startswith("D"):
lst=line.split(";")
geneinfo=lst[0].split("\t")[0]
mth=re.search("(\d+)(\s+)(\S+)",geneinfo) #"(\d+)(\s+)(\S+)" 匹配的是一个或多个数字字符,后面跟一个或多个空白字符,再后面跟的是一个或多个非空白字符 gene_ID=mth.group(1)
gene_symbol=mth.group(3)
keggD[cate1][cate2][pathway][0].append(gene_ID)
keggD[cate1][cate2][pathway][1].append(gene_symbol)
for cate1,value1 in keggD.items():
for cate2,value2 in value1.items():
for pathway,value3 in value2.items():
geneIDs=";".join(value3[0])
genes=";".join(value3[1])
print(cate1,cate2,pathway,geneIDs,genes,sep="\t")
这里需要说明D开头的读取方法和其他的不同的原因在于,并不是所有都是这么写的
D 3101 HK3; hexokinase 3 K00844 HK; hexokinase [EC:2.7.1.1]
有的可能是
D 3101 HK3 XXXX; hexokinase 3 K00844 HK; hexokinase [EC:2.7.1.1]
所以需要统一
这一条单独拿出来,要懂得怎么去写
keggD[cate1][cate2][pathway]=[[],[]]
还有就是
pathname=re.sub("(\s+)\[(\S+)\]","",pathname)
这里是把中括号在内以及括号除掉
如果是只去掉中括号,比如《生信编程实战第一题》中提到的
pathname=re.sub("(\s+)\[|\]","",pathname)
再就是统计一下,总共有多少pathway,以及总共多少个基因,针对上一步生成的文件
import sys
args=sys.argv
filename=args[1]
cnt_pathway=0
all_gene=[]
for line in open(filename):
if len(line)>3 :
cnt_pathway += 1
lineL=line.split("\t")
gene_symL=lineL[-1] .split(";")
for gene in gene_symL:
if gene not in all_gene:
all_gene.append(gene)
print(cnt_pathway,len(all_gene))
~
python3 count.py hsa.kegg.txt
513 13972
最后把python正则表达式的知识补充一下
1.re.match函数
re.match 尝试从字符串的起始位置匹配一个模式,如果不是起始位置匹配成功的话,match()就返回none
语法: re.match(pattern, string, flags=0)
举例子:
import re
mth1=re.match("ATG","ATGCGCGCGCGC")
mth2=re.match("ATG","GATGCGCGCGCGC")
mth1.group()
mth2.group()
'ATG'
none
2.re.search函数
re.search 扫描整个字符串并返回第一个成功的匹配
语法: re.search(pattern, string, flags=0)
举例子:
import re
mth1=re.search("ATG","ATGCGCGCGCGC")
mth2=re.search("ATG","GATGCGCGCGCGC")
mth1.group()
mth2.group()
'ATG'
'ATG'
由上,可以很容易很出re.match 和re.search的区别,
re.match只匹配字符串的开始,如果字符串开始不符合正则表达式,则匹配失败,函数返回None;而re.search匹配整个字符串,直到找到一个匹配。
所以实际上,re.search 应该更实用
3.检索和替换
Python 的 re 模块提供了re.sub用于替换字符串中的匹配项
语法 : re.sub(pattern, repl, string, count=0, flags=0)
举个例子
import re
a=" tomorrow is valentine's day ,but you have to go to shanghai "
a=re.sub("(\s+)t","t",a)
a
"tomorrow is valentine's day ,but you haveto goto shanghai "
4.re.compile 函数
compile 函数用于编译正则表达式,生成一个正则表达式( Pattern )对象,供 match() 和 search() 这两个函数使用
语法 : re.compile(pattern[, flags])
其实这个实际上就是将要匹配的对象用re.compile包装了一下,将要匹配的对象赋值给pattern,然后原来的re.match和re.search改成pattern.match和pattern.search
,然后括号里面的要匹配的对象去掉就好了。其实一样。
比如,上面的例子
import re
pattern=re.compile("ATG")
mth1=pattern.match("ATGCGCGCGCGC")
mth1.group()
'ATG'
这里还需要提一个知识点
正则表达式中group()为输出结果
对于有括号的():
group()表示就是匹配正则表达式整体结果
group(1)表示匹配正则表达式中第一个括号内的内容
group(2)表示匹配正则表达式中第二个括号内的内容
.
.
.
5.findall
在字符串中找到正则表达式所匹配的所有子串,并返回一个列表,如果没有找到匹配的,则返回空列表
注意: match 和 search 是匹配一次 findall 匹配所有。
显然这个findall也是可以用re.compile的
但我的例子先不用吧
还是上面的例子
import re
a=" tomorrow is valentine's day ,but you have to go to shanghai "
a=re.findall("\S+\s+",a)
a
['tomorrow ',
'is ',
"valentine's ",
'day ',
',but ',
'you ',
'have ',
'to ',
'go ',
'to ',
'shanghai ']
6.re.finditer
7.re.split