- 定积分及其在概率论与统计学中的应用
AI天才研究院
DeepSeekR1&大数据AI人工智能大模型AI大模型企业级应用开发实战计算科学神经计算深度学习神经网络大数据人工智能大型语言模型AIAGILLMJavaPython架构设计AgentRPA
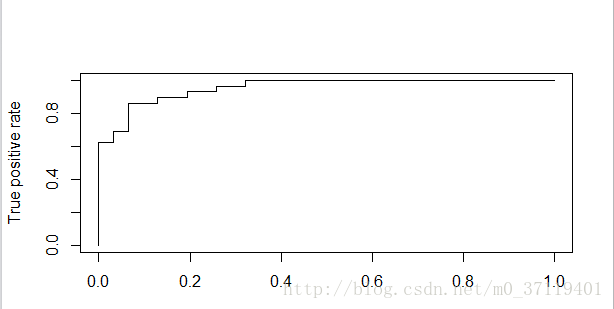
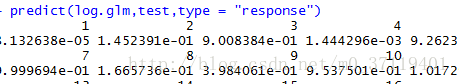
定积分及其在概率论与统计学中的应用1.背景介绍1.1定积分的概念定积分是微积分学中一个基本概念,它是对连续函数在一个区间上的累积变化量进行测度。定积分可以看作是对无限小量的累加,是对函数在给定区间内的面积进行测量。1.2定积分在概率论与统计学中的重要性在概率论和统计学中,定积分扮演着非常重要的角色。概率论中的概率密度函数、累积分布函数等核心概念都需要借助定积分来定义和计算。统计学中的置信区间估计、
- AI人工智能中的概率论与统计学原理与Python实战:Python实现概率模型
AI天才研究院
AI实战AI大模型企业级应用开发实战大数据人工智能语言模型AILLMJavaPython架构设计AgentRPA
1.背景介绍随着人工智能技术的不断发展,概率论与统计学在人工智能领域的应用越来越广泛。概率论与统计学是人工智能中的基础知识之一,它们在机器学习、深度学习、自然语言处理等领域都有着重要的作用。本文将介绍概率论与统计学的核心概念、算法原理、具体操作步骤以及Python实现方法,并通过具体代码实例进行详细解释。2.核心概念与联系2.1概率论与统计学的区别概率论是一门数学学科,它研究随机事件发生的可能性。
- 运维面试常问的100道题(大数据统计)
無爲謂
人工智能运维面试
一、基础知识类1、请解释什么是运维?运维是指对企业的IT系统进行运行维护,包括硬件设备、软件系统、网络等的监控、管理、优化和故障处理,以确保系统的稳定、高效运行,满足业务需求。2、简述运维的主要职责有哪些?服务器的安装、配置、维护和监控。网络设备的管理和维护。数据库的管理和维护。应用系统的部署、升级和维护。故障处理和应急响应。性能优化和容量规划。安全管理和漏洞修复。3、什么是服务器?有哪些类型?服
- 西安电子科技大学 | 近期官方视频+文章(涵盖招生+录取统计)
西电研梦
西电经验分享考研
今年、明年考研的学弟学妹们可以看看涵盖今年讲课西电录取统计【西安电子科技大学官方】研究生招生宣传你一定值得一看02、计算机科学与技术学院(近期视频+文章)QQ群:867684593①、西电考研|833、834初+复试第一名许学长经验分享https://www.bilibili.com/video/BV1oE4m1R72a/②、25西电考研|计科院专业课139分学长经验分享https://www.b
- 【第14届蓝桥杯】软件赛CB组省赛
Guiat
算法竞赛真题题解蓝桥杯
个人主页:Guiat归属专栏:算法竞赛真题题解文章目录A.日期统计B.01串的熵C.冶炼金属D.飞机降落E.接龙数列F.岛屿个数G.子串简写H.整数删除I.景区导游J.砍树正文总共10道题。A.日期统计【题目】日期统计【分析】【答案】235【AC_Code】#include#defineIOSios::sync_with_stdio(0);cin.tie(0);cout.tie(0);usingn
- 突破反爬终极指南:如何用Python实现100%隐形数据抓取(附实战代码)
煜bart
机器人人工智能web3.py
引言:当爬虫遭遇铜墙铁壁2023年Q2最新统计显示,全球Top100网站中89%部署了AI驱动的反爬系统,传统爬虫存活率暴跌至17%。本文将揭秘一套基于深度伪装技术的爬虫方案,在最近三个月实测中保持100%成功率,成功突破Cloudflare、Distil等顶级防护系统。---###一、指纹伪装:让爬虫"隐身"的核心科技####1.1浏览器指纹深度克隆(代码实现)```pythonfromsele
- 苹果签名的战略价值:解析六大核心优势与商业赋能逻辑
ios
苹果签名的战略价值:解析六大核心优势与商业赋能逻辑(因篇幅过长所以分为两篇帖子发~)在iOS应用生态中,签名机制既是技术护城河,也是开发者突破分发限制的关键武器。本文将从开发效率、商业变现、安全管控等维度,深度剖析苹果签名体系带来的独特价值。一、突破AppStore审核壁垒,加速产品验证苹果签名最核心的优势在于规避冗长审核流程。根据2023年统计,AppStore平均审核周期为24小时,首次提交通
- 养生,开启健康生活之门
yy0821yy
生活
在这个快节奏的时代,人们在忙碌奔波中,往往忽略了自身健康。养生保健,不再是老年人的专属,而是各个年龄段维持良好生活状态的关键,它是我们开启健康生活的一把钥匙。规律作息是养生的基石。人体就像一台精密的仪器,各个器官按照生物钟有条不紊地运转。熬夜追剧、通宵工作,看似是在争取时间,实则是在打乱身体的正常节奏。长期熬夜会导致内分泌失调,脸上冒出痘痘,精神萎靡不振,免疫力下降,各种疾病也容易找上门来。因此,
- 2025-03-15 学习记录--C/C++-PTA 练习3-4 统计字符
小呀小萝卜儿
学习-C/C++学习c语言
合抱之木,生于毫末;九层之台,起于累土;千里之行,始于足下。一、题目描述⭐️练习3-4统计字符本题要求编写程序,输入10个字符,统计其中英文字母、空格或回车、数字字符和其他字符的个数。输入格式:输入为10个字符。最后一个回车表示输入结束,不算在内。输出格式:在一行内按照letter=英文字母个数,blank=空格或回车个数,digit=数字字符个数,other=其他字符个数的格式输出。输入样例:a
- 第二十四篇 SQL优化之高级用法
随缘而动,随遇而安
SQL之道——从入门到精通数据库sql
目录一、调优基本功:看懂SQL的"体检报告"1.1给SQL做体检(执行计划分析)1.2给数据表"建索引"的正确姿势二、不改SQL也能提速的秘籍2.1分区表:把大象切成牛排2.2物化视图:给常用查询拍快照三、SQL改写:让你的查询脱胎换骨3.1告别全表扫描:日期查询优化3.2子查询转JOIN:速度提升10倍四、高级SQL:让你的代码更优雅4.1分组统计绝招(GROUPBY扩展)4.2MERGE语句:
- 新手村:数据预处理-异常值检测方法
嘉羽很烦
机器学习机器学习
机器学习中异常值检测方法一、前置条件知识领域要求编程基础Python基础(变量、循环、函数)、JupyterNotebook或PyCharm使用。统计学基础理解均值、中位数、标准差、四分位数、正态分布、Z-score等概念。机器学习基础熟悉监督/无监督学习、分类、聚类、回归等基本概念。数据预处理数据清洗、特征缩放(标准化/归一化)、数据可视化(Matplotlib/Seaborn)。二、渐进式学习
- python记录运行时间_计算python程序运行时间
weixin_39668408
python记录运行时间
本文介绍三种方法用来计算python程序的运行时间,考虑多现在计算机都是多进程执行环境,本文介绍的方法,前两种统计了其它进程的时间,实际上是python程序执行开始和结束的时间,只有最后一个方法,是计算的python程序的独自占用的CPU时间,但是python官方已经不再推荐。各位同学可以根据自己的应用情况选择考虑。1,用datatime模块,秒级精度>>>importdatetime>>>dat
- R语言的移动应用开发
滕若岚
包罗万象golang开发语言后端
R语言的移动应用开发在数据科学和统计分析的大潮中,R语言因其强大的数据处理和可视化能力而备受青睐。然而,R语言对移动应用开发的适用性并未得到广泛关注。本文将探讨R语言在移动应用开发中的潜力及其工具,并提供一些实践示例,希望能为读者在这一新兴领域的探索提供帮助。一、R语言概述R语言是一种用于统计计算和图形绘制的编程语言,因其简洁的语法和丰富的包生态系统受到数据科学家的广泛使用。R语言的强大之处在于其
- C++设计模式-原型模式:从基本介绍,内部原理、应用场景、使用方法,常见问题和解决方案进行深度解析
牵牛老人
C++专栏c++设计模式原型模式
一、基本介绍:理解"克隆"的设计哲学1.1什么是原型模式原型模式(PrototypePattern)是一种创建型设计模式,其核心思想是通过复制现有对象来创建新对象,而非传统的新建实例方式。如同生物学的细胞分裂机制,原型对象作为"母体",通过自我复制产生完全相同的新个体。该模式在C++中通常借助拷贝构造函数或克隆接口实现,特别适用于以下场景:对象初始化成本高昂(如需要读取大文件);系统需要动态生成多
- 四个学生三门成绩 C语言二维数组,C语言程序设计第6章-6.2.pptx
马宇宸
四个学生三门成绩C语言二维数组
C语言程序设计第6章-6.2.pptxC语言程序设计实例教程6.2二维数组,C语言程序设计实例教程,第6章数组,二维数组的定义和数组元素的引用方法二维数组的初始化方法,本节要点实例21二维数组的定义与引用-统计总成绩及平均成绩,【实例任务】从键盘上任意输入某班n个学生的三门课程的成绩,计算每个学生的平均成绩、计算每门课程的平均成绩,并且打印成绩单,输出三门课程成绩的平均分及课程的平均分。运行结果如
- 蓝桥杯-刷题(铺地毯,多项式输出,玩具谜题,乒乓球,数字统计,明明的随机数)
一个人在码代码的章鱼
刷题蓝桥杯算法职场和发展
铺地毯题目描述为了准备一个独特的颁奖典礼,组织者在会场的一片矩形区域(可看做是平面直角坐标系的第一象限)铺上一些矩形地毯。一共有n张地毯,编号从1到n。现在将这些地毯按照编号从小到大的顺序平行于坐标轴先后铺设,后铺的地毯覆盖在前面已经铺好的地毯之上。地毯铺设完成后,组织者想知道覆盖地面某个点的最上面的那张地毯的编号。注意:在矩形地毯边界和四个顶点上的点也算被地毯覆盖。输入格式输入共n+2行。第一行
- 【数据分享】我国分省份的七普乡镇(街道)人口数据
小鲨鱼-立方数据学社
数据分享立方数据学社人口普查七普数据乡镇人口人口数据
人口数据是我们在各项研究中都经常使用的数据!人口数据的主要来源是人口普查,全国性的人口普查每十年进行一次。最近一次的人口普查是第七次全国人口普查,简称七普。七普统计的是2020年的人口数据。之前我们分享过省市县三个层级的七普的人口数据(可查看之前的文章获悉详情),很多小伙伴在咨询有没有到乡镇(城市地区对应街道)层级的七普人口数据!本次我们为大家分享的就是来自七普的乡镇(街道)人口数据,包括地区;总
- 【数据分享】1999—2023年地级市地方一般公共预算收支状况数据(科学技术支出/教育支出等)
小鲨鱼-立方数据学社
数据分享中国城市统计年鉴地方一般公共预算收支状况立方数据学社
在之前的文章中,我们分享过基于2000-2024年《中国城市统计年鉴》整理的1999-2023年地级市的人口相关数据、染物排放和环境治理相关数据、房地产投资情况和商品房销售面积相关指标数据和社会消费品零售总额和年末金融机构存贷款余额(均可查看之前的文章获悉详情)!本次我们分享的是从2000—2024年《中国城市统计年鉴》整理的地级市一般公共预算收支状况!数据年份为1999-2023年,对于数据年份
- Python精进系列: K-Means 聚类算法调用库函数和手动实现对比分析
进一步有进一步的欢喜
Python精进系列算法pythonkmeans
一、引言在机器学习领域,聚类分析是一种重要的无监督学习方法,用于将数据集中的样本划分为不同的组或簇,使得同一簇内的样本具有较高的相似性,而不同簇之间的样本具有较大的差异性。K-Means聚类算法是最常用的聚类算法之一,它以其简单性和高效性在数据挖掘、图像分割、模式识别等领域得到了广泛应用。本文将详细介绍K-Means聚类算法,并分别给出调用现成函数和不调用任何现成函数实现K-Means聚类的代码示
- 计算机视觉入门
109702008
人工智能#深度学习计算机视觉人工智能
计算机视觉(ComputerVision)是一门涉及使机器能够从图像或者多维数据中提取信息,解释、理解并对物体或场景进行处理的学科。以下是一个基本的计算机视觉入门学习路线,旨在为刚刚接触这一领域的学习者提供指导。1.基础知识储备数学基础:线性代数、概率论和数理统计、微积分、优化理论。编程语言:掌握至少一门编程语言,Python是目前在计算机视觉领域最流行的语言,其次是C++。2.计算机视觉基础数字
- 计算机视觉(Computer Vision, CV)的入门到实践的详细学习路线
云梦优选
计算机数据库大数据计算机视觉学习人工智能
一、基础准备1.数学基础线性代数深入矩阵运算,理解矩阵乘法、转置、逆等基本概念。掌握特征值与特征向量的几何意义,理解其在图像压缩、特征提取中的应用。学习奇异值分解(SVD)及其在降维和数据压缩中的具体应用。概率与统计熟悉贝叶斯定理及其在分类任务中的应用,如朴素贝叶斯分类器。理解常见概率分布(如正态分布、二项分布)及其性质。学习统计推断方法,如假设检验、置信区间估计,以评估模型性能。微积分掌握梯度、
- 新手村:统计量均值、中位数、标准差、四分位数
嘉羽很烦
机器学习均值算法算法
新手村:统计量均值、中位数、标准差、四分位数统计量定义与讲解统计量定义计算公式示例说明均值数据集中的所有数值之和除以数值的个数。Mean=∑i=1nxin\text{Mean}=\frac{\sum_{i=1}^{n}x_i}{n}Mean=n∑i=1nxi对于数据集[1,2,3,4,5],均值为(1+2+3+4+5)/5=3(1+2+3+4+5)/5=3(1+2+3+4+5)/5=3中位数将数据
- 第九届蓝桥杯大赛软件赛省赛C/C++ 大学 B 组 递增三元组
徽京人
蓝桥解析蓝桥杯c语言c++
递增三元组题目描述给定三个整数数组A=[A1,A2,⋯AN]A=[A1,A2,⋯AN],B=[B1,B2,⋯BN]B=[B1,B2,⋯BN],C=[C1,C2,⋯CN]C=[C1,C2,⋯CN],请你统计有多少个三元组(i,j,k)(i,j,k)满足:1≤i,j,k≤N1≤i,j,k≤N;Aiusingnamespacestd;constintN=1e5+10;inta[N],b[N],c[N],
- MATLAB代码开发实战:从入门到高效应用
vvvae1234
matlab开发语言
一、MATLAB生态系统的核心优势(扩展原有内容,增加行业数据)MATLAB在全球工程领域的市场占有率已达67%(2024年IEEE统计),其核心优势体现在:矩阵运算速度比传统编程快3-5倍包含22个专业工具箱的完整工具链与硬件设备(如Arduino)的即插即用接口自动生成C/C++代码的部署能力案例佐证:2023年NASA火星探测器使用MATLAB/Simulink完成97%的导航算法验证二、代
- Redis缓存判断热点数据及进行数据预热的几种方式介绍
hxj..
分布式缓存redis数据库热点数据数据预热
Redis缓存如何判断热点数据?热点数据计算整体来讲就是基于访问频率,可以是整体的访问次数,可以是一定时间内的频率,可以是部分请求的采样,可以借助成熟工具等,要根据业务需求来定1.基于访问频率原理:通过统计每个键的访问频率(如每秒访问次数),识别出访问频率最高的数据。实现方法:使用Redis的INCR命令或监控工具(如RedisMonitor)统计键的访问频率。统计访问频率要确保并发场景下数据操作
- Python----数据分析(Pandas四:一维数组Series的统计计算,分组和聚合)
蹦蹦跳跳真可爱589
数据分析Pythonpandaspython数据分析
一、统计计算1.1、count用于计算Series中非NaN(非空)值的数量。importpandasaspds=pd.Series([1,2,None,4,None])count_non_na=s.count()print(count_non_na)1.2、sumsum()函数会计算所有值的总和。Series.sum(axis=None,skipna=True,numeric_only=None
- 订单管理系统,大学生数据结构期末作业/C语言实践作业
陌路物是人非
排序算法数据结构算法
任务:订单管理系统的设计与实现设计并实现一个订单管理系统界面分成两部分,分别是管理员和用户的界面主要功能:用户:(1)用户的登录及注册(2)用户信息修改(3)购买物品(4)充值(5)升序排序(按金额)物品管理员:(1)显示所有订单(2)插入订单信息(3)删除订单信息(4)排序订单(快排按编号)(5)统计订单信息(6)添加物品注意事项:一共需要建立4个文件(key.txt、物品清单.txt、用户信息
- CNBr活化琼脂糖凝胶4B,CNBr-Activated Sepharose 4B
陕西星贝爱科
CNBr活化琼脂糖凝胶4B
CNBr活化琼脂糖凝胶4B是一种用于固定含伯胺配基的预活化填料,以下是其详细介绍:基本信息中文名称:溴化氰活化琼脂糖凝胶4B英文名称:CNBr-ActivatedSepharose4B外观:白色浆状物,放置可分层/白色粉末状固体基架:4%琼脂糖颗粒大小:45~165μm偶联官能团:伯氨基储存条件:2~8℃,100%丙酮特点反应条件温和:与蛋白等生物大分子的反应条件温和,可直接偶联生物大分子,不需偶
- Python3 【项目实战】深度解析:赛跑成绩统计分析工具
李智 - 重庆
Python精讲精练-从入门到实战python案例学习编程技巧时间处理项目实战
Python3【项目实战】深度解析:赛跑成绩统计分析工具一、项目概述1.开发背景:田径比赛的成绩统计需要快速准确的计算选手成绩,传统人工计时和统计效率低且易出错。本工具通过程序化处理赛跑数据,自动计算各选手成绩及整体统计指标,主要应用于:学校运动会成绩实时统计田径锦标赛的自动化成绩公示运动员训练数据分析2.技术定位:时间数据处理与统计计算的典型案例字典数据结构的实践应用面向过程编程的教学范例二、项
- ❤【纯干货】Matplotlib总结,任何项目都用得到❤
.Boss.
支持向量机人工智能算法机器学习python
.................❤纯干货❤..........目录1、绘制基本图形2、定制化图形3、支持多个坐标轴4、3D绘图5、动态交互绘图6、绘制地图7、绘制统计图表最后Matplotlib在很多人眼里是无敌的存在,而且可以说是无敌的存在。走过数据科学的路,路上必然有Matplotlib的风景在你周围。如果同一个项目,你的用了matplotlib不仅有基本图形、定制化图形、多个坐标轴、3D绘
- java数字签名三种方式
知了ing
javajdk
以下3钟数字签名都是基于jdk7的
1,RSA
String password="test";
// 1.初始化密钥
KeyPairGenerator keyPairGenerator = KeyPairGenerator.getInstance("RSA");
keyPairGenerator.initialize(51
- Hibernate学习笔记
caoyong
Hibernate
1>、Hibernate是数据访问层框架,是一个ORM(Object Relation Mapping)框架,作者为:Gavin King
2>、搭建Hibernate的开发环境
a>、添加jar包:
aa>、hibernatte开发包中/lib/required/所
- 设计模式之装饰器模式Decorator(结构型)
漂泊一剑客
Decorator
1. 概述
若你从事过面向对象开发,实现给一个类或对象增加行为,使用继承机制,这是所有面向对象语言的一个基本特性。如果已经存在的一个类缺少某些方法,或者须要给方法添加更多的功能(魅力),你也许会仅仅继承这个类来产生一个新类—这建立在额外的代码上。
- 读取磁盘文件txt,并输入String
一炮送你回车库
String
public static void main(String[] args) throws IOException {
String fileContent = readFileContent("d:/aaa.txt");
System.out.println(fileContent);
- js三级联动下拉框
3213213333332132
三级联动
//三级联动
省/直辖市<select id="province"></select>
市/省直辖<select id="city"></select>
县/区 <select id="area"></select>
- erlang之parse_transform编译选项的应用
616050468
parse_transform游戏服务器属性同步abstract_code
最近使用erlang重构了游戏服务器的所有代码,之前看过C++/lua写的服务器引擎代码,引擎实现了玩家属性自动同步给前端和增量更新玩家数据到数据库的功能,这也是现在很多游戏服务器的优化方向,在引擎层面去解决数据同步和数据持久化,数据发生变化了业务层不需要关心怎么去同步给前端。由于游戏过程中玩家每个业务中玩家数据更改的量其实是很少
- JAVA JSON的解析
darkranger
java
// {
// “Total”:“条数”,
// Code: 1,
//
// “PaymentItems”:[
// {
// “PaymentItemID”:”支款单ID”,
// “PaymentCode”:”支款单编号”,
// “PaymentTime”:”支款日期”,
// ”ContractNo”:”合同号”,
//
- POJ-1273-Drainage Ditches
aijuans
ACM_POJ
POJ-1273-Drainage Ditches
http://poj.org/problem?id=1273
基本的最大流,按LRJ的白书写的
#include<iostream>
#include<cstring>
#include<queue>
using namespace std;
#define INF 0x7fffffff
int ma
- 工作流Activiti5表的命名及含义
atongyeye
工作流Activiti
activiti5 - http://activiti.org/designer/update在线插件安装
activiti5一共23张表
Activiti的表都以ACT_开头。 第二部分是表示表的用途的两个字母标识。 用途也和服务的API对应。
ACT_RE_*: 'RE'表示repository。 这个前缀的表包含了流程定义和流程静态资源 (图片,规则,等等)。
A
- android的广播机制和广播的简单使用
百合不是茶
android广播机制广播的注册
Android广播机制简介 在Android中,有一些操作完成以后,会发送广播,比如说发出一条短信,或打出一个电话,如果某个程序接收了这个广播,就会做相应的处理。这个广播跟我们传统意义中的电台广播有些相似之处。之所以叫做广播,就是因为它只负责“说”而不管你“听不听”,也就是不管你接收方如何处理。另外,广播可以被不只一个应用程序所接收,当然也可能不被任何应
- Spring事务传播行为详解
bijian1013
javaspring事务传播行为
在service类前加上@Transactional,声明这个service所有方法需要事务管理。每一个业务方法开始时都会打开一个事务。
Spring默认情况下会对运行期例外(RunTimeException)进行事务回滚。这
- eidtplus operate
征客丶
eidtplus
开启列模式: Alt+C 鼠标选择 OR Alt+鼠标左键拖动
列模式替换或复制内容(多行):
右键-->格式-->填充所选内容-->选择相应操作
OR
Ctrl+Shift+V(复制多行数据,必须行数一致)
-------------------------------------------------------
- 【Kafka一】Kafka入门
bit1129
kafka
这篇文章来自Spark集成Kafka(http://bit1129.iteye.com/blog/2174765),这里把它单独取出来,作为Kafka的入门吧
下载Kafka
http://mirror.bit.edu.cn/apache/kafka/0.8.1.1/kafka_2.10-0.8.1.1.tgz
2.10表示Scala的版本,而0.8.1.1表示Kafka
- Spring 事务实现机制
BlueSkator
spring代理事务
Spring是以代理的方式实现对事务的管理。我们在Action中所使用的Service对象,其实是代理对象的实例,并不是我们所写的Service对象实例。既然是两个不同的对象,那为什么我们在Action中可以象使用Service对象一样的使用代理对象呢?为了说明问题,假设有个Service类叫AService,它的Spring事务代理类为AProxyService,AService实现了一个接口
- bootstrap源码学习与示例:bootstrap-dropdown(转帖)
BreakingBad
bootstrapdropdown
bootstrap-dropdown组件是个烂东西,我读后的整体感觉。
一个下拉开菜单的设计:
<ul class="nav pull-right">
<li id="fat-menu" class="dropdown">
- 读《研磨设计模式》-代码笔记-中介者模式-Mediator
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
/*
* 中介者模式(Mediator):用一个中介对象来封装一系列的对象交互。
* 中介者使各对象不需要显式地相互引用,从而使其耦合松散,而且可以独立地改变它们之间的交互。
*
* 在我看来,Mediator模式是把多个对象(
- 常用代码记录
chenjunt3
UIExcelJ#
1、单据设置某行或某字段不能修改
//i是行号,"cash"是字段名称
getBillCardPanelWrapper().getBillCardPanel().getBillModel().setCellEditable(i, "cash", false);
//取得单据表体所有项用以上语句做循环就能设置整行了
getBillC
- 搜索引擎与工作流引擎
comsci
算法工作搜索引擎网络应用
最近在公司做和搜索有关的工作,(只是简单的应用开源工具集成到自己的产品中)工作流系统的进一步设计暂时放在一边了,偶然看到谷歌的研究员吴军写的数学之美系列中的搜索引擎与图论这篇文章中的介绍,我发现这样一个关系(仅仅是猜想)
-----搜索引擎和流程引擎的基础--都是图论,至少像在我在JWFD中引擎算法中用到的是自定义的广度优先
- oracle Health Monitor
daizj
oracleHealth Monitor
About Health Monitor
Beginning with Release 11g, Oracle Database includes a framework called Health Monitor for running diagnostic checks on the database.
About Health Monitor Checks
Health M
- JSON字符串转换为对象
dieslrae
javajson
作为前言,首先是要吐槽一下公司的脑残编译部署方式,web和core分开部署本来没什么问题,但是这丫居然不把json的包作为基础包而作为web的包,导致了core端不能使用,而且我们的core是可以当web来用的(不要在意这些细节),所以在core中处理json串就是个问题.没办法,跟编译那帮人也扯不清楚,只有自己写json的解析了.
- C语言学习八结构体,综合应用,学生管理系统
dcj3sjt126com
C语言
实现功能的代码:
# include <stdio.h>
# include <malloc.h>
struct Student
{
int age;
float score;
char name[100];
};
int main(void)
{
int len;
struct Student * pArr;
int i,
- vagrant学习笔记
dcj3sjt126com
vagrant
想了解多主机是如何定义和使用的, 所以又学习了一遍vagrant
1. vagrant virtualbox 下载安装
https://www.vagrantup.com/downloads.html
https://www.virtualbox.org/wiki/Downloads
查看安装在命令行输入vagrant
2.
- 14.性能优化-优化-软件配置优化
frank1234
软件配置性能优化
1.Tomcat线程池
修改tomcat的server.xml文件:
<Connector port="8080" protocol="HTTP/1.1" connectionTimeout="20000" redirectPort="8443" maxThreads="1200" m
- 一个不错的shell 脚本教程 入门级
HarborChung
linuxshell
一个不错的shell 脚本教程 入门级
建立一个脚本 Linux中有好多中不同的shell,但是通常我们使用bash (bourne again shell) 进行shell编程,因为bash是免费的并且很容易使用。所以在本文中笔者所提供的脚本都是使用bash(但是在大多数情况下,这些脚本同样可以在 bash的大姐,bourne shell中运行)。 如同其他语言一样
- Spring4新特性——核心容器的其他改进
jinnianshilongnian
spring动态代理spring4依赖注入
Spring4新特性——泛型限定式依赖注入
Spring4新特性——核心容器的其他改进
Spring4新特性——Web开发的增强
Spring4新特性——集成Bean Validation 1.1(JSR-349)到SpringMVC
Spring4新特性——Groovy Bean定义DSL
Spring4新特性——更好的Java泛型操作API
Spring4新
- Linux设置tomcat开机启动
liuxingguome
tomcatlinux开机自启动
执行命令sudo gedit /etc/init.d/tomcat6
然后把以下英文部分复制过去。(注意第一句#!/bin/sh如果不写,就不是一个shell文件。然后将对应的jdk和tomcat换成你自己的目录就行了。
#!/bin/bash
#
# /etc/rc.d/init.d/tomcat
# init script for tomcat precesses
- 第13章 Ajax进阶(下)
onestopweb
Ajax
index.html
<!DOCTYPE html PUBLIC "-//W3C//DTD XHTML 1.0 Transitional//EN" "http://www.w3.org/TR/xhtml1/DTD/xhtml1-transitional.dtd">
<html xmlns="http://www.w3.org/
- Troubleshooting Crystal Reports off BW
blueoxygen
BO
http://wiki.sdn.sap.com/wiki/display/BOBJ/Troubleshooting+Crystal+Reports+off+BW#TroubleshootingCrystalReportsoffBW-TracingBOE
Quite useful, especially this part:
SAP BW connectivity
For t
- Java开发熟手该当心的11个错误
tomcat_oracle
javajvm多线程单元测试
#1、不在属性文件或XML文件中外化配置属性。比如,没有把批处理使用的线程数设置成可在属性文件中配置。你的批处理程序无论在DEV环境中,还是UAT(用户验收
测试)环境中,都可以顺畅无阻地运行,但是一旦部署在PROD 上,把它作为多线程程序处理更大的数据集时,就会抛出IOException,原因可能是JDBC驱动版本不同,也可能是#2中讨论的问题。如果线程数目 可以在属性文件中配置,那么使它成为
- 正则表达式大全
yang852220741
html编程正则表达式
今天向大家分享正则表达式大全,它可以大提高你的工作效率
正则表达式也可以被当作是一门语言,当你学习一门新的编程语言的时候,他们是一个小的子语言。初看时觉得它没有任何的意义,但是很多时候,你不得不阅读一些教程,或文章来理解这些简单的描述模式。
一、校验数字的表达式
数字:^[0-9]*$
n位的数字:^\d{n}$
至少n位的数字:^\d{n,}$
m-n位的数字:^\d{m,n}$