libsvm--Ubuntu--linux操作指南
我使用的是Ubuntu版本的svm,最重点的求分类面的部分在最下面..
在libsvm-3.23的文件夹路径下打开终端,执行./svm-train heart_scale
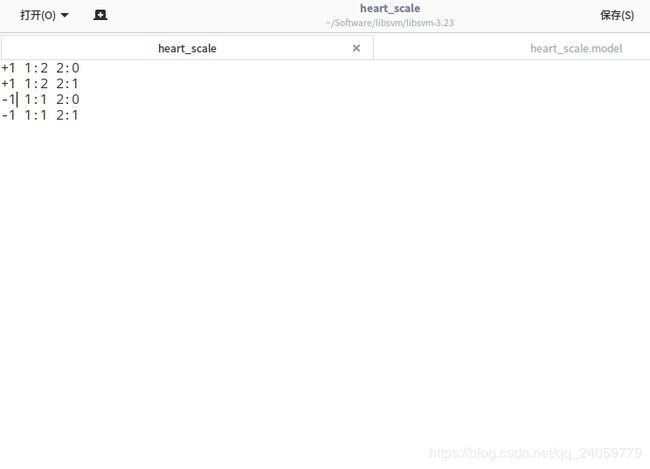
其中heart_scale是训练集,它的元素集合是这样 样本类别 1:样本数据 2:样本数据 (其中1,2代表维度)
经过上面执行会出现一个新的文件: heart_scale.model (训练之后的到的模型,可以作为预测使用)
终端会显示:
代表模型的参数:
#iter代表迭代的次数
nu是你选择核函数类型的参数
obj为SVM文件转换为二次规划求得的最小值
rho为判决函数的偏置项b
nSV为标准支持向量个数(0 nBSV为边界上支持向量的个数(ai=c) total nSV为支持向量的总个数 在heart_scale.model里面的参数: svm_type c_svc % 训练所采用的svm类型,此处为C- SVC kernel_type rbf %训练采用的核函数类型,此处为RBF核 gamma 0.5 %设置核函数中的g ,默认值为1/ k nr_class 2 %分类时的类别数,此处为两分类问题 total_sv 3 %总共的支持向量个数 rho -0.2066442060 %决策函数中的常数项b label 1 -1 %类别标签 nr_sv 2 1 %各类别标签对应的支持向量个数 SV %以下为支持向量 如果想使用easy.py和grid.py更快的执行以上第7步骤的工作。因为Ubuntu中已经安装了python和gunplot,所以这两个软件对安装工作可以省略。进入/home/xxxxxx/libsvm-2.91/tools到目录后,只需要在终端中输入: python easy.py /home/xxxxxx/libsvm-2.91/SYN /home/xxxxxx/libsvm-2.91/SYN2NMF 这里SYN为用来构建模型对训练文件;SYN2NMF为需要预测对文件 scale输入数据,简单优化数据以提高精度。 扫描数据. 因为原始数据可能范围过大或过小, svmscale可以先将数据重新scale (縮放) 到适当范围使训练与预测速度更快 使用方法: ./svm-scale [-l lower] [-u upper] [-y y_lower y_upper] [-s save_name] [-r store_name] filename;其中 -l:数据下限标记; lower:缩放后数据下限 缺省值: lower = -1; -u:数据上限标记; upper:缺省值: upper = 1,代表着没有对y进行缩放; -y:是否对目标值同时进行缩放; y_lower:为下限值; y_upper:为上限值;(回归需要对目标进行缩放,因此该参数可以设定为 –y -1 1 ); -s save_name:表示将缩放的规则保存为文件save_name; -r store_name:表示将缩放规则文件store_name载入后按此缩放; filename:待缩放的数据文件(要求满足前面所述的格式)。 缩放规则文件可以用文本浏览器打开,看到其格式为: y lower upper min max x lower upper index1 min1 max1 index2 min2 max2 其中的lower 与upper 与使用时所设置的lower 与upper 含义相同;index 表 示特征序号;min 转换前该特征的最小值;max 转换前该特征的最大值。 使用方法: ./svm-train [options] training_set_file [model_file] options(操作参数):可用的选项即表示的涵义如下所示 -s svm类型:设置SVM 类型,默认值为0,可选类型有(对于回归只能选3或4): 0 – C-SVC C-支持向量分类机;参数C为惩罚系数,C越大表示对错误分类的惩罚越大,适当的参数C对分类Accuracy很关键 1 – v-SVC v-支持向量分类机;由于C的选取比较困难,用另一个参数v代替C。C是“无意义”的,v是有意义的。(与C_SVC其实采 用的模型相同,但是它们的参数C的范围不同,C_SVC采用的是0到正无穷,该类型是[0,1]。) 2 -- one-class-SVM 单类别-支持向量机,不需要类标号,用于支持向量的密度估计和聚类。 3 – e-SVR ε-支持向量回归机,不敏感损失函数,对样本点来说,存在着一个不为目标函数提供任何损失值的区域。 4 – n-SVR n-支持向量回归机,由于EPSILON_SVR需要事先确定参数,然而在某些情况下选择合适的参数却不是一件容易的事 情.而NU_SVR能够自动计算参数。 -t 核函数类型:设置核函数类型,默认值为2,可选类型有: 0 -- 线性核:u'*v 1 -- 多项式核: (g*u'*v+ coef 0)deg ree 2 -- RBF 核:e( u v 2) g – 3 -- sigmoid 核:tanh(g*u'*v+ coef 0) -d degree:核函数中的degree设置,默认值为3; -g g :设置核函数中的g ,默认值为1/ k ; -r coef0:设置核函数中的coef 0,默认值为0; -c cost:设置C- SVC、e - SVR、n - SVR中从惩罚系数C,默认值为1; -n n :设置n - SVC、one-class-SVM 与n - SVR 中参数n ,默认值0.5; -p e :设置n - SVR的损失函数中的e ,默认值为0.1; -m cachesize:设置cache内存大小,以MB为单位,默认值为40; -e e :设置终止准则中的可容忍偏差,默认值为0.001; -h shrinking:是否使用启发式,可选值为0 或1,默认值为1; -b 概率估计:是否计算SVC或SVR的概率估计,可选值0 或1,默认0; -wi weight:对各类样本的惩罚系数C加权,默认值为1; -v n:n折交叉验证模式。 training_set_file:是要进行训练的数据集; model_file:是训练结束后产生的模型文件,该参数如果不设置将采用默认的文件名,也可以设置成自己惯用的文件名。 用来测试训练结果的准确率 使用方法: ./svm-predict [options] test_file model_file output_file options(操作参数): -b probability_estimates:是否需要进行概率估计预测,可选值为0或1,默认值为0。 model_file: 是由svm-train.exe 产生的模型文件; test_file: 是要进行预测的数据文件; output_file: 是svmpredict 的输出文件,表示预测的结果值。 文件grid.py是对C-SVC的参数c和γ做优选的,原理也是网格遍历,假设我们要对目录libsvm-3.0下的样本文件heart_scale做优选,其具体用法为: 第一步:打开\libsvm-3.0下的tools文件夹,找到grid.py文件,将其拷贝到libsvm-3.0下。用python打开,修改svmtrain_exe和gnuplot_exe的路径。 self.svmtrain_pathname = os.path.join(dirname, '/home/godl/Software/libsvm/libsvm-3.23/svm-train') 第二步:进入grid.py的地方。 第三步:输入以下命令: python grid.py heart_scale 直接看最后一行: 2048.0 0.0001220703125 84.0741 其意义表示:C = 2048.0;γ=0.0001220703125 ;交叉验证精度CV Rate = 84.0741%,这就是最优结果。 第四步:打开目录libsvm-3.0\windows,我们可以看到新生成了两个文件:heart_scale.out和heart_scale.png,第一个文件就是搜索过程中的[local]和最优数据,第二文件就是gnuplot图像。 现在,grid.py已经运行完了,你可以把最优参数输入到svm-train.exe中进行训练了。 训练大的数据集会是非常耗时的事情,在某一些场合上,我们可以先工作在一个更小的一个训练集的子集上实验,这也就是subset.py的用处所在,它的主要功能就是从大的数据中抽取一定数量的数据子集,它的应用格式是如下的: subset.py [options] dataset number [output1] [output2] options: -s method : method of selection (default 0) 0 -- stratified selection (classification only) 1 -- random selection dataset : 数据文件 number: 要选定的subset的个数 output1: the subset(optional) output2: the rest of data(optional) 例如: python subset.py heart_scale 100 file1 file2 From heart_scale 100 samples are randomly selected and stored in file1. All remaining instances are stored in file2. checkdata.py 用于检测样本集存储格式是否正确在控制台下定位到subset.py所在的目录(将其拷贝到C:\libsvm-3.0\windows下)运行: python checkdata.py heart_scale No error. (表示数据文件heart_scale格式没有错误,可以进行后续的调用) 文件easy.py对样本文件做了“一条龙服务”,从参数优选,到文件预测。因此,其对grid.py、svm-train、svm-scal和svm-predict都进行了调用(当然还有必须的python和gnuplot)。因此,运行easy.py需要保证这些文件的路径都要正确。当然还需要样本文件和预测文件,再简单的测试中,可以使用heart_scale作为训练样本,预测文件同样使用heart_scale,只是我们复制一份后将其改名为heart_test,下面说一下使用方法: 第一步:将easy.py拷贝到libsvm-3.0目录下,用python打开(不能双击,而要右键选择“Edit with IDLE”),修改 if not is_win32: 第二步:easy.py的目录打开终端 第三步:输入命令: python easy.py heart_scale heart_test 你就会看到一个gnuplot的动态绘图窗口。大约20s以后停止,dos窗口显示为: Scaling training data... Cross validation... Best c=2048.0, g=0.0001220703125 CV rate=84.0741 Training... Output model: heart_scale.model Scaling testing data... Testing... Accuracy = 85.1852% (230/270) (classification) Output prediction: heart_test.predict 这就是最终预测结果,可以看到第三行就是调用grid.py的结果。在C:\libsvm-3.0\windows下你会看到又多了7个文件,都是以前我们碰到的过程文件,都可以用记事本打开 可以看出模型最后的部分,权重不显示的时候默认权重是1,于是第一个系数就是:1x2+1x2+(-1)x1+(-1)x1=2 第二个系数就是:1x0+1x1+(-1)x0+(-1)x1=0 b的值用rho表示 公式中一般都是用-b 所以如图测试所示的分类面就是 2x1-3=0 即x1=1.5
运行结果:
Best c=2.0, g=0.0078125 CV rate=77.45
Training...
Output model: SYN.model
Scaling testing data...
Testing...
Accuracy = 83.35% (1667/2000) (classification)
Output prediction: SYN2NMF.predict
①./svm-scale
② ./svm-train:训练数据,生成模型
③ ./svm-predict:
grid.py
self.gnuplot_pathname = '/usr/local/bin/gnuplot'
subset.py
checkdata.py
easy.py
svmscale_exe = "/home/godl/Software/libsvm/libsvm-3.23/svm-scale"
svmtrain_exe = "/home/godl/Software/libsvm/libsvm-3.23/svm-train"
svmpredict_exe = "/home/godl/Software/libsvm/libsvm-3.23/svm-predict"
grid_py = "/home/godl/Software/libsvm/libsvm-3.23/tools/grid.py"
gnuplot_exe = "/usr/local/bin/gnuplot"重点来了:分类线到底怎么求呢??
举一个简单的例子,我的输入样本是这样: