在Python中一步一步实现Principal Component Analysis(PCA)
原文链接:http://sebastianraschka.com/Articles/2014_pca_step_by_step.html#drop_labels
引言
主成分分析的主要目的是分析数据以识别模式和查找模式,以最小的信息丢失来降低数据集的维度。
主成分分析的期望结果是将一个特征空间(包括n个d维的样本的数据集)映射到一个较小的子空间上来较好的表示数据。较多的应用是模式分类任务,我们希望通过提取能够描述数据的“最佳”子空间来减少特征空间的维数,以此来减少计算成本和参数估计的误差。
PCA方法总结
下面列出了执行主成分分析的6个一般步骤,我们将在以下部分中进行研究。
- 获取忽略类标签的d维样本的整个数据集;
- 计算d维平均向量(即整个数据集的每个维度的均值);
- 计算整个数据集的协方差矩阵;
- 计算协方差矩阵的特征向量( e1,e2,...,ed )和特征值( λ1,λ2,...,λd )
- 对特征值进行降序排序,并选择前k个特征值对应的特征向量组成一个 d×k 维度的矩阵 W (其中每列表示一个特征向量);
- 使用这个 d×k 的特征向量矩阵将样本映射到新的子空间中。用数学公式表示为 y=WT×x ,其中 x 是一个 d×1 维的向量,代表一个样本, y 是转换后的新的子空间中的 k×1 维的样本。
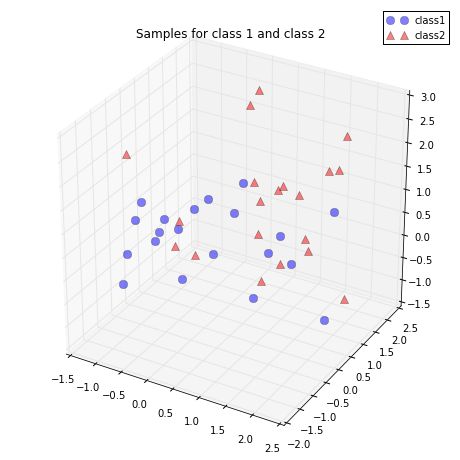
生成一些三维样本数据
对于下面的实例,我们将生成从多元高斯分布随机抽取的40个三维样本。在这里我们假设样本来源于两个不同的类别,其中一半数据集样本(即20个)标记为 ω1 (类别1),另一半标记为 ω2 (类别2)。
为什么选择3维样本
多维数据的问题是其可视化,这将使我们很难按照我们的示例进行主成分分析(至少在视觉上)。我发现从三维数据集开始,我们通过删除1维来减少到二维数据集更直观,更具视觉吸引力。
# get_random_3Dsample.py
import numpy as np
np.random.seed(20170412)
mu_vec1 = np.array([0, 0, 0])
cov_mat1 = np.array([[1,0,0],[0,1,0],[0,0,1]])
class1_sample = np.random.multivariate_normal(mu_vec1, cov_mat1, 20).T
assert class1_sample.shape == (3, 20), "The matrix has not the dimensions 3x20"
mu_vec2 = np.array([1,1,1])
cov_mat2 = np.array([[1,0,0],[0,1,0],[0,0,1]])
class2_sample = np.random.multivariate_normal(mu_vec2, cov_mat2, 20).T
assert class2_sample.shape == (3,20), "The matrix has not the dimensions 3x20"
使用上面的代码生成了两个 3×20 的数据集,分别属于两个类。其中每一列可以表示为一个3维向量
因此,数据集可以表示为
只要知道简单的概念:如何分布类 ω1 和类 ω2 的样本。下面绘制它们的三维散点图:
# plot_3Dscatter.py
#%matplotlib inline
from matplotlib import pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from mpl_toolkits.mplot3d import proj3d
# 导入get_random_3Dsample文件
from get_random_3Dsample import *
fig = plt.figure(figsize=(8,8))
ax = fig.add_subplot(111, projection='3d')
plt.rcParams['legend.fontsize'] = 10
ax.plot(class1_sample[0,:], class1_sample[1,:], class1_sample[2,:], 'o', markersize=8, color='blue', alpha=0.5, label='class1')
ax.plot(class2_sample[0,:], class2_sample[1,:], class2_sample[2,:], '^', markersize=8, alpha=0.5, color='red', label='class2')
plt.title('Samples for class 1 and class 2')
ax.legend(loc='upper right')
plt.show()
PCA算法步骤:
1. 获取忽略类标签的d维样本的整个数据集;
进行主成分分析时不需要类标签,所以融合两类样本为一个 3×40 的数组
all_samples = np.concatenate((class1_sample, class2_sample), axis=1)
assert all_samples.shape == (3,40), "The matrix has not the dimensions 3x40"
2. 计算d维平均向量(即整个数据集的每个维度的均值);
mean_x = np.mean(all_samples[0,:])
mean_y = np.mean(all_samples[1,:])
mean_z = np.mean(all_samples[2,:])
mean_vector = np.array([[mean_x],[mean_y],[mean_z]])
print('Mean Vector:\n', mean_vector)3. 计算整个数据集的协方差矩阵;
通过下面的矩阵来计算散点矩阵
其中 m 是均值向量
scatter_matrix = np.zeros((3,3))
for i in range(all_samples.shape[1]):
scatter_matrix += (all_samples[:,i].reshape(3,1) - mean_vector).dot((all_samples[:,i].reshape(3,1) - mean_vector).T)
print('Scatter Matrix:\n', scatter_matrix)使用内建函数numpy.cov()来计算协方差矩阵。协方差矩阵和散射矩阵的方程非常相似,唯一的区别是我们使用协方差矩阵的缩放因子为 1N−1 ,因此它们的本征空间将是想通的(相同的特征向量,只有特征值被常因子缩放)。
cov_mat = np.cov([all_samples[0,:],all_samples[1,:],all_samples[2,:]])
print('Covariance Matrix:\n', cov_mat)4. 计算协方差矩阵的特征向量和特征值
为了表明从散点矩阵和协方差矩阵导出的特征向量确实是相同的,我们用assert语句来验证。同时可以看到从散点矩阵中求出的特征值确实被因子 39 (即N−1) 缩放
# eigenvectors and eigenvalues for the from the scatter matrix
eig_val_sc, eig_vec_sc = np.linalg.eig(scatter_matrix)
# eigenvectors and eigenvalues for the from the covariance matrix
eig_val_cov, eig_vec_cov = np.linalg.eig(cov_mat)
for i in range(len(eig_val_sc)):
eigvec_sc = eig_vec_sc[:,i].reshape(1,3).T
eigvec_cov = eig_vec_cov[:,i].reshape(1,3).T
assert eigvec_sc.all() == eigvec_cov.all(), 'Eigenvectors are not identical'
print('Eigenvector {}: \n{}'.format(i+1, eigvec_sc))
print('Eigenvalue {} from scatter matrix: {}'.format(i+1, eig_val_sc[i]))
print('Eigenvalue {} from covariance matrix: {}'.format(i+1, eig_val_cov[i]))
print('Scaling factor: ', eig_val_sc[i]/eig_val_cov[i])
print(40 * '-')
验证特征根特征向量的计算
快速验证特征根特征向量的计算是正确的并且满足方程
其中
for i in range(len(eig_val_sc)):
eigv = eig_vec_sc[:,i].reshape(1,3).T
np.testing.assert_array_almost_equal(scatter_matrix.dot(eigv), eig_val_sc[i] * eigv,
decimal=6, err_msg='', verbose=True)
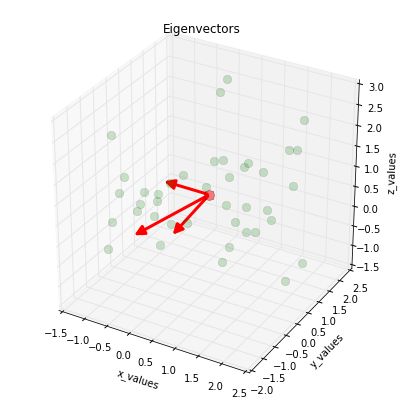
可视化特征向量
%pylab inline
from matplotlib import pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from mpl_toolkits.mplot3d import proj3d
from matplotlib.patches import FancyArrowPatch
class Arrow3D(FancyArrowPatch):
def __init__(self, xs, ys, zs, *args, **kwargs):
FancyArrowPatch.__init__(self, (0,0), (0,0), *args, **kwargs)
self._verts3d = xs, ys, zs
def draw(self, renderer):
xs3d, ys3d, zs3d = self._verts3d
xs, ys, zs = proj3d.proj_transform(xs3d, ys3d, zs3d, renderer.M)
self.set_positions((xs[0],ys[0]),(xs[1],ys[1]))
FancyArrowPatch.draw(self, renderer)
fig = plt.figure(figsize=(7,7))
ax = fig.add_subplot(111, projection='3d')
ax.plot(all_samples[0,:], all_samples[1,:], all_samples[2,:], 'o', markersize=8, color='green', alpha=0.2)
ax.plot([mean_x], [mean_y], [mean_z], 'o', markersize=10, color='red', alpha=0.5)
for v in eig_vec_sc.T:
a = Arrow3D([mean_x, v[0]], [mean_y, v[1]], [mean_z, v[2]], mutation_scale=20, lw=3, arrowstyle="-|>", color="r")
ax.add_artist(a)
ax.set_xlabel('x_values')
ax.set_ylabel('y_values')
ax.set_zlabel('z_values')
plt.title('Eigenvectors')
plt.show()
5. 对特征值进行降序排序,并选择前k个特征值对应的特征向量组成一个 d×k 维度的矩阵 W (其中每列表示一个特征向量);
我们开始的目的是减少我们的特征空间的维度,即通过PCA将特征空间投影到更小的子空间上,其中特征向量将形成这个新特征子空间的轴。然而,特征向量仅定义新轴的方向,因为它们具有所有相同的单位长度1,我们可以通过以下代码确认:
for ev in eig_vec_sc:
numpy.testing.assert_array_almost_equal(1.0, np.linalg.norm(ev))
# instead of 'assert' because of rounding errors因此,为了确定去掉低维子空间中的哪个特征向量,我们必须看一下特征向量对应的特征值。粗略来说,具有最低特征值的特征向量具有关于数据分布的最少信息,并且那些是我们想要去掉的信息。通常的方法是将特征向量从最高到最低对应的特征值进行排序,并选择前k个特征向量。
# Make a list of (eigenvalue, eigenvector) tuples
eig_pairs = [(np.abs(eig_val_sc[i]), eig_vec_sc[:,i]) for i in range(len(eig_val_sc))]
# Sort the (eigenvalue, eigenvector) tuples from high to low
eig_pairs.sort(key=lambda x: x[0], reverse=True)
# Visually confirm that the list is correctly sorted by decreasing eigenvalues
for i in eig_pairs:
print(i[0])对于这个简单的例子,我们将3维特征空间降低到2维特征子空间,并将两个最高特征值对应的特征向量进行组合,构建我们的 d×k 维特征向量矩阵 W 。
matrix_w = np.hstack((eig_pairs[0][1].reshape(3,1), eig_pairs[1][1].reshape(3,1)))
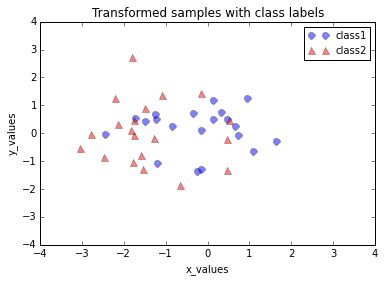
print('Matrix W:\n', matrix_w)6. 使用这个 d×k 的特征向量矩阵将样本映射到新的子空间中。用数学公式表示为 y=WT×x ,其中 x 是一个 d×1 维的向量,代表一个样本, y 是转换后的新的子空间中的 k×1 维的样本。
最后使用刚才计算出来的 2×3 维矩阵 W 进过方程 y=WT×x 转换样本到新的子空间中.
transformed = matrix_w.T.dot(all_samples)
assert transformed.shape == (2,40), "The matrix is not 2x40 dimensional."plt.plot(transformed[0,0:20], transformed[1,0:20], 'o', markersize=7, color='blue', alpha=0.5, label='class1')
plt.plot(transformed[0,20:40], transformed[1,20:40], '^', markersize=7, color='red', alpha=0.5, label='class2')
plt.xlim([-4,4])
plt.ylim([-4,4])
plt.xlabel('x_values')
plt.ylabel('y_values')
plt.legend()
plt.title('Transformed samples with class labels')
plt.show()最终代码<-点我下载O(∩_∩)O
# -*- coding:utf-8 -*-
#%matplotlib inline
from matplotlib import pyplot as plt
import numpy as np
from mpl_toolkits.mplot3d import Axes3D
from mpl_toolkits.mplot3d import proj3d
from matplotlib.patches import FancyArrowPatch
#定义一个画3D箭头的类
class Arrow3D(FancyArrowPatch):
def __init__(self, xs, ys, zs, *args, **kwargs):
FancyArrowPatch.__init__(self, (0,0), (0,0), *args, **kwargs)
self._verts3d = xs, ys, zs
def draw(self, renderer):
xs3d, ys3d, zs3d = self._verts3d
xs, ys, zs = proj3d.proj_transform(xs3d, ys3d, zs3d, renderer.M)
self.set_positions((xs[0],ys[0]),(xs[1],ys[1]))
FancyArrowPatch.draw(self, renderer)
#设置一个随机种子,这里设置的是当前日期
np.random.seed(20170412)
#赋值类别1的均值向量和方差矩阵,并生成类别1的样本
mu_vec1 = np.array([0,0,0])
cov_mat1 = np.array([[1,0,0],[0,1,0],[0,0,1]])
class1_sample = np.random.multivariate_normal(mu_vec1, cov_mat1, 20).T
assert class1_sample.shape == (3,20), "The matrix has not the dimensions 3x20"
#赋值类别2的均值向量和方差矩阵,并生成类别2的样本
mu_vec2 = np.array([1,1,1])
cov_mat2 = np.array([[1,0,0],[0,1,0],[0,0,1]])
class2_sample = np.random.multivariate_normal(mu_vec2, cov_mat2, 20).T
assert class2_sample.shape == (3,20), "The matrix has not the dimensions 3x20"
#画出两个类别对应的图形
fig = plt.figure(1,figsize=(8,8))
ax = fig.add_subplot(111, projection='3d')
plt.rcParams['legend.fontsize'] = 10
ax.plot(class1_sample[0,:], class1_sample[1,:], class1_sample[2,:], 'o', markersize=8, color='blue', alpha=0.5, label='class1')
ax.plot(class2_sample[0,:], class2_sample[1,:], class2_sample[2,:], '^', markersize=8, alpha=0.5, color='red', label='class2')
plt.title('Samples for class 1 and class 2')
ax.legend(loc='upper right')
#将类别1的样本和类别2的样本进行融合
all_samples = np.concatenate((class1_sample, class2_sample), axis=1)
assert all_samples.shape == (3,40), "The matrix has not the dimensions 3x40"
#计算各个维度的均值,并组合成均值向量
mean_x = np.mean(all_samples[0,:])
mean_y = np.mean(all_samples[1,:])
mean_z = np.mean(all_samples[2,:])
mean_vector = np.array([[mean_x],[mean_y],[mean_z]])
print('Mean Vector:\n', mean_vector)
#计算散点矩阵
scatter_matrix = np.zeros((3,3))
for i in range(all_samples.shape[1]):
scatter_matrix += (all_samples[:,i].reshape(3,1) - mean_vector).dot((all_samples[:,i].reshape(3,1) - mean_vector).T)
print('Scatter Matrix:\n', scatter_matrix)
#计算协方差矩阵
cov_mat = np.cov([all_samples[0,:],all_samples[1,:],all_samples[2,:]])
print('Covariance Matrix:\n', cov_mat)
#计算散点矩阵的特征根特征向量
eig_val_sc, eig_vec_sc = np.linalg.eig(scatter_matrix)
#计算协方差矩阵的特征根特征向量
eig_val_cov, eig_vec_cov = np.linalg.eig(cov_mat)
#比较散点矩阵和协方差矩阵特征根特征向量关系
for i in range(len(eig_val_sc)):
eigvec_sc = eig_vec_sc[:,i].reshape(1,3).T
eigvec_cov = eig_vec_cov[:,i].reshape(1,3).T
assert eigvec_sc.all() == eigvec_cov.all(), 'Eigenvectors are not identical'
print('Eigenvector {}: \n{}'.format(i+1, eigvec_sc))
print('Eigenvalue {} from scatter matrix: {}'.format(i+1, eig_val_sc[i]))
print('Eigenvalue {} from covariance matrix: {}'.format(i+1, eig_val_cov[i]))
print('Scaling factor: ', eig_val_sc[i]/eig_val_cov[i])
print(40 * '-')
#验证特征根特征向量计算是否正确
for i in range(len(eig_val_sc)):
eigv = eig_vec_sc[:,i].reshape(1,3).T
np.testing.assert_array_almost_equal(scatter_matrix.dot(eigv), eig_val_sc[i] * eigv,
decimal=6, err_msg='', verbose=True)
#画出特征向量图
fig2 = plt.figure(2,figsize=(7,7))
ax = fig2.add_subplot(111, projection='3d')
ax.plot(all_samples[0,:], all_samples[1,:], all_samples[2,:], 'o', markersize=8, color='green', alpha=0.2)
ax.plot([mean_x], [mean_y], [mean_z], 'o', markersize=10, color='red', alpha=0.5)
for v in eig_vec_sc.T:
a = Arrow3D([mean_x, v[0]], [mean_y, v[1]], [mean_z, v[2]], mutation_scale=20, lw=3, arrowstyle="-|>", color="r")
ax.add_artist(a)
ax.set_xlabel('x_values')
ax.set_ylabel('y_values')
ax.set_zlabel('z_values')
plt.title('Eigenvectors')
#对协方差矩阵的特征根进行排序
for ev in eig_vec_sc:
np.testing.assert_array_almost_equal(1.0, np.linalg.norm(ev))
eig_pairs = [(np.abs(eig_val_sc[i]), eig_vec_sc[:,i]) for i in range(len(eig_val_sc))]
eig_pairs.sort(key=lambda x: x[0], reverse=True)
for i in eig_pairs:
print(i[0])
#选出排序前2个特征根对应的特征向量组合成映射矩阵
matrix_w = np.hstack((eig_pairs[0][1].reshape(3,1), eig_pairs[1][1].reshape(3,1)))
print('Matrix W:\n', matrix_w)
#计算映射后的子空间
transformed = matrix_w.T.dot(all_samples)
assert transformed.shape == (2,40), "The matrix is not 2x40 dimensional."
#画出映射后的样本
plt.figure(3, figsize=(7,7))
plt.plot(transformed[0,0:20], transformed[1,0:20], 'o', markersize=7, color='blue', alpha=0.5, label='class1')
plt.plot(transformed[0,20:40], transformed[1,20:40], '^', markersize=7, color='red', alpha=0.5, label='class2')
plt.xlim([-4,4])
plt.ylim([-4,4])
plt.xlabel('x_values')
plt.ylabel('y_values')
plt.legend()
plt.title('Transformed samples with class labels')
plt.show()