机器学习之聚类(Clustering,包含k-means,DBSCAN等算法)
聚类(Clustering)
前言 : 总结了许多聚类算法相关内容,并使用python自编程实现 kmeans, DBSCAN算法.待续包括:层次聚类算法,FCM算法,高斯混合聚类,还有性能度量.
参考书籍:<机器学习>,<数据挖掘概念与技术>
文章目录
- 聚类(Clustering)
- 0. 基本内容
- 1. 关于主要思想(聚类任务)
- 2. 关于聚类规则(聚类方法)
- 2.1 划分方法(partitioning method)
- 2.2 层次方法(hierarchical method)
- 2.3 基于密度(density-based method)
- 2.4 基于网格(grid-based method)
- 3. 聚类算法
- 3.1 k-means 算法
- 算法步骤
- 编程实现
- 结果
- 心得
- 优缺点
- 3.2 模糊C聚类(Fuzzy C-means cluster, FCM)
- 3.3 高斯混合聚类
- 3.4 具有噪声应用的基于密度的空间聚类(Density-Based Spatial Clustering of Applications with Noise, DBSCAN)
- 思想
- 算法步骤
- 编程实现
- 结果
- 心得
- 3.5 层次聚类
- 4. 关于结果评测(性能度量)
0. 基本内容
- 无监督学习
- 物以类聚:簇内相似度(intra-cluster similarity)和簇间相似度(inter-cluster similarity)
- 距离度量(distance measure):闵可夫斯基距离
- 性能度量/有效性指标(validity index):外部指标(external index)和内部指标(internal index)
- 外部指标:Jaccard系数(Jaccard coefficient, JC) ; FM指数(Fowlkes and Mallows Index, FMI) ; Rand指数(Rand Index, RI)
- 内部指标:DB指数(Davies-Bouldin Index, DBI) ; Dunn指数(Dunn Index, DI)
1. 关于主要思想(聚类任务)
聚类的基本思想比较简单,就是把看着像同类的数据划分到一块。
术语点说就是,将规则相似的数据聚集在一起,这些数据预先没有分类,即没有标签,最终将一些相似数据聚集到一块,产生了数据间的区分,但仍不能将其称作为每个具体的实际意义上的类,最终聚类的结果只是用于区分或者说是标注。所以聚类是一种非监督(no label)学习,解决的是标注问题。在聚类中被划分后每一块称作为一个簇(cluster)
可以根据一些示意图很好明白聚类的意图和思想
显而易见的是,相同的数据,使用不同的聚类规则会产生不同的结果。 聚类结果没有正误之分,只有对解决问题的适宜好坏之分。
对于聚类分析,有两点比较重要:
- 能够处理任意形状的数据
- 能够较好的处理噪声数据,离群点
对于聚类分析,有两个基本问题:
- 如何选择聚类规则(算法)
- 如何评价聚类结果(评测)
2. 关于聚类规则(聚类方法)
2.1 划分方法(partitioning method)
n 个对象集合划分为 k 簇,通常划分方法是基于距离,其主体思想类似与 LDA ,即使得划分后同簇对象间尽可能靠近,异簇对象间尽可能远离。如果要达到全局最优,要穷举遍历所有可能划分,计算量巨大,因此,通常使用逼近最优解的方法进行迭代(EM算法的意味渐浓)。这种方法通常适用于小规模球形数据结构的簇。
比较代表性的算法是 k-mens 算法
2.2 层次方法(hierarchical method)
2.3 基于密度(density-based method)
2.4 基于网格(grid-based method)
3. 聚类算法
3.1 k-means 算法
算法步骤
参考<机器学习>书本
编程实现
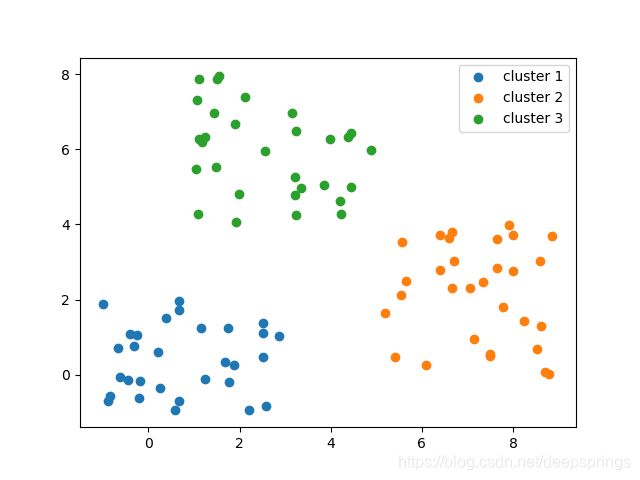
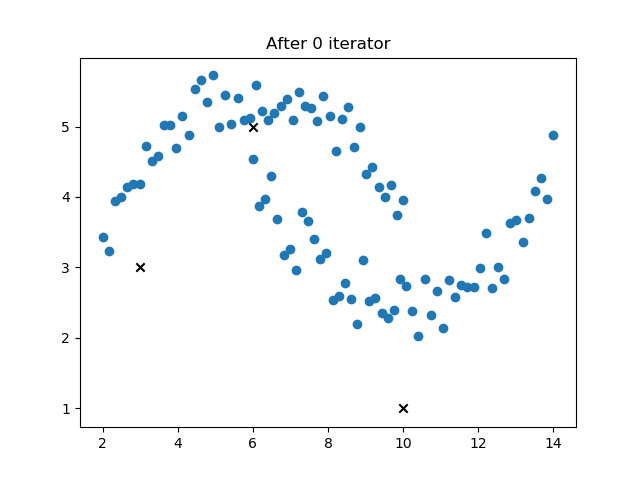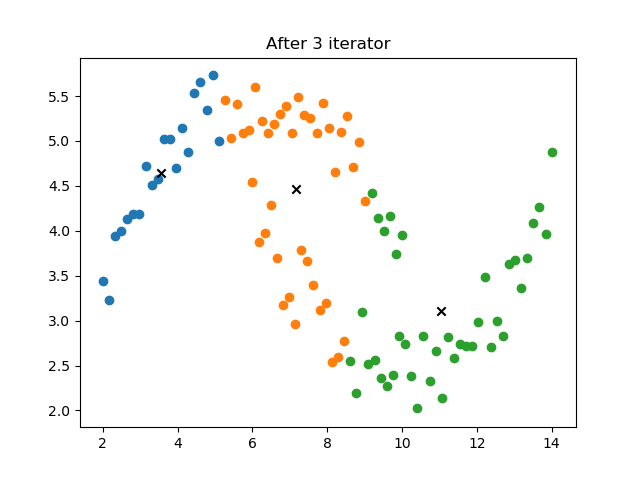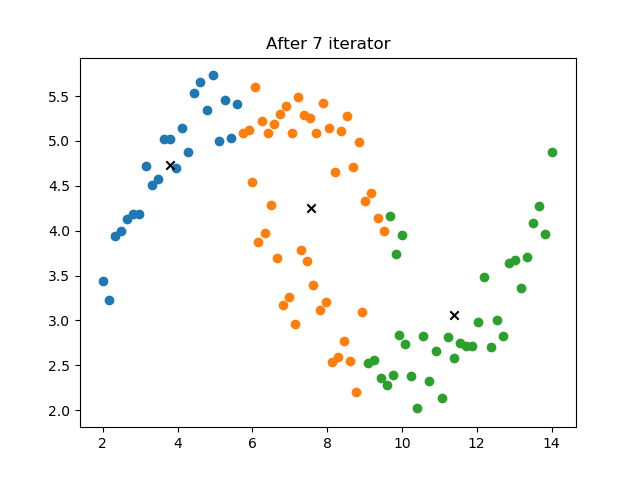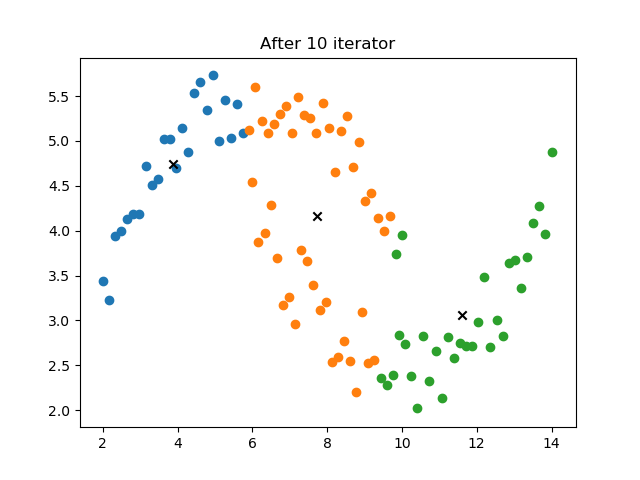
对上述第一个图中数据,实现 k-means 算法
# 实现输入原始数据data:list和初始聚类点数据kdata:list,可视化k-means算法过程
import numpy as np
import matplotlib.pyplot as plt
import imageio
class kmeans:
def __init__(self, data, kdata):
self.data = data # 原始数据
self.kdata = kdata # 聚类中心
self.k = len(self.kdata) # 聚类数
self.it = 0 # 迭代次数
self.clusterIni(0)
def clusterIni(self, sig):
self.cluster = {} # 聚类
for i in range(self.k):
if i==0 and sig==0:
self.cluster[i] = data
else:
self.cluster[i] = []
# 迭代
def iter(self):
# 聚类
self.clusterIni(1)
for _i,i in enumerate(self.data):
index = 0
distance = 1.0
for _j,j in enumerate(self.kdata):
dis = (i[0]-j[0])**2 + (i[1]-j[1])**2
if _j == 0:
distance = dis
index = 0
elif dis <= distance:
distance = dis
index = _j
self.cluster[index].append(i)
# 计算新聚类中心
count = 0
for c in self.cluster:
sx = 0
sy = 0
for d in self.cluster[c]:
sx = sx + d[0]
sy = sy + d[1]
avgx = sx / len(self.cluster[c])
avgy = sy / len(self.cluster[c])
if avgx == self.kdata[c][0] and avgy == self.kdata[c][1]:
count = count + 1
else:
self.kdata[c] = [avgx,avgy]
self.it = self.it + 1
if(count == len(self.kdata)):
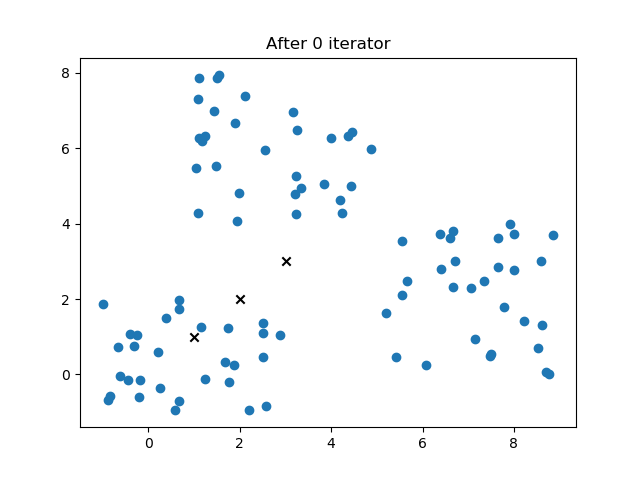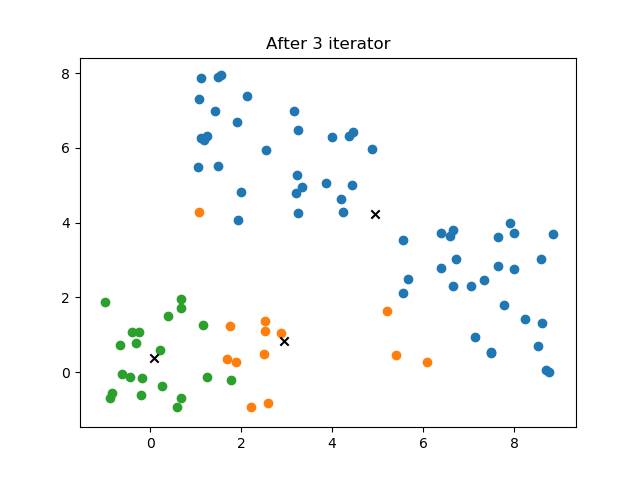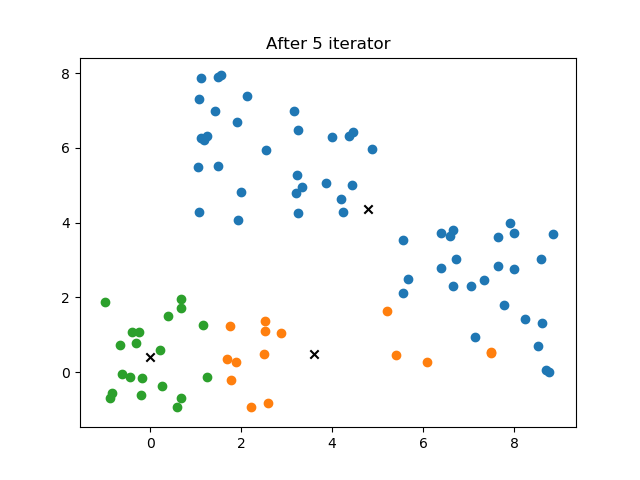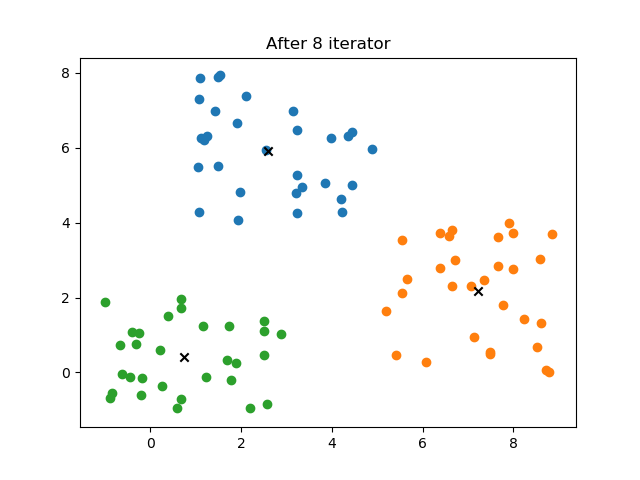
print("总迭代次数:%d"%(self.it-1))
return 0
else:
return 1
# 根据cluster可视化
def plot(self, isSave = 0):
# 显示簇
for c in self.cluster:
if(self.cluster[c] == []):
continue
x = np.array(self.cluster[c])[:,0]
y = np.array(self.cluster[c])[:,1]
plt.scatter(x, y)
# 显示中心点
mx = np.array(self.kdata)[:,0]
my = np.array(self.kdata)[:,1]
plt.scatter(mx, my, marker='x', color='black')
plt.title("After %d iterator"%self.it)
if isSave:
plt.savefig("./cluster/%d.png"%self.it)
plt.show()
if __name__ == '__main__':
# 原始数据data
data = []
f = open('./clusterData.txt', 'r')
for _d in f:
dat = _d.rstrip().split(' ')
data.append([float(dat[0]), float(dat[1])])
f.close()
# 初始聚类中心点kdata
kdata = [[3,3],[2,2],[1,1]]
obj = kmeans(data, kdata)
# 迭代10次
for i in range(10):
obj.plot(1)
b = obj.iter()
# 或到稳定时结束
if not b:
print('迭代结束!')
break
#obj.plot(1)
#obj.plot()
# 合成成gif动画
inp = []
for i in range(obj.it):
inp.append(imageio.imread('./cluster/%d.png'%i))
outp = './cluster/cluster.gif'
imageio.mimsave(outp, inp, duration=1)
结果
心得
-
初始点选择一般是原始数据中存在的点,不然上面自编的k-means可能会出问题(不知官方包怎么样),因为可能出现分母为0情况
-
收敛速度确实很快,通常几步就可以得到最终结果
-
初始簇数的选择问题:k 的选择,即分簇的个数有时候难以预见
-
初始点的选择问题:初始中心点选择影响到最终结果,且最终是收敛于局部最优
-
适应的数据类型:只适用与球形数据,如上面的第二类数据就不适用
优缺点
- 思想简单,实现简单,收敛很快,时间复杂度 O ( t ⋅ k ⋅ n ) O(t\cdot k\cdot n) O(t⋅k⋅n)
- k 值选择很麻烦,且是局部收敛
- 因为是基于均值,所以i对噪点十分敏感
- 只适应于球形数据
实践中,为了得到好的结果,通常使用不同的初始簇,多次运行 k-means 算法
3.2 模糊C聚类(Fuzzy C-means cluster, FCM)
另见:https://blog.csdn.net/deepsprings/article/details/106820626
3.3 高斯混合聚类
3.4 具有噪声应用的基于密度的空间聚类(Density-Based Spatial Clustering of Applications with Noise, DBSCAN)
思想
该种聚类的思想也极其简单,其比较符合人眼判别结果。即,数据点密集的认为更可能是同一类。而为了表征这种解决问题的思路,使用了一些术语:
- ϵ \epsilon ϵ 邻域:与点距离不大于 ϵ \epsilon ϵ 的样本集。可以通过指定距离和符合条件的样本点集的数目,而这正是体现了密度的方面。这两个参数也是DBSCAN算法调参参数。
- 核心对象:某点临域内的样本点数大于指定值,那么该点就是核心对象。
- 密度直达:两个核心对象点临域相互包含对方。
- 密度可达:两个点相距较远,但可以通过多个密度直达点相连。
- 密度相连:密度可达点可以相连通,实现给定参数下同簇间的连通性。
在为一簇的情况下,结果有点类似于区域增长算法。但区域增长仅考虑了距离因素,而没有密度的因素。
算法步骤
参考<机器学习>书本
编程实现
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import queue
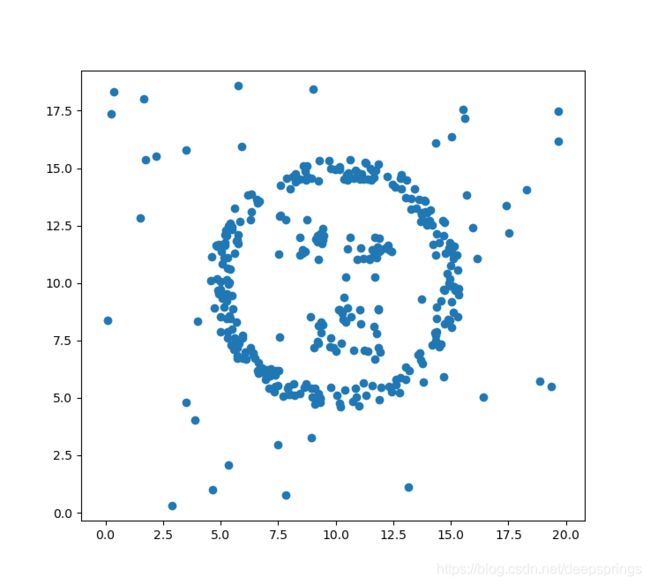
# 原始数据生成
def genData():
theta = np.random.uniform(0, 2*np.pi, 250)
r = np.random.uniform(4.5, 5.5, 250)
x1 = r*np.sin(theta) + 5
y1 = r*np.cos(theta) + 5
x2 = np.random.uniform(1, 3, 20) + 1.5
y2 = np.random.uniform(1, 3, 20) + 5
x3 = np.random.uniform(1, 3, 20) + 4.5
y3 = np.random.uniform(1, 2, 20) + 5
x4 = np.random.uniform(4, 7, 30)
y4 = np.random.uniform(2, 4, 30)
x_ = np.random.uniform(0, 20, 50) - 5
y_ = np.random.uniform(0, 20, 50) - 5
x = np.hstack((x1, x2, x3, x4, x_)) + 5
y = np.hstack((y1, y2, y3, y4, y_)) + 5
plt.scatter(x, y)
plt.xlim((0, 15))
plt.xlim((0, 15))
plt.axis('equal')
plt.show()
dataF = pd.DataFrame({'x':x, 'y':y})
dataF.to_csv('./data.csv')
# 读取原始数据集
def getData():
data_0 = pd.read_csv('./data.csv')
dataA = np.array(data_0)[:,1:]
dataL = dataA.tolist()
# plt.scatter(dataA[:,0], dataA[:,1])
# plt.show()
return dataL
# 自编程实现
class DBSCAN:
def __init__(self, data, eps, min_samples):
self.data = data # 原始数据
self.eps = eps
self.minP = min_samples - 1
self.core = []
self.k = 0
self.cluster = {} # 聚类结果
# 得到某点 d<=r 的邻域点, data数据集,isCont是否包含自身
def getN(self, point, r, isCont = 0):
data = self.data[:]
data.remove(point)
Nei = []
for i in data:
dis = (i[0]-point[0])**2 + (i[1]-point[1])**2
if dis <= r**2:
Nei.append(i)
if isCont:
Nei.append(point)
return Nei
# 获取两点集的交集
def intersection(self, ptL1, ptL2):
inters = []
for i in ptL1:
if i in ptL2:
inters.append(i)
return inters
# 获取核心对象, isPlot是否可视化
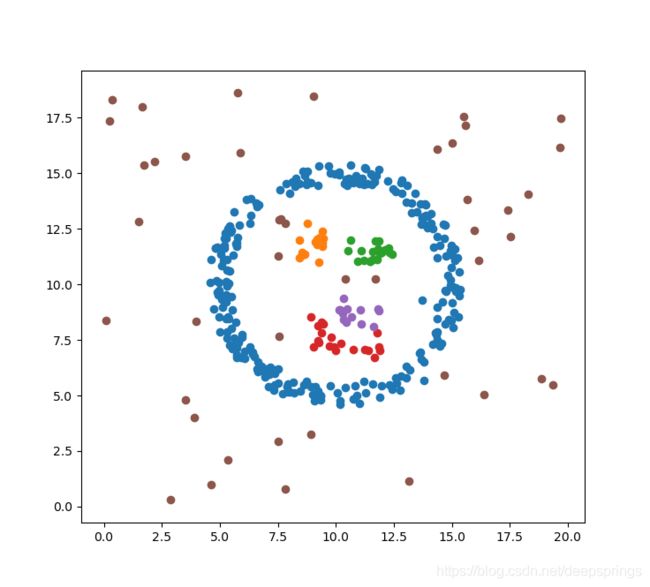
def getCore(self, isPlot = 0):
data = self.data[:]
for i in data:
temp = self.getN(i, self.eps)
if len(temp) >= self.minP:
self.core.append(i)
if isPlot:
x = np.array(self.core)[:,0]
y = np.array(self.core)[:,1]
plt.scatter(np.array(self.data)[:,0], np.array(self.data)[:,1])
plt.scatter(x, y)
plt.legend(('rawPoint','corePoint'))
plt.show()
# 算法迭代
def dbscan(self):
q = queue.Queue()
core = self.core[:]
Data = self.data[:]
while len(core) != 0:
dat = Data[:]
q.put(core[0])
Data.remove(core[0])
while not q.empty():
pt = q.get()
Nei = self.getN(pt, self.eps)
if len(Nei) >= self.minP:
delta = self.intersection(Nei, Data)
for i in delta:
q.put(i)
Data.remove(i)
self.k = self.k + 1
self.cluster[self.k] = dat[:]
for j in Data:
self.cluster[self.k].remove(j)
for k in self.cluster[self.k]:
try:
core.remove(k)
except:
continue
self.cluster[0] = Data # key = 0,对应于噪点
def plot(self):
for i in self.cluster:
x = np.array(self.cluster[i])[:,0]
y = np.array(self.cluster[i])[:,1]
plt.scatter(x, y)
plt.show()
# 测试
if __name__ == '__main__':
genData()
dataL = getData()
db = DBSCAN(dataL, 0.8, 5)
db.getCore()
db.dbscan()
db.plot()
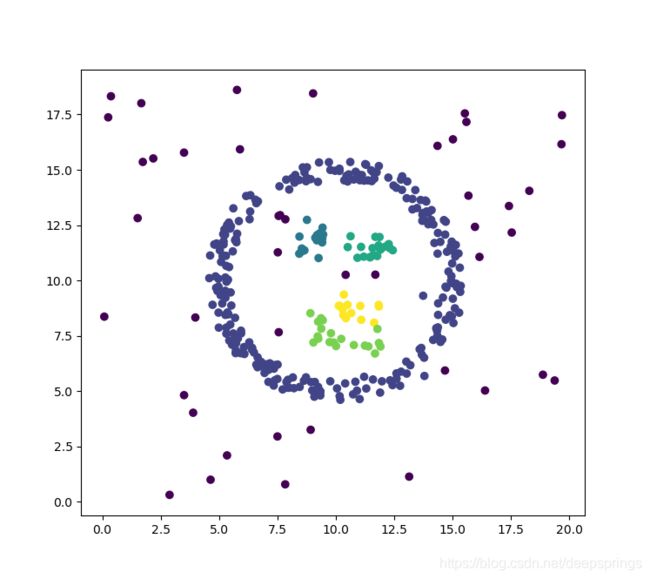
# 使用官方库
import sklearn.cluster as skc
dataA = np.array(dataL)
y_pred = skc.DBSCAN(eps = 0.8, min_samples = 5).fit_predict(dataA)
plt.scatter(dataA[:, 0], dataA[:, 1], c=y_pred)
plt.show()
结果
原始数据
官方库
心得
- 可以适用于各种形状,可以处理噪点离群点,不必烦恼k-means 的k值
- 参数 ϵ \epsilon ϵ 和 m i n P t s minPts minPts 选择尝试也并不简单
3.5 层次聚类
4. 关于结果评测(性能度量)
一种显而易见的方法是类似与 LDA 算法中同类间距分布的度量,即通过查看簇内数据散布情况,
J e = ∑ i = 1 n ∑ x ∈ D i ∣ ∣ x − m i ∣ ∣ 2 h e r e , m i = 1 n i ∑ x ∈ D i x J_e = \sum\limits_{i=1}^n\sum\limits_{x\in D_i}||x-m_i||^2 \\ here,\ m_i=\frac{1}{n_i}\sum_{x\in D_i}x Je=i=1∑nx∈Di∑∣∣x−mi∣∣2here, mi=ni1x∈Di∑x