机器学习 | 吴恩达机器学习第八周编程作业(Python版)
实验指导书 下载密码:963j
本篇博客主要讲解,吴恩达机器学习第八周的编程作业,主要包含KMeans实验和PCA实验两部分。原始实验使用Matlab实现,本篇博客提供Python版本。
目录
1.实验包含的文件
2.KMeans实验
3.K-means实验完整代码
4.PCA实验
5.PCA实验完整代码
1.实验包含的文件
| 文件名称 | 含义 |
| ex7.py | K-means实验主程序 |
| ex7_pca.py | PCA实验主程序 |
| ex7data1.mat | PCA实验数据集 |
| ex7data2.mat | K-means实验数据集 |
| ex7faces.mat | 人脸数据集 |
| bird_small.png | 示例图片 |
| displayData.py | 可视化数据 |
| runkMeans.py | 运行K-means算法 |
| pca.py | 运行PCA |
| projectData.py | 将原始数据映射到低维空间 |
| recoverData.py | 将压缩数据恢复到原始数据 |
| findClosestCentroids.py | 找到最近的簇 |
| computeCentroids.py | 更新聚类中心 |
| kMeansInitCentroids.py | 初始化k-means的初始聚类中心 |
完成红色部分程序的关键代码。
2.KMeans实验
- 打开KMeans实验主程序ex7.py
'''第1部分 为每个样本点找到离他最近的聚类中心'''
print('Finding closest centroids.')
data = scio.loadmat('ex7data2.mat') #加载矩阵格式的数据
X = data['X'] #提取输入特征矩阵
k = 3 # 随机初始化3个聚类中心
initial_centroids = np.array([[3, 3], [6, 2], [8, 5]])
#找到离每个样本最近的初始聚类中心序号
idx = fc.find_closest_centroids(X, initial_centroids)
print('Closest centroids for the first 3 examples: ')
print('{}'.format(idx[0:3]))
print('(the closest centroids should be 0, 2, 1 respectively)')- 编写findClosestCentroids.py 簇分配
def find_closest_centroids(X, centroids):
K = centroids.shape[0] #聚类中心数量
m = X.shape[0] #样本数
idx = np.zeros(m) #存储m个样本对应的最近的聚类中心序号
for i in range(m):
a=(X[i]-centroids).dot((X[i]-centroids).T) #得到一个方阵 对角线上的元素为该样本点到每个聚类中心的距离
idx[i]=np.argsort(a.diagonal())[0] #取出对角线元素 对其索引进行排序 返回离该样本最近的聚类中心的序号
return idx验证正确性:
- 更新聚类中心
'''第2部分 更新聚类中心'''
print('Computing centroids means.')
centroids = cc.compute_centroids(X, idx, k) #在簇分配结束后 对每个簇的样本点重新计算聚类中心
print('Centroids computed after initial finding of closest centroids: \n{}'.format(centroids))
print('the centroids should be')
print('[[ 2.428301 3.157924 ]')
print(' [ 5.813503 2.633656 ]')
print(' [ 7.119387 3.616684 ]]')- 编写computeCentroids.py
def compute_centroids(X, idx, K):
(m, n) = X.shape #m为样本数 n为每个样本的特征数
centroids = np.zeros((K, n)) #存储新的聚类中心的位置
for i in range(K):
centroids[i]=np.mean(X[idx==i],axis=0) #对每个簇 计算新的聚类中心 axis=0对每一列求均值
return centroids验证正确性:
- 运行k-means算法
'''第3部分 运行k-means聚类算法'''
print('Running K-Means Clustering on example dataset.')
#加载数据集
data = scio.loadmat('ex7data2.mat')
X = data['X']
K = 3 #聚类中心数量
max_iters = 10 #设置外循环迭代次数
initial_centroids = np.array([[3, 3], [6, 2], [8, 5]]) #初始化聚类中心
centroids, idx = km.run_kmeans(X, initial_centroids, max_iters, True) #运行k-means算法 返回最终聚类中心位置即每个样本点所属的聚类中心
#并把中间过程以及最终聚类效果可视化
print('K-Means Done.')- 查看runKMeans.py
def run_kmeans(X, initial_centroids, max_iters, plot): #plot设置是否进行可视化
if plot:
plt.figure()
(m, n) = X.shape #m样本数 n样本特征数
K = initial_centroids.shape[0] #聚类中心数量
centroids = initial_centroids
previous_centroids = centroids
idx = np.zeros(m) #存放每个样本所属的聚类中心序号
# 运行k-means
for i in range(max_iters): #外循环
print('K-Means iteration {}/{}'.format((i + 1), max_iters))
idx = fc.find_closest_centroids(X, centroids) #第一个内循环 为每个样本找到最近的聚类中心
if plot:
plot_progress(X, centroids, previous_centroids, idx, K, i) #画出此时簇分配的状态
previous_centroids = centroids
input('Press ENTER to continue')
centroids = cc.compute_centroids(X, idx, K) #第2个内循环 更新聚类中心
return centroids, idx #返回最终聚类中心的位置 和每个样本所属的聚类中心序号
def plot_progress(X, centroids, previous, idx, K, i):
plt.scatter(X[:, 0], X[:, 1], c=idx, s=15) #不同聚类中心用不同的颜色表示
plt.scatter(centroids[:, 0], centroids[:, 1], marker='x', c='black', s=25) #标出聚类中心
for j in range(centroids.shape[0]): #为更新后的聚类中心和之前的聚类中心连线
draw_line(centroids[j], previous[j])
plt.title('Iteration number {}'.format(i + 1))
def draw_line(p1, p2):
plt.plot(np.array([p1[0], p2[0]]), np.array([p1[1], p2[1]]), c='black', linewidth=1)
- 最终的聚类效果和聚类中心的移动过程
- 使用k-means压缩图片
'''第4部分 运行k-means聚类算法 压缩图片'''
print('Running K-Means clustering on pixels from an image')
#加载图片
image = io.imread('bird_small.png')
image = img_as_float(image)
# 图片大小
img_shape = image.shape
X = image.reshape(img_shape[0] * img_shape[1], 3) #把图片转换成3个列向量构成的矩阵 每个列向量代表每个颜色通道的所有像素点
#可以设置不同的参数 观察效果
K = 16 #聚类中心数量
max_iters = 10 #外循环迭代次数
#初始化聚类中心位置很重要 初始化不同 最终聚类效果也会不同
initial_centroids = kmic.kmeans_init_centroids(X, K)
# 运行k-means
centroids, idx = km.run_kmeans(X, initial_centroids, max_iters, False) #False不进行可视化
print('K-Means Done.')
input('Program paused. Press ENTER to continue')
print('Applying K-Means to compress an image.')
# 得到最终聚类结束后 每个样本所属的聚类中心序号
idx = fc.find_closest_centroids(X, centroids)
#用idx做索引
idx=idx.astype(int) #将数值类型转换为整型
idx=idx.tolist() #将数组转换为列表
X_recovered = centroids[idx] #将每个样本点位置转换为它所属簇的聚类中心的位置 实现压缩
X_recovered = np.reshape(X_recovered, (img_shape[0], img_shape[1], 3)) #把图像转换为之前的维度
io.imsave('compress.png',X_recovered) #保存压缩后的图片文件
plt.subplot(2, 1, 1) #可视化原始图片
plt.imshow(image)
plt.title('Original')
plt.subplot(2, 1, 2) #压缩后的图片
plt.imshow(X_recovered)
plt.title('Compressed, with {} colors'.format(K))
input('ex7 Finished. Press ENTER to exit')
- 编写kMeansInitCentroids.py
def kmeans_init_centroids(X, K):
#随机初始化聚类中心
centroids = np.zeros((K, X.shape[1]))
#初始化聚类中心为数据集中的样本点
centroids=X[np.random.randint(0,X.shape[0],K)]
return centroids- 图片压缩效果
3.K-means实验完整代码
下载链接 下载密码:rqk6
4.PCA实验
- 打开PCA实验主程序ex7_pca.py
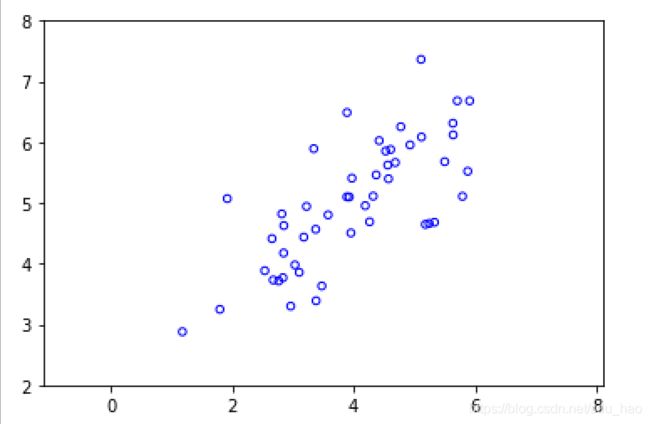
'''第1部分 加载数据集 并可视化'''
#小数据集方便可视化
print('Visualizing example dataset for PCA.')
data = scio.loadmat('ex7data1.mat')
X = data['X'] #两个特征
# 可视化
plt.figure()
plt.scatter(X[:, 0], X[:, 1], facecolors='none', edgecolors='b', s=20)
plt.axis('equal')
plt.axis([0.5, 6.5, 2, 8])- 可视化效果
- 实现PCA算法
'''第2部分 实现PCA 进行数据压缩'''
print('Running PCA on example dataset.')
# 在PCA之前 要对特征进行缩放
X_norm, mu, sigma = fn.feature_normalize(X)
# 执行PCA 返回U矩阵 和S矩阵
U, S = pca.pca(X_norm)
#对比两个不同的特征向量 U[:,0]更好 投影误差最小 U中的各个特征向量(列)都是正交的 2D->1D 取前1个特征向量 作为Ureduce
rk.draw_line(mu, mu + 1.5 * S[0] * U[:, 0])
rk.draw_line(mu, mu + 1.5 * S[1] * U[:, 1])
print('Top eigenvector: \nU[:, 0] = {}'.format(U[:, 0])) #利用PCA得到的特征向量矩阵Ureduce(降维后子空间的基)
print('You should expect to see [-0.707107 -0.707107]')- 查看特征缩放程序featureNormalize.py
def feature_normalize(X):
mu = np.mean(X, 0) #对特征矩阵每一列求均值
sigma = np.std(X, 0, ddof=1) #特征矩阵每一列求标准差
X_norm = (X - mu) / sigma #特征矩阵每一列的元素减去该列均值 除以该列标准差 得到特征缩放后的矩阵
return X_norm, mu, sigma- 编写pca.py
def pca(X):
(m, n) = X.shape #m 样本数 n特征数
U = np.zeros((n,n)) #U 为n*n的矩阵
S = np.zeros(n) #S也是n*n的对角矩阵 只不过svd返回的是其对角线的非0元素
#计算协方差矩阵
Sigma=(1/m)*(X.T.dot(X))
#对协方差矩阵进行奇异值分解
U,S,V=scipy.linalg.svd(Sigma)
return U, S- 可视化降维后的特征向量(子空间的基向量)
验证程序正确性:
- 得到降维后的样本点并进行压缩重放
'''第3部分 得到降维后的样本点 再进行压缩重放'''
print('Dimension reductino on example dataset.')
# 可视化特征缩放后的数据集
plt.figure()
plt.scatter(X_norm[:, 0], X_norm[:, 1], facecolors='none', edgecolors='b', s=20)
plt.axis('equal')
plt.axis([-4, 3, -4, 3])
# 将2维数据映射到1维
K = 1
Z = pd.project_data(X_norm, U, K)
print('Projection of the first example: {}'.format(Z[0]))
print('(this value should be about 1.481274)')
X_rec = rd.recover_data(Z, U, K) #将降维后的1维数据 转换为2维(在特征向量上的投影点)
print('Approximation of the first example: {}'.format(X_rec[0]))
print('(this value should be about [-1.047419 -1.047419])')
# 画出特征缩放后的样本在特征向量上的投影点 并在2者之间连线
plt.scatter(X_rec[:, 0], X_rec[:, 1], facecolors='none', edgecolors='r', s=20)
for i in range(X_norm.shape[0]):
rk.draw_line(X_norm[i], X_rec[i])- 编写降维程序projectData.py
def project_data(X, U, K): #得到降维后的样本点
Z = np.zeros((X.shape[0], K)) #降维后的特征矩阵 Z:m*K X:m*n
Z=X.dot(U[:,:K])
return Z- 编写压缩重放程序recoverData.py
def recover_data(Z, U, K): #进行压缩重放
X_rec = np.zeros((Z.shape[0], U.shape[0])) #原始样本在特征向量上的投影点 X_rec:m*n Z:m*K U:n*n
X_rec=Z.dot(U[:,:K].T)
return X_rec
- 可视化效果
验证程序正确性:
- 加载并可视化人脸数据
'''第4部分 加载并可视化人脸数据集'''
print('Loading face dataset.')
data = scio.loadmat('ex7faces.mat')
X = data['X'] #得到输入特征矩阵
print(X.shape[1]) #特征为1024维
disp.display_data(X[0:100]) #可视化前100个人脸- 可视化效果
- 可视化人脸数据的特征向量
'''第5部分 可视化人脸数据的特征向量'''
print('Running PCA on face dataset.\n(this might take a minute or two ...)')
X_norm, mu, sigma = fn.feature_normalize(X) #对输入特征矩阵进行特征缩放
#执行PCA算法
U, S = pca.pca(X_norm)
#可视化前36个特征向量(每个向量1024维)
disp.display_data(U[:, 0:36].T)
- 可视化效果
- 对人脸数据进行降维(1024->100)
'''第6部分 对人脸数据进行降维 从1024维降到100维'''
print('Dimension reduction for face dataset.')
K = 100
Z = pd.project_data(X_norm, U, K) #得到降维后的特征矩阵(样本点)
print('The projected data Z has a shape of: {}'.format(Z.shape)) #m*100- 可视化降维后,再压缩重放后的人脸数据与原数据比较
'''第7部分 可视化降维后,再压缩重放的人脸数据和原始数据比较'''
print('Visualizing the projected (reduced dimension) faces.')
K = 100
X_rec = rd.recover_data(Z, U, K) #压缩重放
#可视化原始数据
disp.display_data(X_norm[0:100])
plt.title('Original faces')
plt.axis('equal')
#压缩到100维 再压缩重放后的数据
disp.display_data(X_rec[0:100])
plt.title('Recovered faces')
plt.axis('equal')PCA要求投影误差最小,所以2者应该是差不多的:
- 利用PCA可视化高维数据
PCA可以把高维数据降至低维再进行可视化:
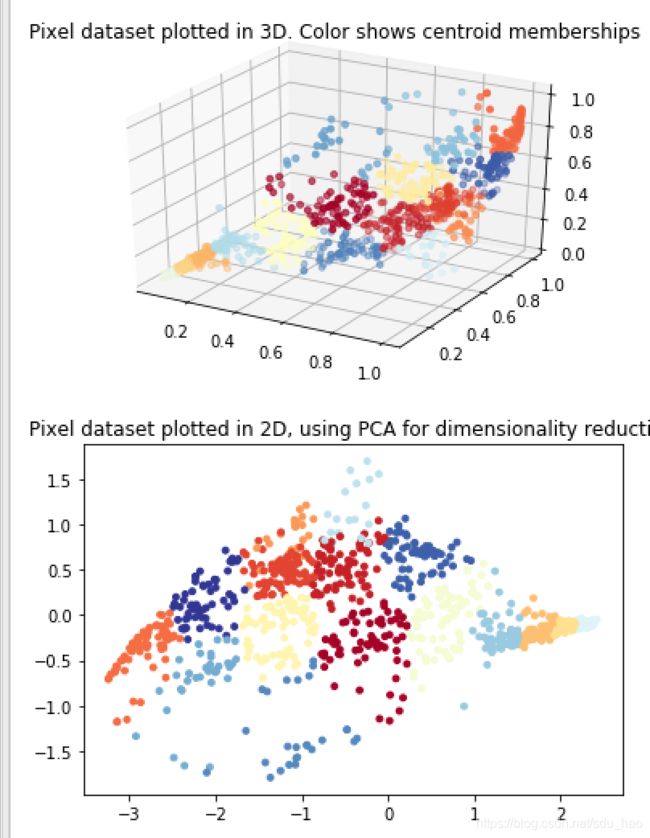
'''第8部分 利用PCA可视化高维数据'''
image = io.imread('bird_small.png') #读取图片
image = img_as_float(image)
img_shape = image.shape
X = image.reshape((img_shape[0] * img_shape[1], 3)) #将图片格式转换为包含3列(3个颜色通道)的矩阵
K = 16 #聚类中心数量
max_iters = 10 #外循环迭代次数
initial_centroids = kmic.kmeans_init_centroids(X, K) #初始化K个聚类中心
centroids, idx = km.run_kmeans(X, initial_centroids, max_iters, False) #执行k-means,得到最终的聚类中心和每个样本点所属的聚类中心序号
selected = np.random.randint(X.shape[0], size=1000) #随机选择1000(可以更改)个样本点 每个样本点3维
#可视化3维数据 不同颜色表示每个样本点的所属的簇
cm = plt.cm.get_cmap('RdYlBu')
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
ax.scatter(X[selected, 0], X[selected, 1], X[selected, 2], c=idx[selected],cmap=cm, s=15, vmin=0, vmax=K)
plt.title('Pixel dataset plotted in 3D. Color shows centroid memberships')
input('Program paused. Press ENTER to continue')
#利用PCA把3维数据 降至2维 进行可视化
X_norm, mu, sigma = fn.feature_normalize(X) #对特征矩阵X 进行特征缩放
#调用pca 3D->2D
U, S = pca.pca(X_norm)
Z = pd.project_data(X_norm, U, 2) #得到降维后的特征矩阵
# 可视化2维数据 不同颜色表示每个样本点的所属的簇
plt.figure()
plt.scatter(Z[selected, 0], Z[selected, 1], c=idx[selected].astype(np.float64), cmap=cm,s=15)
plt.title('Pixel dataset plotted in 2D, using PCA for dimensionality reduction')
- 可视化效果
5.PCA实验完整代码
下载链接