DGL-图属性
1. 图的创建
1.1 同质图
存储图的类:dgl.DGLGraph
1.1.1 直接创建:dgl.graph()
创建图的函数:dgl.graph()
首先创建边:0->1, 0->2, 0->3, 1->3
import torch
u, v = torch.tensor([0,0,0,1]), torch.tensor([1,2,3,3])
在dgl的图中,所有边都是有向的,如果要创建无向图,需要创建双向边
import torch
src, dst = torch.tensor([0,0,0,1]), torch.tensor([1,2,3,3])
u = torch.cat((src,dst))
v = torch.cat((dst,src))
创建图
import dgl
g = dgl.graph((u,v))
除了通过双向边外,还可通过dgl.to_bidirected直接由单向图创建双向图
bg = dgl.to_bidirected(g)
如果在图中有没有边链接的点,需要通过graph函数的num_nodes属性指定有多少单点
# 有4个edge,总共有8个点,则有4个单点
g = dgl.graph((u, v), num_nodes=8)
1.1.3 节点与边的特征
通过ndata与edata可以指定节点与边的特征
"建图"
g = dgl.graph(([0, 0, 1, 5], [1, 2, 2, 0])) # 6 nodes, 4 edges
"指定特征"
g.ndata['x']=torch.ones(g.num_nodes(), 3)
g.ndata['y']=torch.randn(g.num_nodes(),5) # 不同名字可以有不同的特征
g.edata['x']=torch.ones(g.num_edges(),dtype=torch.int32)
"查看特征"
print(g.ndata) # 输出dict {'x':tensor, 'y':tensor}
print(g.edata) # {'x': tensor([1, 1, 1, 1], dtype=torch.int32)}
print(g.ndata['x'][1]) # tensor([1., 1., 1.])
print(g.edata['x'][torch.tensor([0, 3])]) # 查看第0和3号的特征
# tensor([1, 1], dtype=torch.int32)
注意事项:
- 只有数字类型可以做特征(e.g., float, double, and int),特征可以是标量,向量,矩阵或多维张量
- 点特征之间,边特征之间名字要不同,但点特征与边特征之间名字可以相同
- 无法给点/边的子集设置特征,特征tensor的最高维必须等于点/边数
- 特征张量是row-major的,即每一行是一个点/边的特征
对于有权图,可将权值作为图的边特征存储
# edges 0->1, 0->2, 0->3, 1->3
edges = th.tensor([0, 0, 0, 1]), th.tensor([1, 2, 3, 3])
weights = th.tensor([0.1, 0.6, 0.9, 0.7]) # weight of each edge
g = dgl.graph(edges)
g.edata['w'] = weights
print(g)
# Graph(num_nodes=4, num_edges=4,
# ndata_schemes={}
# edata_schemes={'w' : Scheme(shape=(,), dtype=torch.float32)})
1.2 异质图
1.2.1 异质图 Heterogeneous Graphs
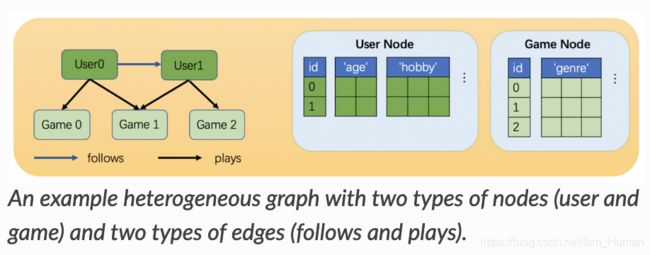
指点/边有不同的类型,每种类型可以有独立的ID及特征,如下图所示,该图有两种类型的节点:User与Game,两种类型有各自的特征,且ID均从0开始
1.2.2 创建异质图-dgl.heterograph()
dgl中,每个关系指定一个图,异质图将由多个关系的图组成
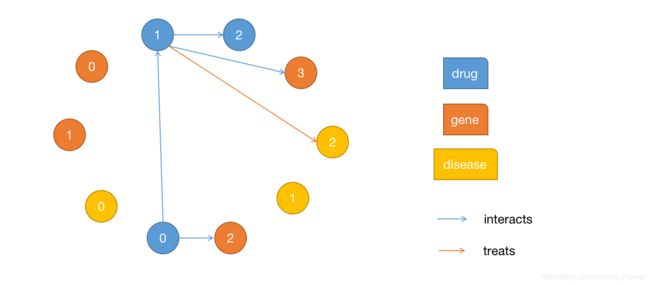
首先,每个关系写成一个三元组(原节点类型,关系类型,目标节点类型),如('drug', 'treats', 'disease')
该关系称为规范边类型(canonical edge types)
接着写出图数据,该数据中每个关系都对应一个图
{
relation1 : (u, v),
relation2 : (u, v),
...}
最后,使用dgl.heterograph()创建异质图
import torch as th
import dgl
# Create a heterograph with 3 node types and 3 edges types.
graph_data = {
('drug', 'interacts', 'drug'): (th.tensor([0, 1]), th.tensor([1, 2])),
('drug', 'interacts', 'gene'): (th.tensor([0, 1]), th.tensor([2, 3])),
('drug', 'treats', 'disease'): (th.tensor([1]), th.tensor([2]))
}
hg = dgl.heterograph(graph_data)
print(hg)
# Graph(num_nodes={'disease': 3, 'drug': 3, 'gene': 4},
# num_edges={('drug', 'interacts', 'drug'): 2, ('drug', 'interacts', 'gene'): 2, ('drug', 'treats', 'disease'): 1},
# metagraph=[('drug', 'drug', 'interacts'), ('drug', 'gene', 'interacts'), ('drug', 'disease', 'treats')])
1.3 Using DGLGraph on a GPU
方法1:构建GPU上的tensor,再传给DGLGraph
>>> u, v = u.to('cuda:0'), v.to('cuda:0')
>>> g = dgl.graph((u, v))
>>> g.device
device(type='cuda', index=0)
方法2:将DGLGraph传到GPU上
>>> u, v = th.tensor([0, 1, 2]), th.tensor([2, 3, 4])
>>> g = dgl.graph((u, v))
>>> g.ndata['x'] = th.randn(5, 3) # original feature is on CPU
>>> g.device
device(type='cpu')
>>> cuda_g = g.to('cuda:0') # accepts any device objects from backend framework
>>> cuda_g.device
device(type='cuda', index=0)
>>> cuda_g.ndata['x'].device # feature data is copied to GPU too
device(type='cuda', index=0)
1.4 从外部资源创建
Scipy sparse matrix 作为图的邻接矩阵 转换成dgl的图 : g = dgl.from_scipy(sp.g)
Networkx graph : g = dgl.from_networkx(nx.g)
import dgl
import scipy.sparse as sp
sp_g = sp.rand(100, 100, density=0.5) # 5% nonzero entries
g = dgl.from_scipy(sp_g)
print(g)
# Graph(num_nodes=100, num_edges=5000,
# ndata_schemes={}
# edata_schemes={})
import networkx as nx
nxg = nx.path_graph(5) # a chain 0-1-2-3-4
# nx.path_graph constructs an undirected NetworkX graph
g = dgl.from_networkx(nxg)
print(g)
# Graph(num_nodes=5, num_edges=8,
# ndata_schemes={}
# edata_schemes={})
nxg = nx.DiGraph([(2, 1), (1, 2), (2, 3), (0, 0)])
# constructs an directed NetworkX graph
g = dgl.from_networkx(nxg)
print(g)
# Graph(num_nodes=4, num_edges=4,
# ndata_schemes={}
# edata_schemes={})
从内存导入图
列举几种常见的存储格式:
从csv文件中加载图: ->pandas -> dgl
- JSON/GML 格式
->Networkx->dgl
- DGL Binary Format
APIs : dgl.save_graphs() dgl.load_graphs()
2 图的属性
创建一个图
u, v = torch.tensor([0,0,0,1,2]), torch.tensor([1,2,3,3,1])
g = dgl.graph((u,v))
g.ndata['n_fe'] = torch.ones((g.num_nodes(), 3))
g.ndata['n_fe_matrix'] = torch.rand((g.num_nodes(), 3, 2))
g.edata['e_fe'] = torch.zeros(g.num_edges(), 5)
查看图信息
print(g)
"""
Graph(num_nodes=4, num_edges=5,
ndata_schemes={'n_fe': Scheme(shape=(3,), dtype=torch.float32),
'n_fe_matrix': Scheme(shape=(3, 2), dtype=torch.float32)}
edata_schemes={'e_fe': Scheme(shape=(5,), dtype=torch.float32)})
"""
2.1 关于图的节点与边的信息
| 同质图 | 异质图 | 功能 | 例 |
|---|---|---|---|
| [’_N’] | hg.ntypes | 节点类型 | [‘disease’, ‘drug’, ‘gene’] |
| [’_E’] | hg.etypes | 边的类型 | [‘interacts’, ‘interacts’, ‘treats’] |
| – | hg.canonical_etypes | 规范边类型(三元组) | [(‘drug’, ‘interacts’, ‘drug’),(‘drug’, ‘interacts’, ‘gene’),(‘drug’, ‘treats’, ‘disease’)] |
| g.num_nodes() | hg.num_nodes(ntype) | 返回图中节点的数量 | 4 |
| g.num_edges() | hg.num_edges(etype) | 返回图中边的数量 | 5 |
| g.num_src_nodes() | hg.num_src_nodes(ntype) | 返回图中源节点的数量 | 4 |
| g.num_dst_nodes() | hg.num_dst_nodes(ntype) | 返回图中汇节点的数量 | 4 |
| g.nodes() | hg.nodes(ntype) | 返回节点id列表 | tensor([0, 1, 2, 3]) |
| g.edges() | hg.edges(etype) | 返回边,以(u,v)形式 | (tensor([0, 0, 0, 1, 2]), tensor([1, 2, 3, 3, 1])) |
| g.edges(form=‘all’) | 边的起始,结束节点,边的ID | (tensor([0, 0, 0, 1, 2]), tensor([1, 2, 3, 3, 1]), tensor([0, 1, 2, 3, 4])) | |
| g.srcnodes() | hg.srcnodes(ntype) | 源节点id列表 | tensor([0, 1, 2, 3]) |
| g.dstnodes() | hg.dstnodes(ntype) | 汇节点id列表 | tensor([0, 1, 2, 3]) |
| g.has_nodes(vid) | hg.has_nodes(vid, ntype) | 是否含有某节点,vid为节点的id | True |
| g.has_edges_between(u,v) | hg.has_edges_between(u,v,etype) | 是否含有某边 | tensor([False, False]) |
| g.edge_ids(u,v) | hg.edge_ids(u,v,etype) | 返回两端点之间边的id | g.edge_ids(0,1)->0 |
| g.find_edges(eid[,etype] | 返回该边的两端点 | g.find_edges((4, 2))->(tensor([2, 0]), tensor([1, 3])) | |
| g.in_edges(v) | hg.in_edges(v,etype) | 该节点的输入边 | g.in_edges(1)->(tensor([0, 2]), tensor([1, 1])) |
| g.out_edges(u) | hg.out_edges(u,etype) | 该节点的输出边 | |
| g.in_degrees(v) | hg.in_degrees(v,etype) | 入度 | g.in_degrees(1)->2 |
| g.out_degrees(u) | hg.out_degrees(u,etype) | 出度 |
2.2 关于图的类型
| – | hg.is_unibipartite | 是否是二分图 | False |
| g.is_multigraph | hg.is_multigraph | 是否是多关系图 | False |
| g.is_homogeneous | hg.is_homogeneous | 是否是同质图 | True |
-
二分图 unibipartite
图中节点属性可以分为两类SRC与DST,使得所有边均为SRC中节点指向DST中节点 -
多关系图 multigraph
多关系图指同一对节点间有多个边,称为并行边parallel edges,对于同质图,只意味着边的两端节点相同,对于异质图,还意味着边的类型相同,即规范边相同
2.3 关于图特征的属性
| g.ndata[fea_name]=tensor | hg.nodes[ntype].data[fea_name]=tensor | 创建特征/data | |
| g.edata[fea_name]=tensor | hg.edges[etype].data[fea_name]=tensor | 创建特征/data | |
| g.ndata | hg.nodes[nodes].data | 返回节点data字典 | {‘n_fe’: tensor([[1., 1., 1.],…]), ‘n_fe_matrix’: tensor([[[0.9555, 0.1653]…]])} |
| g.edata | hg.edges[etype].data | 返回边data字典 | {‘e_fe’: tensor([[0., 0., 0., 0., 0.]…} |
| g.srcdata | 源节点data | ||
| g.dstdata | 汇节点data |
图特征的切片操作:
| g.ndata[‘x’]=th.randn(10,3) | 对所有节点赋特征值 |
| g.ndata[‘x’][0]=th.zeros(1,3) | 对单个节点赋特征值 |
| g.ndata[‘x’][[0,5,6]]=th.zeros(3,3) | 对多个节点赋特征值 |
| g.ndata[‘x’][th.tensor([0, 5, 6])]=th.randn(3,3) | 对多个节点赋特征值 |
| g.edata[‘w’]=th.randn(9, 2) | 对所有边赋特征值 |
| g.data[‘w’][1]=th.randn(1,2) | 对单个边赋特征值 |
| g.edata[‘w’][[0,5,6]]=th.randn(3,2) | 对多个边赋特征值 |
| g.edata[‘w’][th.tensor([0,5,6])]=th.randn(3,2) | 对多个边赋特征值 |
2.4 数据类型
DGL可使用int32或着int64类型存储节点和边的ID。节点和边ID的数据类型应当一致。
| ID数据类型 | node和edge的个数上限 |
|---|---|
| int32 | 2 31 − 1 2^{31}-1 231−1 |
| int64 | 2 63 − 1 2^{63}-1 263−1 |
节点数较小时,应使用int32类型,因其可以有更快的速度和更小的存储空间
指定,转换与查看ID的数据类型
| 查看数据类型 | g.idtype | torch.int64(默认) |
| 指定 | g = dgl.graph(edges, idtype=torch.int32) | |
变换为int64 |
g64 = g.long() | |
变换为int32 |
g32 = g.int() |
1.3.5 邻接与指示矩阵
| DGLGraph.adj([transpose, ctx, scipy_fmt, etype]) | 返回指定类型边的邻接矩阵 | |
| DGLGraph.inc(typestr[, ctx, etype]) | 返回指定边类型的指示矩阵 |