glmnet提供了LASSO或ridge regression的Cox-PH分析模式,用于研究预测变量与生存时间的关系。具体做法先训练模型再使用最佳参数构建模型。更多知识分享请到 https://zouhua.top/。
加载数据
library(glmnet)
library(survival)
data(CoxExample)
phen <- y
rownames(phen) <- paste0("S", c(1:nrow(phen)))
head(phen) # 行样本名字,列是生存时间和状态
time status
S1 1.76877757 1
S2 0.54528404 1
S3 0.04485918 0
S4 0.85032298 0
S5 0.61488426 1
S6 0.29860939 0
prof <- x
rownames(prof) <- rownames(phen)
colnames(prof) <- paste0("Feature", c(1:ncol(prof)))
head(prof)
Feature1 Feature2 Feature3 Feature4 Feature5 Feature6 Feature7 Feature8 Feature9 Feature10
S1 -0.8767670 -0.6135224 -0.56757380 0.6621599 1.82218019 -1.0906678 -0.33186564 3.6754612 0.24580798 1.1382203
S2 -0.7463894 -1.7519457 0.28545898 1.1392105 0.80178007 1.8501985 0.30663005 -1.3729036 -0.03249051 0.7477848
S3 1.3759148 -0.2641132 0.88727408 0.3841870 0.05751801 -1.0917341 0.82119791 2.2960618 -0.44769567 -0.3046003
S4 0.2375820 0.7859162 -0.89670281 -0.8339338 -0.58237643 0.1874136 -0.58595131 0.4762090 -0.60580025 -1.2703322
S5 0.1086275 0.4665686 -0.57637261 1.7041314 0.32750715 -0.1211972 0.88537209 0.4505604 0.58878157 0.5504976
S6 1.2027213 -0.4187073 -0.05735193 0.5948491 0.44328682 -0.1191545 0.08097645 0.1645867 0.35648515 0.7186709
训练参数
设置alpha=0是ridge regression; alpha=1是LASSO;设置lambda,nlambda = 100或者lambda = 10^seq(3, -2, by = -.1);family选择不同数据分布情况,cox是cox-ph(其他选择"gaussian", "binomial", "poisson", "multinomial", "mgaussian"符合不同的数据)。
set.seed(123)
cv.fit <- cv.glmnet(prof, phen,
family = "cox",
type.measure = "C",
nfolds = 10,
alpha = 0,
nlambda = 100)
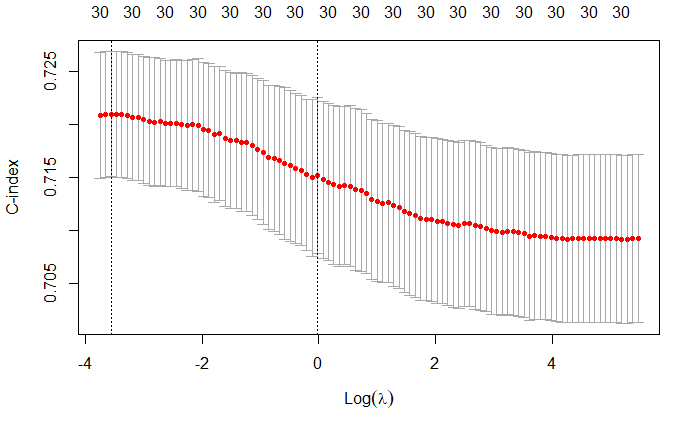
plot(cv.fit)
y轴坐标C-index原名是Harrell’concordance index,是用于评估模型的预测精度,常用于临床研究。x轴是lambda的log化结果,我们常选择最小的lambda值作为建模参数,也即是途中最大的C-index值。
C-index的计算方法是把所研究的资料中的所有研究对象随机地两两组成对子,以生存分析为例,两个病人如果生存时间较长的一位其预测生存时间长于另一位,或预测的生存概率高的一位的生存时间长于另一位,则称之为预测结果与实际结果相符,称之为一致。
计算C-index=K/M。
从上述计算方法可以看出C-index在0.5-1之间(任意配对随机情况下一致与不一致刚好是0.5的概率)。0.5为完全不一致,说明该模型没有预测作用,1为完全一致,说明该模型预测结果与实际完全一致。一般情况下C-index在0.50-0.70为准确度较低:在0.71-0.90之间为准确度中等;而高于0.90则为高准确度,跟相关系数有点类似。
构建模型
选择cv.fit最小lambda值
fit <- glmnet(prof, phen,
family = "cox",
alpha = 0,
lambda = cv.fit$lambda.min)
summary(fit)
Length Class Mode
a0 0 -none- NULL
beta 30 dgCMatrix S4
df 1 -none- numeric
dim 2 -none- numeric
lambda 1 -none- numeric
dev.ratio 1 -none- numeric
nulldev 1 -none- numeric
npasses 1 -none- numeric
jerr 1 -none- numeric
offset 1 -none- logical
call 6 -none- call
nobs 1 -none- numeric
- 每个features的coefficient
coef(fit)
30 x 1 sparse Matrix of class "dgCMatrix"
s0
Feature1 0.5114403878
Feature2 -0.1954830557
Feature3 -0.2405974927
Feature4 0.1957977902
Feature5 -0.2074132864
Feature6 -0.5056236002
Feature7 0.3552934341
Feature8 0.1057532384
Feature9 0.4648827155
Feature10 0.1375101610
Feature11 -0.0194344395
Feature12 0.0047078816
Feature13 0.0410461245
Feature14 0.0021407848
Feature15 -0.0009016771
Feature16 0.0001994629
Feature17 -0.0372298071
Feature18 -0.0100297637
Feature19 0.0080887542
Feature20 -0.0001315014
Feature21 -0.0218432109
Feature22 -0.0238062971
Feature23 -0.0054209272
Feature24 -0.0067311829
Feature25 -0.0488808852
Feature26 -0.0040028665
Feature27 0.0286530927
Feature28 -0.0136744813
Feature29 0.0086380442
Feature30 -0.0336064180
可根据系数选择重要的features进行后续Cox-PH分析。
计算样本C-index
最佳lambda参数下构建模型的C-index值,反应模型的预测精度,越高越好
pred <- predict(fit, newx = prof)
apply(pred, 2, Cindex, y=phen)
s0
0.7344005
参考
Regularized Cox Regression
How and when: ridge regression with glmnet
一致性指数:Harrell’concordance index:C-index
Calculate Concordance index
Calculate Brier score on glmnet object
参考文章如引起任何侵权问题,可以与我联系,谢谢。