由于每个月都会有相关的数据库文章发表,所以我们打算在每个月都会先出一个汇总贴来介绍一下这个月都发表了哪些在线的数据库。之后也会挑选我们自己感兴趣的一些数据库来详细的介绍。所以这次就先看一下五月份都发表了哪些在线的数据库吧。
五月份一共发表了20个在线的数据库,主要涉及到基因RNA、蛋白的分析以及药物相关的预测。同时对于高通量的一站式的分析方法也有几个数据库。接下来,我们就简单的结下一下这些数据库吧
RNA相关数据库
5月份的数据库总共包括四个RNA相关的数据库。
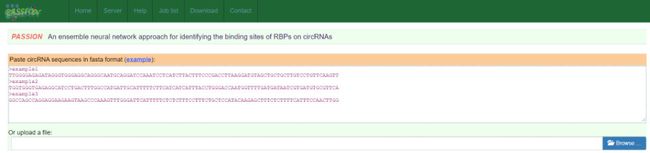
1. PASSION
PASSION(http://flagship.erc.monash.edu/PASSION/)数据库是用来预测在circRNA上的RNA绑定蛋白的绑定位点的数据库。我们需要数据circRNA的序列来预测其相关相关的绑定位点
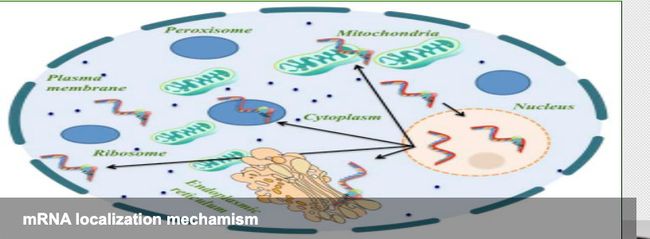
2. mRNALoc
mRNALoc(http://proteininformatics.org/mkumar/mrnaloc)数据库可以预测mRNA的定位。关于预测mRNA数据库网站后很多。这个数据库和他的有什么区别。这个等以后看具体文章在介绍吧。
3. FGviewer
FGviewer(https://ccsmweb.uth.edu/FGviewer)融合基因的功能注释工具。
4. SkeleDock
SkeleDock(https://playmolecule.org/SkeleDock/)是一个人和老鼠lncRNA查询的综合性数据库。我们可以输入一段lncRNA的序列来就可以获得这个lncRNA相关的功能和调控
蛋白相关数据库
5月份总共发表了6篇和蛋白相关的数据库。由于蛋白方面涉及较少,所以介绍就偏向于数据库当中的简介,需要的可以自己去具体看一下。
1. webPSN v2.0
webPSN v2.0(http://webpsn.hpc.unimo.it/wpsn.php)是一个预测蛋白质变构作用的数据库。它采用了混合蛋白质结构网络(PSN)和弹性网络模型-简正模式分析(ENM-NMA)策略来研究蛋白质和核酸中的结构结合。
2. PL-search
PL-search(http://bliulab.net/PL-search/)是一个研究蛋白同源性检测的数据库。
3. PlaToLoCo
PlaToLoCo(http://platoloco.aei.polsl.pl/)蛋白质低复杂度区域可视化和注释数据库。我们可以输入想要比较多蛋白ID。然后就可以获得结果了。
4. PLIDflow
PLIDflow(https://pistacho.ac.uma.es/galaxy-bitlab)是一个在线分析蛋白质-配体对接的流程型的数据库。
5. MISCAST
MISCAST(http://miscast.broadinstitute.org/)预测基因的突变对于蛋白结构影响的数据库。我们输入相关的基因即可获得结果
6. Procleave
Procleave(http://procleave.erc.monash.edu/)是一个通过结合序列以及结构位点来预测蛋白酶特异性裂解位点的数据库。我们需要输入蛋白的名称、蛋白链、以及想要查看的裂解酶
7. GalaxySagittarius
GalaxySagittarius(http://galaxy.seoklab.org/cgi-bin/submit.cgi?type=SAGITTARIUS)是一个预测蛋白质-配体对接的数据库
药物相关数据库
五月份一共发表了三篇和药物相关的数据库
1. ARTS 2.0
ARTS 2.0(http://arts.ziemertlab.com/)是一个预测抗生素抵抗靶点的数据库。
2. PaccMann
PaccMann(https://ibm.biz/paccmann-aas)是一个抗癌药物敏感性预测的数据库。
分析方法集成数据库
对于高通量的数据,分析方法都有相对应的流程。往往这个流程需要一定的变成技巧,现在一些的数据库也致力于把分析方法的门槛降低。这样的数据库需要我们提供原始数据,但是不需要变成的技巧就行完成分析。五月份有4个分析方法方面的数据库
1.RiboToolkit
RiboToolkit(http://rnabioinfor.tch.harvard.edu/RiboToolkit/)是一个网络端的分析核糖体测序(Ribo-seq)数据的数据库。这个数据库要求我们提供测序的原始数据。来进行下游的分析。
2. PolySTest
PolySTest(http://computproteomics.bmb.sdu.dk/Apps/PolySTest)这个数据库主要针对的是缺失值很多的蛋白组学的数据库。对于缺失值很多的蛋白组学的数据,这个数据库提供了一种折中的分析方法来进行相对应的分析
3. tappAS
tappAS(https://app.tappas.org/)是一个桌面端的软件,这个软件主要是用来分析可变剪切的变化,同时对其进行功能注释
其他数据库
有一些不知道怎么归类的就先放到这里吧。一共有4个数据库
[图片上传中...(image-94748e-1590374338138-4)]
1.HDVdb
HDVdb(http://hdvdb.bio.wzw.tum.de/hdvdb/)是一个综合性查询丙肝的数据库。如果要研究丙肝的同学可以用一下。
2. IRIS3
IRIS3(https://bmbl.bmi.osumc.edu/iris3/)是一个通过单细胞测序的数据来寻找细胞特异性调控因子的数据库。
3. AcrFinder
AcrFinder(http://bcb.unl.edu/AcrFinder/)是一个通过基因组数据挖掘原核生物及其病毒中的反CRISPR操纵子。
4. mmCSM-AB
mmCSM-AB(http://biosig.unimelb.edu.au/mmcsm_ab/)是一个分析多点突变对抗体-抗原结合亲和力影响的工具。
基本上五月份发表的数据库就这些。中间有一些数据库,有可能基于检索策略我们会漏掉一些,但是大部分的都在这里吧。有需要哪个的同学可以具体的看看哈。