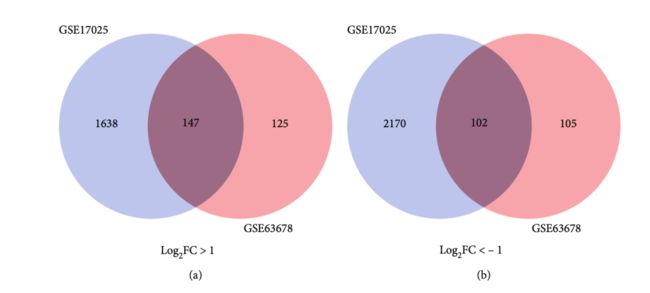
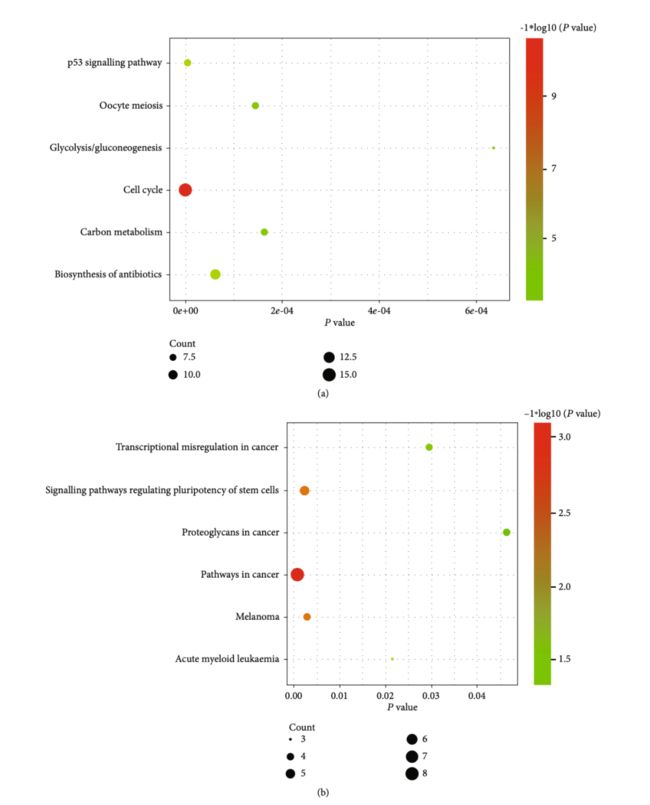
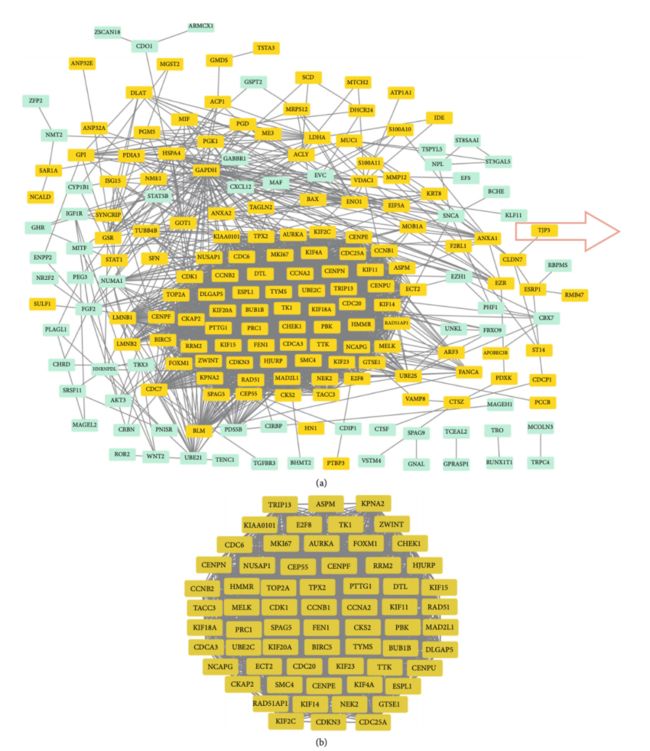
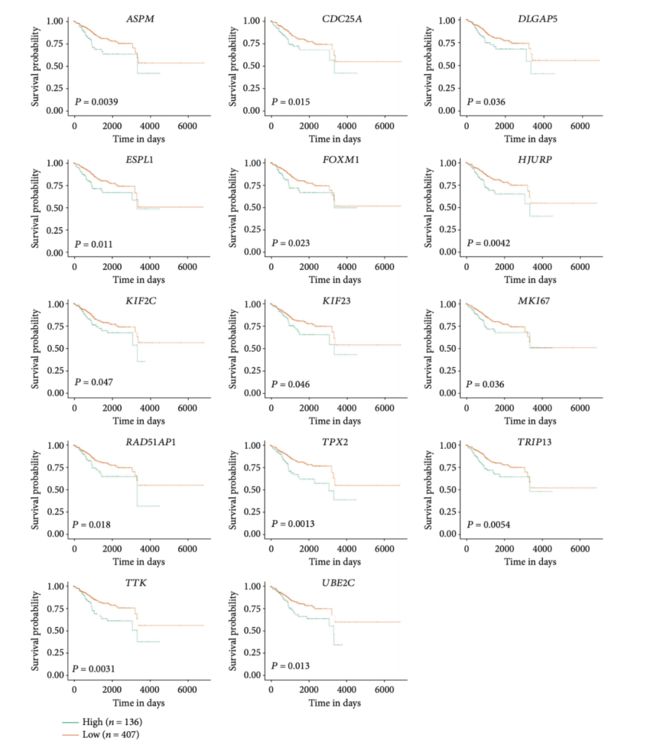
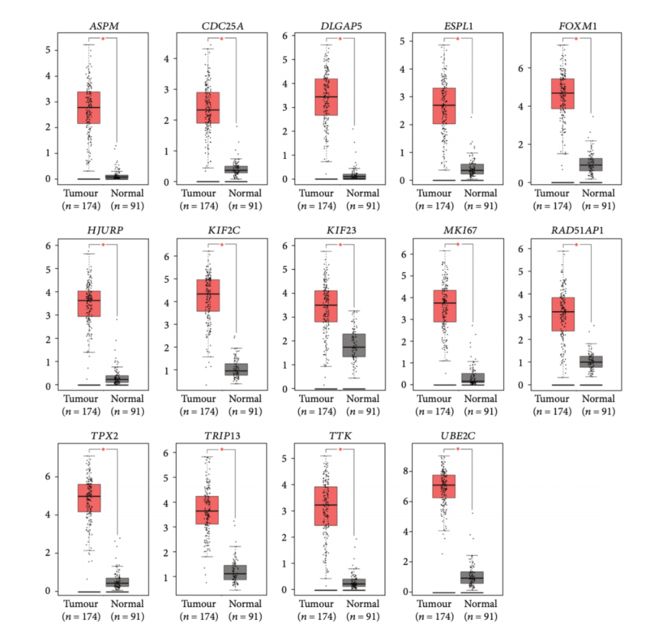
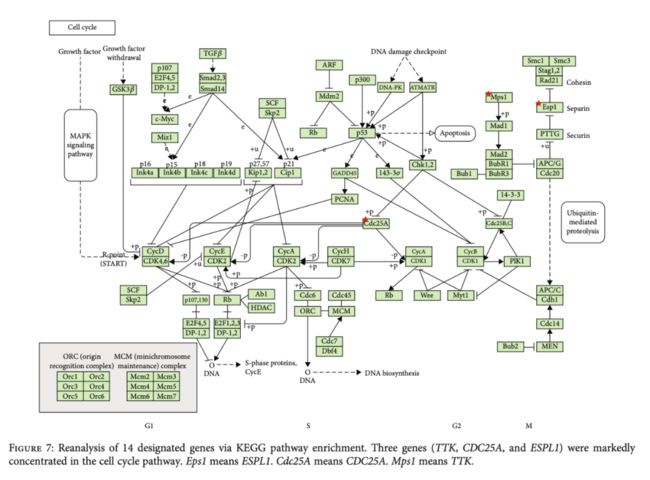
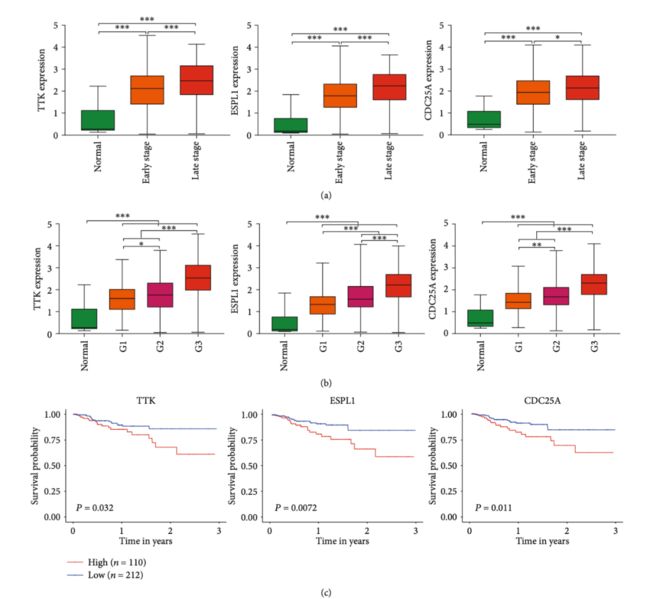
一、什么是宏函数?通过宏定义的函数是宏函数。如下,编译器在预处理阶段会将Add(x,y)替换为((x)*(y))#defineAdd(x,y)((x)*(y))#defineAdd(x,y)((x)*(y))intmain(){inta=10;intb=20;intd=10;intc=Add(a+d,b)*2;cout<
地推话术,如何应对地推过程中家长的拒绝
校师学
相信校长们在做地推的时候经常遇到这种情况:市场专员反馈家长不接单,咨询师反馈难以邀约这些家长上门,校区地推疲软,招生难。为什么?仅从地推层面分析,一方面因为家长受到的信息轰炸越来越多,对信息越来越“免疫”;而另一方面地推人员的专业能力和营销话术没有提高,无法应对家长的拒绝,对有意向的家长也不知如何跟进,眼睁睁看着家长走远;对于家长的疑问,更不知道如何有技巧地回答,机会白白流失。由于回答没技巧和专业
谢谢你们,爱你们!
鹿游儿
昨天家人去泡温泉,二个孩子也带着去,出发前一晚,匆匆下班,赶回家和孩子一起收拾。饭后,我拿出笔和本子(上次去澳门时做手帐的本子)写下了1\2\3\4\5\6\7\8\9,让后让小壹去思考,带什么出发去旅游呢?她在对应的数字旁边画上了,泳衣、泳圈、肖恩、内衣内裤、tapuy、拖鞋……画完后,就让她自己对着这个本子,将要带的,一一带上,没想到这次带的书还是这本《便便工厂》(晚上姑婆发照片过来,妹妹累得
C语言如何定义宏函数?
小九格物
c语言
在C语言中,宏函数是通过预处理器定义的,它在编译之前替换代码中的宏调用。宏函数可以模拟函数的行为,但它们不是真正的函数,因为它们在编译时不会进行类型检查,也不会分配存储空间。宏函数的定义通常使用#define指令,后面跟着宏的名称和参数列表,以及宏展开后的代码。宏函数的定义方式:1.基本宏函数:这是最简单的宏函数形式,它直接定义一个表达式。#defineSQUARE(x)((x)*(x))2.带参
微服务下功能权限与数据权限的设计与实现
nbsaas-boot
微服务java架构
在微服务架构下,系统的功能权限和数据权限控制显得尤为重要。随着系统规模的扩大和微服务数量的增加,如何保证不同用户和服务之间的访问权限准确、细粒度地控制,成为设计安全策略的关键。本文将讨论如何在微服务体系中设计和实现功能权限与数据权限控制。1.功能权限与数据权限的定义功能权限:指用户或系统角色对特定功能的访问权限。通常是某个用户角色能否执行某个操作,比如查看订单、创建订单、修改用户资料等。数据权限:
理解Gunicorn:Python WSGI服务器的基石
范范0825
ipythonlinux运维
理解Gunicorn:PythonWSGI服务器的基石介绍Gunicorn,全称GreenUnicorn,是一个为PythonWSGI(WebServerGatewayInterface)应用设计的高效、轻量级HTTP服务器。作为PythonWeb应用部署的常用工具,Gunicorn以其高性能和易用性著称。本文将介绍Gunicorn的基本概念、安装和配置,帮助初学者快速上手。1.什么是Gunico
小丽成长记(四十三)
玲玲54321
小丽发现,即使她好不容易调整好自己的心态下一秒总会有不确定的伤脑筋的事出现,一个接一个的问题,人生就没有停下的时候,小问题不断出现。不过她今天看的书,她接受了人生就是不确定的,厉害的人就是不断创造确定性,在Ta的领域比别人多的确定性就能让自己脱颖而出,显示价值从而获得的比别人多的利益。正是这样的原因,因为从前修炼自己太少,使得她现在在人生道路上打怪起来困难重重,她似乎永远摆脱不了那种无力感,有种习
学点心理知识,呵护孩子健康
静候花开_7090
昨天听了华中师范大学教育管理学系副教授张玲老师的《哪里才是学生心理健康的最后庇护所,超越教育与技术的思考》的讲座。今天又重新学习了一遍,收获匪浅。张玲博士也注意到了当今社会上的孩子由于心理问题导致的自残、自杀及伤害他人等恶性事件。她向我们普及了一个重要的命题,她说心理健康的一些基本命题,我们与我们通常的一些教育命题是不同的,她还举了几个例子,让我们明白我们原来以为的健康并非心理学上的健康。比如如果
2021年12月19日,春蕾教育集团团建活动感受——黄晓丹
黄错错加油
感受:1.从陌生到熟悉的过程。游戏环节让我们在轻松的氛围中得到了锻炼,也增长了不少知识。2.游戏过程中,我们贡献的是个人力量,展现的是团队的力量。它磨合的往往不止是工作的熟悉,更是观念上契合度的贴近。3.这和工作是一样的道理。在各自的岗位上,每个人摆正自己的位置、各司其职充分发挥才能,并团结一致劲往一处使,才能实现最大的成功。新知:1.团队精神需要不断地创新。过去,人们把创新看作是冒风险,现在人们
Cell Insight | 单细胞测序技术又一新发现,可用于HIV-1和Mtb共感染个体诊断
尐尐呅
结核病是艾滋病合并其他疾病中导致患者死亡的主要原因。其中结核病由结核分枝杆菌(Mycobacteriumtuberculosis,Mtb)感染引起,获得性免疫缺陷综合症(艾滋病)由人免疫缺陷病毒(Humanimmunodeficiencyvirustype1,HIV-1)感染引起。国家感染性疾病临床医学研究中心/深圳市第三人民医院张国良团队携手深圳华大生命科学研究院吴靓团队,共同研究得出单细胞测序
c++ 的iostream 和 c++的stdio的区别和联系
黄卷青灯77
c++算法开发语言iostreamstdio
在C++中,iostream和C语言的stdio.h都是用于处理输入输出的库,但它们在设计、用法和功能上有许多不同。以下是两者的区别和联系:区别1.编程风格iostream(C++风格):C++标准库中的输入输出流类库,支持面向对象的输入输出操作。典型用法是cin(输入)和cout(输出),使用>操作符来处理数据。更加类型安全,支持用户自定义类型的输入输出。#includeintmain(){in
瑶池防线
谜影梦蝶
冥华虽然逃过了影梦的军队,但他是一个忠臣,他选择上报战况。败给影梦后成逃兵,高层亡尔还活着,七重天失守......随便一条,即可处死冥华。冥华自然是知道以仙界高层的习性此信一发自己必死无疑,但他还选择上报实情,因为责任。同样此信送到仙宫后,知道此事的人,大多数人都认定冥华要完了,所以上到仙界高层,下到扫大街的,包括冥华自己,全都准备好迎接冥华之死。如果仙界现在还属于两方之争的话,冥华必死无疑。然而
爬山后遗症
璃绛
爬山,攀登,一步一步走向制高点,是一种挑战。成功抵达是一种无法言语的快乐,在山顶吹吹风,看看风景,这是从未有过的体验。然而,爬山一时爽,下山腿打颤,颠簸的路,一路向下走,腿部力量不够,走起来抖到不行,停不下来了!第二天必定腿疼,浑身酸痛,坐立难安!
knob UI插件使用
换个号韩国红果果
JavaScriptjsonpknob
图形是用canvas绘制的
js代码
var paras = {
max:800,
min:100,
skin:'tron',//button type
thickness:.3,//button width
width:'200',//define canvas width.,canvas height
displayInput:'tr
Android+Jquery Mobile学习系列(5)-SQLite数据库
白糖_
JQuery Mobile
目录导航
SQLite是轻量级的、嵌入式的、关系型数据库,目前已经在iPhone、Android等手机系统中使用,SQLite可移植性好,很容易使用,很小,高效而且可靠。
因为Android已经集成了SQLite,所以开发人员无需引入任何JAR包,而且Android也针对SQLite封装了专属的API,调用起来非常快捷方便。
我也是第一次接触S
impala-2.1.2-CDH5.3.2
dayutianfei
impala
最近在整理impala编译的东西,简单记录几个要点:
根据官网的信息(https://github.com/cloudera/Impala/wiki/How-to-build-Impala):
1. 首次编译impala,推荐使用命令:
${IMPALA_HOME}/buildall.sh -skiptests -build_shared_libs -format
2.仅编译BE
${I
求二进制数中1的个数
周凡杨
java算法二进制
解法一:
对于一个正整数如果是偶数,该数的二进制数的最后一位是 0 ,反之若是奇数,则该数的二进制数的最后一位是 1 。因此,可以考虑利用位移、判断奇偶来实现。
public int bitCount(int x){
int count = 0;
while(x!=0){
if(x%2!=0){ /
spring中hibernate及事务配置
g21121
Hibernate
hibernate的sessionFactory配置:
<!-- hibernate sessionFactory配置 -->
<bean id="sessionFactory"
class="org.springframework.orm.hibernate3.LocalSessionFactoryBean">
<
log4j.properties 使用
510888780
log4j
log4j.properties 使用
一.参数意义说明
输出级别的种类
ERROR、WARN、INFO、DEBUG
ERROR 为严重错误 主要是程序的错误
WARN 为一般警告,比如session丢失
INFO 为一般要显示的信息,比如登录登出
DEBUG 为程序的调试信息
配置日志信息输出目的地
log4j.appender.appenderName = fully.qua
Spring mvc-jfreeChart柱图(2)
布衣凌宇
jfreechart
上一篇中生成的图是静态的,这篇将按条件进行搜索,并统计成图表,左面为统计图,右面显示搜索出的结果。
第一步:导包
第二步;配置web.xml(上一篇有代码)
建BarRenderer类用于柱子颜色
import java.awt.Color;
import java.awt.Paint;
import org.jfree.chart.renderer.category.BarR
我的spring学习笔记14-容器扩展点之PropertyPlaceholderConfigurer
aijuans
Spring3
PropertyPlaceholderConfigurer是个bean工厂后置处理器的实现,也就是BeanFactoryPostProcessor接口的一个实现。关于BeanFactoryPostProcessor和BeanPostProcessor类似。我会在其他地方介绍。
PropertyPlaceholderConfigurer可以将上下文(配置文件)中的属性值放在另一个单独的标准java
maven 之 cobertura 简单使用
antlove
maventestunitcoberturareport
1. 创建一个maven项目
2. 创建com.CoberturaStart.java
package com;
public class CoberturaStart {
public void helloEveryone(){
System.out.println("=================================================
程序的执行顺序
百合不是茶
JAVA执行顺序
刚在看java核心技术时发现对java的执行顺序不是很明白了,百度一下也没有找到适合自己的资料,所以就简单的回顾一下吧
代码如下;
经典的程序执行面试题
//关于程序执行的顺序
//例如:
//定义一个基类
public class A(){
public A(
设置session失效的几种方法
bijian1013
web.xmlsession失效监听器
在系统登录后,都会设置一个当前session失效的时间,以确保在用户长时间不与服务器交互,自动退出登录,销毁session。具体设置很简单,方法有三种:(1)在主页面或者公共页面中加入:session.setMaxInactiveInterval(900);参数900单位是秒,即在没有活动15分钟后,session将失效。这里要注意这个session设置的时间是根据服务器来计算的,而不是客户端。所
java jvm常用命令工具
bijian1013
javajvm
一.概述
程序运行中经常会遇到各种问题,定位问题时通常需要综合各种信息,如系统日志、堆dump文件、线程dump文件、GC日志等。通过虚拟机监控和诊断工具可以帮忙我们快速获取、分析需要的数据,进而提高问题解决速度。 本文将介绍虚拟机常用监控和问题诊断命令工具的使用方法,主要包含以下工具:
&nbs
【Spring框架一】Spring常用注解之Autowired和Resource注解
bit1129
Spring常用注解
Spring自从2.0引入注解的方式取代XML配置的方式来做IOC之后,对Spring一些常用注解的含义行为一直处于比较模糊的状态,写几篇总结下Spring常用的注解。本篇包含的注解有如下几个:
Autowired
Resource
Component
Service
Controller
Transactional
根据它们的功能、目的,可以分为三组,Autow
mysql 操作遇到safe update mode问题
bitray
update
我并不知道出现这个问题的实际原理,只是通过其他朋友的博客,文章得知的一个解决方案,目前先记录一个解决方法,未来要是真了解以后,还会继续补全.
在mysql5中有一个safe update mode,这个模式让sql操作更加安全,据说要求有where条件,防止全表更新操作.如果必须要进行全表操作,我们可以执行
SET
nginx_perl试用
ronin47
nginx_perl试用
因为空闲时间比较多,所以在CPAN上乱翻,看到了nginx_perl这个项目(原名Nginx::Engine),现在托管在github.com上。地址见:https://github.com/zzzcpan/nginx-perl
这个模块的目的,是在nginx内置官方perl模块的基础上,实现一系列异步非阻塞的api。用connector/writer/reader完成类似proxy的功能(这里
java-63-在字符串中删除特定的字符
bylijinnan
java
public class DeleteSpecificChars {
/**
* Q 63 在字符串中删除特定的字符
* 输入两个字符串,从第一字符串中删除第二个字符串中所有的字符。
* 例如,输入”They are students.”和”aeiou”,则删除之后的第一个字符串变成”Thy r stdnts.”
*/
public static voi
EffectiveJava--创建和销毁对象
ccii
创建和销毁对象
本章内容:
1. 考虑用静态工厂方法代替构造器
2. 遇到多个构造器参数时要考虑用构建器(Builder模式)
3. 用私有构造器或者枚举类型强化Singleton属性
4. 通过私有构造器强化不可实例化的能力
5. 避免创建不必要的对象
6. 消除过期的对象引用
7. 避免使用终结方法
1. 考虑用静态工厂方法代替构造器
类可以通过
[宇宙时代]四边形理论与光速飞行
comsci
从四边形理论来推论 为什么光子飞船必须获得星光信号才能够进行光速飞行?
一组星体组成星座 向空间辐射一组由复杂星光信号组成的辐射频带,按照四边形-频率假说 一组频率就代表一个时空的入口
那么这种由星光信号组成的辐射频带就代表由这些星体所控制的时空通道,该时空通道在三维空间的投影是一
ubuntu server下python脚本迁移数据
cywhoyi
pythonKettlepymysqlcx_Oracleubuntu server
因为是在Ubuntu下,所以安装python、pip、pymysql等都极其方便,sudo apt-get install pymysql,
但是在安装cx_Oracle(连接oracle的模块)出现许多问题,查阅相关资料,发现这边文章能够帮我解决,希望大家少走点弯路。http://www.tbdazhe.com/archives/602
1.安装python
2.安装pip、pymysql
Ajax正确但是请求不到值解决方案
dashuaifu
Ajaxasync
Ajax正确但是请求不到值解决方案
解决方案:1 . async: false , 2. 设置延时执行js里的ajax或者延时后台java方法!!!!!!!
例如:
$.ajax({ &
windows安装配置php+memcached
dcj3sjt126com
PHPInstallmemcache
Windows下Memcached的安装配置方法
1、将第一个包解压放某个盘下面,比如在c:\memcached。
2、在终端(也即cmd命令界面)下输入 'c:\memcached\memcached.exe -d install' 安装。
3、再输入: 'c:\memcached\memcached.exe -d start' 启动。(需要注意的: 以后memcached将作为windo
iOS开发学习路径的一些建议
dcj3sjt126com
ios
iOS论坛里有朋友要求回答帖子,帖子的标题是: 想学IOS开发高阶一点的东西,从何开始,然后我吧啦吧啦回答写了很多。既然敲了那么多字,我就把我写的回复也贴到博客里来分享,希望能对大家有帮助。欢迎大家也到帖子里讨论和分享,地址:http://bbs.csdn.net/topics/390920759
下面是我回复的内容:
结合自己情况聊下iOS学习建议,
Javascript闭包概念
fanfanlovey
JavaScript闭包
1.参考资料
http://www.jb51.net/article/24101.htm
http://blog.csdn.net/yn49782026/article/details/8549462
2.内容概述
要理解闭包,首先需要理解变量作用域问题
内部函数可以饮用外面全局变量
var n=999;
functio
yum安装mysql5.6
haisheng
mysql
1、安装http://dev.mysql.com/get/mysql-community-release-el7-5.noarch.rpm
2、yum install mysql
3、yum install mysql-server
4、vi /etc/my.cnf 添加character_set_server=utf8
po/bo/vo/dao/pojo的详介
IT_zhlp80
javaBOVODAOPOJOpo
JAVA几种对象的解释
PO:persistant object持久对象,可以看成是与数据库中的表相映射的java对象。最简单的PO就是对应数据库中某个表中的一条记录,多个记录可以用PO的集合。PO中应该不包含任何对数据库的操作.
VO:value object值对象。通常用于业务层之间的数据传递,和PO一样也是仅仅包含数据而已。但应是抽象出的业务对象,可
java设计模式
kerryg
java设计模式
设计模式的分类:
一、 设计模式总体分为三大类:
1、创建型模式(5种):工厂方法模式,抽象工厂模式,单例模式,建造者模式,原型模式。
2、结构型模式(7种):适配器模式,装饰器模式,代理模式,外观模式,桥接模式,组合模式,享元模式。
3、行为型模式(11种):策略模式,模版方法模式,观察者模式,迭代子模式,责任链模式,命令模式,备忘录模式,状态模式,访问者
[1]CXF3.1整合Spring开发webservice——helloworld篇
木头.java
springwebserviceCXF
Spring 版本3.2.10
CXF 版本3.1.1
项目采用MAVEN组织依赖jar
我这里是有parent的pom,为了简洁明了,我直接把所有的依赖都列一起了,所以都没version,反正上面已经写了版本
<project xmlns="http://maven.apache.org/POM/4.0.0" xmlns:xsi="ht
Google 工程师亲授:菜鸟开发者一定要投资的十大目标
qindongliang1922
工作感悟人生
身为软件开发者,有什么是一定得投资的? Google 软件工程师 Emanuel Saringan 整理了十项他认为必要的投资,第一项就是身体健康,英文与数学也都是必备能力吗?来看看他怎么说。(以下文字以作者第一人称撰写)) 你的健康 无疑地,软件开发者是世界上最久坐不动的职业之一。 每天连坐八到十六小时,休息时间只有一点点,绝对会让你的鲔鱼肚肆无忌惮的生长。肥胖容易扩大罹患其他疾病的风险,
linux打开最大文件数量1,048,576
tianzhihehe
clinux
File descriptors are represented by the C int type. Not using a special type is often considered odd, but is, historically, the Unix way. Each Linux process has a maximum number of files th
java语言中PO、VO、DAO、BO、POJO几种对象的解释
衞酆夼
javaVOBOPOJOpo
PO:persistant object持久对象
最形象的理解就是一个PO就是数据库中的一条记录。好处是可以把一条记录作为一个对象处理,可以方便的转为其它对象。可以看成是与数据库中的表相映射的java对象。最简单的PO就是对应数据库中某个表中的一条记录,多个记录可以用PO的集合。PO中应该不包含任何对数据库的操作。
BO:business object业务对象
封装业务逻辑的java对象