同系列文章:
- sc-RAN-seq 数据分析||Seurat新版教程:Guided Clustering Tutorial
- sc-RAN-seq 数据分析||Seurat新版教程: Integrating datasets to learn cell-type specific responses
- sc-RAN-seq 数据分析||Seurat新版教程: Using sctransform in Seurat
- 单细胞转录组数据分析||Seurat新版教程:Differential expression testing
- 单细胞转录组 数据分析||Seurat新版教程:New data visualization methods in v3.0
- 单细胞转录组数据分析||Seurat并行策略
- Seurat Weekly NO.0 || 开刊词
- Seurat Weekly NO.1 || 到底分多少个群是合适的?!
- Seurat Weekly NO.2 || 我该如何取子集
- 你到底想要什么样的umap/tsne图?
- scRNA-seq拟时分析 || Monocle2 踩坑教程
- scRNA-seq数据分析 || Monocle3
关于拟时分析的一些基本知识请参看以下两篇及其参考文章:
- scRNA-seq拟时分析 || Monocle2 踩坑教程
- STREAM:一款scRNA-seq拟时分析工具
更多详细内容请参考monocle3官网,本文仅为个人学习笔记。
在发育过程中,细胞对刺激作出反应,并在整个生命过程中,从一种功能“状态”过渡到另一种功能“状态”。不同状态的细胞表达不同的基因,产生蛋白质和代谢物的动态重复序列,从而完成它们的工作。当细胞在状态之间移动时,它们经历一个转录重组的过程,一些基因被沉默,另一些基因被激活。这些瞬时状态通常很难描述,因为在更稳定的端点状态之间纯化细胞可能是困难的或不可能的。单细胞RNA-Seq可以使您在不需要纯化的情况下看到这些状态。然而,要做到这一点,我们必须确定每个cell在可能的状态范围内的位置。
Monocle介绍了利用RNA-Seq进行单细胞轨迹分析的策略。Monocle不是通过实验将细胞纯化成离散状态,而是使用一种算法来学习每个细胞必须经历的基因表达变化序列,作为动态生物学过程的一部分。一旦它了解了基因表达变化的整体“轨迹”,Monocle就可以将每个细胞置于轨迹中的适当位置。然后,您可以使用Monocle的微分分析工具包来查找在轨迹过程中受到调控的基因,如查找作为伪时间函数变化的基因一节所述。如果这个过程有多个结果,Monocle将重建一个“分支”轨迹。这些分支与细胞的“决策”相对应,Monocle提供了强大的工具来识别受它们影响的基因,并参与这些基因的形成。在分析单细胞轨迹中的分支的小节中,您可以看到如何分析分支。
monocle 能做的不只是拟时分析,或者说为了做拟时分析他也做了sc-rna-seq的基本分析流程:数据读入,均一化,降维(PCA,umap,tsne,),聚类,marker基因筛选以及可视化函数。在新的学习中我们发现monocle能做的远不只这些,例如用shiny开发了web程序,更加用户友好;借助garnett包可以做细胞定义-----monocle已经是一个sc-rna-seq数据分析的工具箱。
monocle3
library(monocle3)
packageVersion("monocle3")
[1] ‘0.1.1’
monocle3是新的开始大部分的函数名称都改变了,连版本号都从零开始。
多种格式的数据读入,这里我们依然是从seurat3对象中读入:
#Generate a cell_data_set
#expression_matrix <- readRDS(url("http://staff.washington.edu/hpliner/data/cao_l2_expression.rds"))
#cell_metadata <- readRDS(url("http://staff.washington.edu/hpliner/data/cao_l2_colData.rds"))
#gene_annotation <- readRDS(url("http://staff.washington.edu/hpliner/data/cao_l2_rowData.rds"))
#cds <- new_cell_data_set(expression_matrix,
# cell_metadata = cell_metadata,
# gene_metadata = gene_annotation)
读10X数据,cellranger V2 V3 均可:
#Generate a cell_data_set from 10X output
# Provide the path to the Cell Ranger output.
#cds <- load_cellranger_data("D:\\Users\\Administrator\\Desktop\\RStudio\\single_cell\\V2")
setwd("D:\\Users\\Administrator\\Desktop\\Novo周运来\\SingleCell\\scrna_tools")
#library(monocle)
#<-importCDS(pbmc,import_all = TRUE)
#Load Seurat object
pbmc <- readRDS('D:\\Users\\Administrator\\Desktop\\RStudio\\single_cell\\filtered_gene_bc_matrices\\hg19pbmc_tutorial.rds')
#Extract data, phenotype data, and feature data from the SeuratObject
data <- as(as.matrix(pbmc@assays$RNA@counts), 'sparseMatrix')
pd <- [email protected]
fData <- data.frame(gene_short_name = row.names(data), row.names = row.names(data))
colnames(pd)
[1] "orig.ident" "nCount_RNA" "nFeature_RNA" "percent.mt" "RNA_snn_res.0.5" "seurat_clusters"
创建cds对象,注意这里每一项是什么。
#Construct monocle cds
cds <- new_cell_data_set(data,
cell_metadata = pd,
gene_metadata = fData)
> cds
class: cell_data_set
dim: 13714 2638
metadata(1): cds_version
assays(1): counts
rownames(13714): AL627309.1 AP006222.2 ... PNRC2.1 SRSF10.1
rowData names(1): gene_short_name
colnames(2638): AAACATACAACCAC AAACATTGAGCTAC ... TTTGCATGAGAGGC TTTGCATGCCTCAC
colData names(10): orig.ident nCount_RNA ... cell_type cluster_ext_type
reducedDimNames(2): PCA UMAP
spikeNames(0):
降维
- 标准化
Monocle3中的大多数分析(包括轨迹推断和聚类)都需要各种规范化和预处理步骤。preprocess_cds执行并存储这些预处理步骤。
具体地说,根据选择的参数,preprocess_cds首先根据日志和因子大小对数据进行规范化(normalizes),以处理深度差异,或者只根据大小因子进行规范化。有三种方法: PCA or LSI. For LS,默认是PCA。接下来,preprocess_cds计算一个较低的维度空间,该空间将用作进一步降维的输入,如tSNE和UMAP。
#Pre-process the data
cds = preprocess_cds(cds, num_dim = 100)
?preprocess_cds
plot_pc_variance_explained(cds)
可视化降维
- UMAP
#Reduce dimensionality and visualize the cells
cds = reduce_dimension(cds) #Monocle uses UMAP by default
plot_cells(cds)
#No trajectory to plot. Has learn_graph() been called yet?
head(rownames(cds))
"AL627309.1" "AP006222.2" "RP11-206L10.2" "RP11-206L10.9" "LINC00115" "NOC2L"
plot_cells(cds, genes=c("S100A9", "RPS27", "FCER1A", "GNLY"))
- tSNE
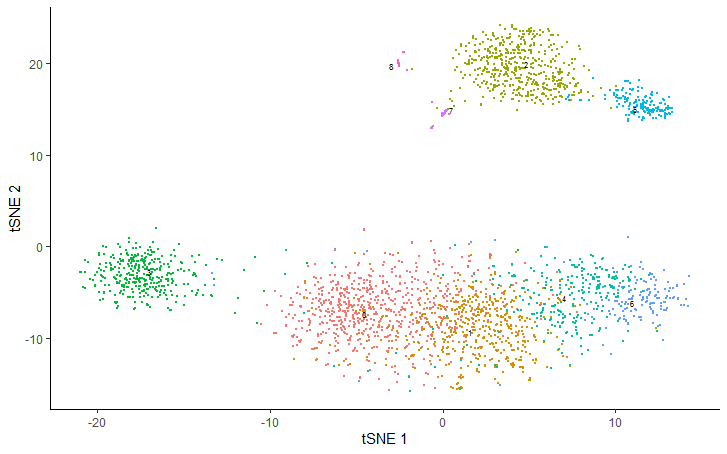
cds = reduce_dimension(cds, reduction_method="tSNE")
plot_cells(cds, reduction_method="tSNE", color_cells_by="seurat_clusters")
可以自己设置分组看看是否有批次效应。
#Check for and remove batch effects
plot_cells(cds, color_cells_by="seurat_clusters", label_cell_groups=FALSE)
聚类
无监督聚类是许多单细胞表达工作中的一个常见步骤。在包含多种细胞类型的实验中,每个cluxter可能对应不同的细胞类型。该函数接受cell_data_set作为输入,使用Louvain community detection对细胞进行聚类,并返回一个cell_data_set,其中包含内部存储的聚类结果。
#Group cells into clusters
#library(reticulate)
#use_python("E:\\conda\\python.exe")
#reticulate::py_install("louvain")
cds = cluster_cells(cds, python_home = "E:\\conda\\python",verbose = T,resolution=c(10^seq(-6,-1)))
Running louvain clustering algorithm ...
Run kNN based graph clustering starts:
-Input data of 2638 rows and 2 columns
-k is set to 20
Finding nearest neighbors...DONE ~ 0.129999999997381 s
Compute jaccard coefficient between nearest-neighbor sets ...DONE ~ 0.129999999997381 s
Build undirected graph from the weighted links ...DONE ~ 0.14 s
Run louvain clustering on the graph ...
Running louvain iteration 1 ...
Current iteration is 1; current resolution is 1e-06; Modularity is 0.547843095426104; Number of clusters are 3
Current iteration is 1; current resolution is 1e-05; Modularity is 0.547843095426104; Number of clusters are 3
Current iteration is 1; current resolution is 1e-04; Modularity is 0.547843095426104; Number of clusters are 3
Current iteration is 1; current resolution is 0.001; Modularity is 0.723545062987134; Number of clusters are 5
Current iteration is 1; current resolution is 0.01; Modularity is 0.865973276843872; Number of clusters are 13
Current iteration is 1; current resolution is 0.1; Modularity is 0.822758898433183; Number of clusters are 49
Maximal modularity is 0.865973276843872; corresponding resolution is 0.01
Run kNN based graph clustering DONE, totally takes 2.71215486526489 s.
-Number of clusters: 13
plot_cells(cds,graph_label_size=15,cell_size=1.5)
寻找marker 基因
#Find marker genes expressed by each cluster
marker_test_res = top_markers(cds, group_cells_by="seurat_clusters", reference_cells=1000, cores=8)
library(dplyr)
top_specific_markers = marker_test_res %>%
filter(fraction_expressing >= 0.10) %>%
group_by(cell_group) %>%
top_n(1, pseudo_R2)
top_specific_marker_ids = unique(top_specific_markers %>% pull(gene_id))
plot_genes_by_group(cds,
top_specific_marker_ids,
group_cells_by="seurat_clusters",
ordering_type="maximal_on_diag",
max.size=3)
top_specific_markers = marker_test_res %>%
filter(fraction_expressing >= 0.10) %>%
group_by(cell_group) %>%
top_n(3, pseudo_R2)
top_specific_marker_ids = unique(top_specific_markers %>% pull(gene_id))
plot_genes_by_group(cds,
top_specific_marker_ids,
group_cells_by="seurat_clusters",
ordering_type="cluster_row_col",
max.size=3)
已知细胞类型
#Annotate your cells according to type
colData(cds)$assigned_cell_type=as.character(clusters(cds))
colData(cds)$assigned_cell_type = dplyr::recode(colData(cds)$assigned_cell_type,
"1"="Body wall muscle",
"2"="Germline","3"="Unclassified neurons","4"="Seam cells",
"5"="Coelomocytes",
"6"="Pharyngeal epithelia",
"7"="Vulval precursors",
"8"="Non-seam hypodermis",
"9"="Intestinal/rectal muscle",
"10"="Touch receptor neurons",
"11"="Unclassified neurons",
"12"="flp-1(+) interneurons",
"13"="Canal associated neurons")
plot_cells(cds, group_cells_by="cluster", color_cells_by="assigned_cell_type",group_label_size=4,cell_size=1.5)
手动选择细胞
cds_subset = choose_cells(cds)
#Warning: package ‘shiny’ was built under R version 3.5.3
轨迹推断
Monocle3的目的是在实验中了解细胞是如何通过一个基因表达变化的生物程序进行转化的。每个细胞都可以看作是高维空间中的一个点,每个维描述了不同基因的表达。识别基因表达变化的程序相当于学习细胞在这个空间中遵循的轨迹。然而,分析中维度越多,学习轨迹就越困难。
幸运的是,许多基因通常彼此共存,因此可以使用各种不同的算法来降低数据的维数。Monocle3通过reduce_dimension提供了两种不同的降维算法(UMAP和tSNE)。两者都使用cell_data_set对象和一些允许用于缩小空间的维度。您还可以提供一个模型公式,指示从数据中“减去”的一些变量(例如批ID或其他技术因素),这样它就不会对轨迹产生影响。
函数learn_graph是继preprocess_cds、reduce_dimensions和cluster_cells之后的轨迹构建过程的第四个步骤。在learn_graph之后,通常调用order_cells。
> cds <- learn_graph(cds)
|===============================================================================================================================================| 100%
|===============================================================================================================================================| 100%
|===============================================================================================================================================| 100%
> heand(colData(cds))
> head(colData(cds))
DataFrame with 6 rows and 11 columns
orig.ident nCount_RNA nFeature_RNA percent.mt RNA_snn_res.0.5 seurat_clusters Size_Factor garnett_cluster cell_type cluster_ext_type assigned_cell_type
AAACATACAACCAC pbmc3k 2419 779 3.01777594047127 1 1 1.10763503821696 NA T cells CD4 T cells Coelomocytes
AAACATTGAGCTAC pbmc3k 4903 1352 3.79359575769937 3 3 2.24503290300858 NA Unknown B cells Unclassified neurons
AAACATTGATCAGC pbmc3k 3147 1129 0.889736256752463 1 1 1.44097869585315 NA CD4 T cells CD4 T cells Seam cells
AAACCGTGCTTCCG pbmc3k 2639 960 1.74308450170519 2 2 1.20837075893119 NA Monocytes Monocytes flp-1(+) interneurons
AAACCGTGTATGCG pbmc3k 980 521 1.22448979591837 6 6 0.44873184681795 NA NK cells NK cells Intestinal/rectal muscle
AAACGCACTGGTAC pbmc3k 2163 781 1.66435506241331 1 1 0.99041529047676 NA T cells CD4 T cells Seam cells
plot_cells(cds,
color_cells_by = "assigned_cell_type",
label_groups_by_cluster=FALSE,
label_leaves=FALSE,
label_branch_points=FALSE,
group_label_size=4,cell_size=1.5)
选择root
#Order the cells in pseudotime
cds = order_cells(cds)
plot_cells(cds,
color_cells_by = "pseudotime",
label_cell_groups=FALSE,
label_leaves=TRUE,
label_branch_points=TRUE,
graph_label_size=1.5,
group_label_size=4,cell_size=1.5)
# a helper function to identify the root principal points:
get_earliest_principal_node <- function(cds, time_bin="Body wall muscle"){
cell_ids <- which(colData(cds)[, "assigned_cell_type"] == time_bin)
closest_vertex <-cds@principal_graph_aux[["UMAP"]]$pr_graph_cell_proj_closest_vertex
closest_vertex <- as.matrix(closest_vertex[colnames(cds), ])
root_pr_nodes <-
igraph::V(principal_graph(cds)[["UMAP"]])$name[as.numeric(names(which.max(table(closest_vertex[cell_ids,]))))]
root_pr_nodes
}
cds = order_cells(cds, root_pr_nodes=get_earliest_principal_node(cds))
plot_cells(cds,
color_cells_by = "pseudotime",
label_cell_groups=FALSE,
label_leaves=FALSE,
label_branch_points=FALSE,
graph_label_size=1.5,
group_label_size=4,cell_size=1.5)
3D 图
#Working with 3D trajectories
cds_3d = reduce_dimension(cds, max_components = 3)
cds_3d = cluster_cells(cds_3d)
cds_3d = learn_graph(cds_3d)
cds_3d = order_cells(cds_3d, root_pr_nodes=get_earliest_principal_node(cds))
cds_3d_plot_obj = plot_cells_3d(cds_3d, color_cells_by="assigned_cell_type")
cds_3d_plot_obj
单个基因的拟时轨迹
AFD_genes = c("S100A9", "RPS27", "FCER1A")
AFD_lineage_cds = cds[AFD_genes,
clusters(cds) %in% c(11, 12, 5)]
plot_genes_in_pseudotime(AFD_lineage_cds,
color_cells_by="pseudotime",
min_expr=0.5)
细胞定义
setwd("D:\\Users\\Administrator\\Desktop\\Novo周运来\\SingleCell\\scrna_tools")
library(garnett)
load("hsPBMC")# 下载好的训练模型
library(org.Hs.eg.db)
cds <- classify_cells(cds, hsPBMC,
db = org.Hs.eg.db,
cluster_extend = TRUE,
cds_gene_id_type = "SYMBOL")
> head(pData(cds))
DataFrame with 6 rows and 11 columns
orig.ident nCount_RNA nFeature_RNA percent.mt RNA_snn_res.0.5 seurat_clusters Size_Factor garnett_cluster cell_type cluster_ext_type assigned_cell_type
AAACATACAACCAC pbmc3k 2419 779 3.01777594047127 1 1 1.10763503821696 NA T cells CD4 T cells Coelomocytes
AAACATTGAGCTAC pbmc3k 4903 1352 3.79359575769937 3 3 2.24503290300858 NA Unknown B cells Unclassified neurons
AAACATTGATCAGC pbmc3k 3147 1129 0.889736256752463 1 1 1.44097869585315 NA CD4 T cells CD4 T cells Seam cells
AAACCGTGCTTCCG pbmc3k 2639 960 1.74308450170519 2 2 1.20837075893119 NA Monocytes Monocytes flp-1(+) interneurons
AAACCGTGTATGCG pbmc3k 980 521 1.22448979591837 6 6 0.44873184681795 NA NK cells NK cells Intestinal/rectal muscle
AAACGCACTGGTAC pbmc3k 2163 781 1.66435506241331 1 1 0.99041529047676 NA T cells CD4 T cells Seam cells
> table(pData(cds)$cell_type)
B cells CD34+ CD4 T cells CD8 T cells Dendritic cells Monocytes NK cells T cells Unknown
297 3 563 147 27 421 252 345 583
plot_cells(cds,
group_cells_by="partition",
color_cells_by="cell_type",
cell_size=1.5,
group_label_size=5)
回归分析
在本节中,我们将探索如何使用Monocle来发现根据几种不同标准表达不同的基因。根据分析的复杂程度,对cell_data_set对象中的所有基因执行差异表达分析可能需要几分钟到几个小时。为了让这个小插曲简单而快速,我们将使用一组小的基因。不过,请放心,Monocle即使在大型实验中也可以分析数千个基因,这对于在您正在研究的生物学过程中发现动态调控基因非常有用。
Monocle中的差异分析工具非常灵活。Monocle的工作原理是为每个基因拟合一个回归模型。您可以指定此模型来考虑实验中的各种因素(时间、治疗等)。例如,在胚胎数据中,细胞是在不同的时间点收集的。我们可以通过先对每个基因拟合一个广义线性模型来测试上述基因的表达是否会随着时间发生变化:
ciliated_genes = top_specific_marker_ids[0:4]
cds_subset = cds[rowData(cds)$gene_short_name %in% ciliated_genes,]
gene_fits = fit_models(cds_subset, model_formula_str = "~seurat_clusters")
# A tibble: 4 x 4
gene_short_name model model_summary status
1 S100A9 OK
2 S100A8 OK
3 RPS27 OK
4 FCER1A OK
gene_fits是一个包含每个基因一行的tibble。模型列包含广义线性模型对象,每个模型对象都旨在使用上面的方程解释基因在细胞间的表达。参数model_formula a_str应该是指定模型公式的字符串。您在测试中使用的模型公式可以包含colData表中作为列存在的任何术语,包括Monocle在其他分析步骤中添加的那些列。例如,如果使用cluster_cells,可以使用cluster或partition(分别)作为模型公式来测试集群和分区之间不同的基因。您还可以包含多个变量,例如~胚。时间+批处理,这对于减去不需要的效果非常有用。
> fit_coefs = coefficient_table(gene_fits)
> fit_coefs
# A tibble: 36 x 10
gene_short_name status term estimate std_err test_val p_value normalized_effect model_component q_value
1 S100A9 OK (Intercept) -1.78 0.196 -9.08 2.07e- 19 0 count 2.07e- 19
2 S100A9 OK seurat_clusters1 0.129 0.295 0.437 6.62e- 1 0.176 count 1.00e+ 0
3 S100A9 OK seurat_clusters2 5.16 0.196 26.3 2.36e-135 7.36 count 7.08e-135
4 S100A9 OK seurat_clusters3 0.0943 0.330 0.286 7.75e- 1 0.129 count 1.00e+ 0
5 S100A9 OK seurat_clusters4 0.00262 0.369 0.0071 9.94e- 1 0.00357 count 9.94e- 1
6 S100A9 OK seurat_clusters5 2.80 0.220 12.7 3.82e- 36 3.96 count 1.15e- 35
7 S100A9 OK seurat_clusters6 -0.223 0.503 -0.443 6.58e- 1 -0.301 count 1.00e+ 0
8 S100A9 OK seurat_clusters7 2.68 0.309 8.68 7.04e- 18 3.79 count 1.41e- 17
9 S100A9 OK seurat_clusters8 1.70 0.621 2.74 6.16e- 3 2.39 count 1.85e- 2
10 S100A8 OK (Intercept) -2.33 0.230 -10.1 1.29e- 23 0 count 2.58e- 23
# ... with 26 more rows
> emb_time_terms = fit_coefs %>% filter(term == "seurat_clusters1")
> emb_time_terms
# A tibble: 4 x 10
gene_short_name status term estimate std_err test_val p_value normalized_effect model_component q_value
1 S100A9 OK seurat_clusters1 0.129 0.295 0.437 6.62e- 1 0.176 count 1.00e+ 0
2 S100A8 OK seurat_clusters1 0.190 0.341 0.558 5.77e- 1 0.251 count 1.00e+ 0
3 RPS27 OK seurat_clusters1 -0.192 0.0174 -11.1 8.74e-28 -0.277 count 3.50e-27
4 FCER1A OK seurat_clusters1 -2.47 1.30 -1.90 5.70e- 2 -1.51 count 1.71e- 1
>
现在,让我们找出那些具有重要时间成分的基因。ent_table()计算每个系数在Wald检验下是否显著不同于零。默认情况下,ent_table()使用Benjamini和Hochberg方法对这些p值进行调整,用于多重假设检验。这些调整值可以在q_value列中找到。我们可以对结果进行过滤,控制假发现率如下:
emb_time_terms = fit_coefs %>% filter(term == "seurat_clusters1")
emb_time_terms %>% filter (q_value < 0.05) %>%
select(gene_short_name, term, q_value, estimate)
plot_genes_violin(cds_subset[,], group_cells_by="cell_type", ncol=2) +
theme(axis.text.x=element_text(angle=45, hjust=1))