Contigs/Scaffolds序列经基因预测、ORF开放阅读框识别(Open Reading fr ame)和蛋白翻译之后,就可以进行功能注释分析了。我们将基因/蛋白序列在特定的数据库中搜索比对,从而完成功能注释分析。常用的功能数据库主要包括KEGG、EggNOG、GO、COG和CAZy等。
功能注释就是我们拿到翻译的蛋白之后,与不同的功能(蛋白)数据库进行对比。至于选择哪种数据库要看研究者的目的以及数据条件(如真核还是原核)。我们选择COG数据库,原因是目前单位还没有相应的流程,在一步步的操作中有助于我们的理解,仅此。
不同的功能数据库可以用来解决具体问题,所以先看看COG是干嘛的,然后是怎样注释的,最后是注释结果的解读。
COG是干嘛的
COG,即Clusters of Orthologous Groups of proteins。可以理解为COG是NCBI的数据库。COG的中文释义即“同源蛋白簇”。COG分为两类,一类是原核生物的,另一类是真核生物。原核生物的一般称为COG数据库;真核生物的一般称为KOG数据库。由NCBI创建并维护的蛋白数据库,根据细菌、藻类和真核生物完整基因组的编码蛋白系统进化关系分类构建而成。通过比对可以将某个蛋白序列注释到某一个COG中,每一簇COG由直系同源序列构成,从而可以推测该序列的功能。COG数据库按照功能一共可以分为二十六类。
蛋白质直系同源簇
- 保守
- 相似的结构和生物学功能(关键性调控蛋白)
- 重现物种的进化历史
其网址主页为:COG
其FTP站点为:COG
COG注释作用:1. 通过已知蛋白对未知序列进行功能注释; 2. 通过查看指定的COG编号对应的protein数目,存在及缺失,从而能推导特定的代谢途径是否存在; 3. 每个COG编号是一类蛋白,将query序列和比对上的COG编号的proteins进行多序列比对,能确定保守位点,分析其进化关系。当然,这里我们说的是第一种作用。
NCBI COG的数据库主要更新历史
- 从 1997 年 第一个公布版本,7个完整基因组,720个COG分类, 包含原核基因组和单细胞真核基因组(酵母),2003 年和2014 年进行了版本升级,最后只保留了细菌和古菌,包含了711个基因组以及4,631个COG分类, 26个功能分类。
- 2013 年构建真核分支COG(KOG, Eukaryotic orthologous groups);
- 2007 年构建古菌分支COG(arCOG, Archaeal Clusters of Orthologous Genes),2012 年和2014 年arCOG进一步升级,arCOG比较适合用于古菌基因组注释;
- 2011 年构建Phage分支COG(POG,phage orthologous groups),2013 年进行了升级;
由于计算资源需求,NCBI COG 构建了不同系统分类分支的COG簇,比如arCOG,KOG, POG等,推荐使用这些分支对新测序基因组进行注释,其实eggNOG 尤其是4.x版本也使用了clade特异的聚类模式。
其实,eggNOG(版本 4.5.1+) 数据库对 NCBI COG 进行扩展,并包含了真核生物信息, 版本 3.0 被广泛应用于(元)基因组数据分析, 另外一个类似数据是 STRING (版本 10.0) 也可以实现COG/KOG的功能注释,主要特色是蛋白质相互作用注释,eggNOG 升级到了 4.0 以后提供基于HMM隐马尔可夫谱的分析,并提供了更细致的 OG 分析,可根据物种所属的clade选择参考数据集,可以有效的降低计算量,另一个特色就是提供了 GO以及其它注释信息(KEGG/COG/SMART)关联分析。
从2003年至2014年NCBI COG一直未更新,EMBL EggNOG(evolutionary genealogy of genes: Non-supervised Orthologous Groups)继承了NCBI COG的衣钵,极大的扩展了基因组信息。 4.5.1 版本, 把包含了2,031个基因组, 其中 352病毒基因组, 190k个直系同源家族。如果做了eggNOG功能注释还有必要做COG注释吗?
eggNOG 数据库包含了丰富的注释信息,除了COG/KOG/NOG的分类和注释信息外,还包含了KEGG/GO/SMART/PFAM信息。
新版本的EggNOG 还提供了自动化注释工具eggnog-mapper,可很方便的完成基因组的功能注释,注释信息可以关联COG/KOG/KEGG/GO/BiGG等。
怎样注释的
既然EggNOG数据库比COG数据库更新更全面,那我们还讲COG干嘛?要知道,COG老是老了一点,那是人家经典。而且目前两者都可以用同一种比对软件来注释,所以不影响阅读。序列决定结构,结构决定功能。功能注释本质是目标蛋白序列同功能蛋白序列数据库的比对过程。
宏基因组数据比对神器 DIAMOND(double index alignment of
next-generation sequencing data))
2015年nature methods上发布了一款新的比对软件DIAMOND,是一款新的用于短DNA测序reads与蛋白参考数据库比对的工具。以Illumina的100~150 bp的reads为例,在快速模式下,DIAMOND比对速度比BLASTX要快20,000倍,可以报告BLASTX发现的80-90%的比对数据,e-value至多为1e-5。如果使用灵敏模式,DIAMOND的比对速度也要比BLASTX快2,500倍,可以报告超过94%的比对数据。
1)使用DIAMOND软件将 Unigenes 与各功能数据库进行比对(blastp,evalue ≤ 1e-5)
2)比对结果过滤:对于每一条序列的 比对结果,选取 score 最高的比对结果(one HSP > 60 bits)进行后续分析
Function/DIAMOND/diamond blastp -q Unigenes_50.fa -d database/COG/cog_clean.fa -t COG/blastout -p 4 -e 1e-5 -k 50 --sensitive -o Unigenes_50.fa.m8
- double indexing
- spaced seeds
比对结果,m8格式12列对应的含义依次是:
Query id:查询序列ID标识
Subject id:比对上的目标序列ID标识
% identity:序列比对的一致性百分比
alignment length:符合比对的比对区域的长度
mismatches:比对区域的错配数
gap openings:比对区域的gap数目
q. start:比对区域在查询序列(Query id)上的起始位点
q. end:比对区域在查询序列(Query id)上的终止位点
s. start:比对区域在目标序列(Subject id)上的起始位点
s. end:比对区域在目标序列(Subject id)上的终止位点
e-value:比对结果的期望值,解释是大概多少次随即比对才能出现一次这个score,Evalue越小,表明这种情况,从概率上越不可能发生,但是现在发生了,所以这个比对具有很重要的意义
bit score:比对结果的bit score值
功能层级:
INFORMATION STORAGE AND PROCESSING
[J] Translation, ribosomal structure and biogenesis
[A] RNA processing and modification
[K] Transcription
[L] Replication, recombination and repair
[B] Chromatin structure and dynamics
CELLULAR PROCESSES AND SIGNALING
[D] Cell cycle control, cell division, chromosome partitioning
[Y] Nuclear structure
[V] Defense mechanisms
[T] Signal transduction mechanisms
[M] Cell wall/membrane/envelope biogenesis
[N] Cell motility
[Z] Cytoskeleton
[W] Extracellular structures
[U] Intracellular trafficking, secretion, and vesicular transport
[O] Posttranslational modification, protein turnover, chaperones
[X] Mobilome: prophages, transposons
METABOLISM
[C] Energy production and conversion
[G] Carbohydrate transport and metabolism
[E] Amino acid transport and metabolism
[F] Nucleotide transport and metabolism
[H] Coenzyme transport and metabolism
[I] Lipid transport and metabolism
[P] Inorganic ion transport and metabolism
[Q] Secondary metabolites biosynthesis, transport and catabolism
POORLY CHARACTERIZED
[R] General function prediction only
[S] Function unknown
3)从比对结果出发,统计不同功能层级的相对丰度(各功能层级的相对丰度等于注释为该功能层级的基因的相对丰度之和),其中,KEGG 数据库划分为 5 个层级,eggNOG 数据库划分为 3 个层级,CAZy 数据库划分为 3 个层级,各数据库的详细划分层级如下所示:
4)从功能注释结果及基因丰度表出发,获得各个样品在各个分类层级上的基因数目表,对于某个功能在某个样品中的基因数目,等于在注释为该功能的基因中,丰度不为 0 的基因数目;
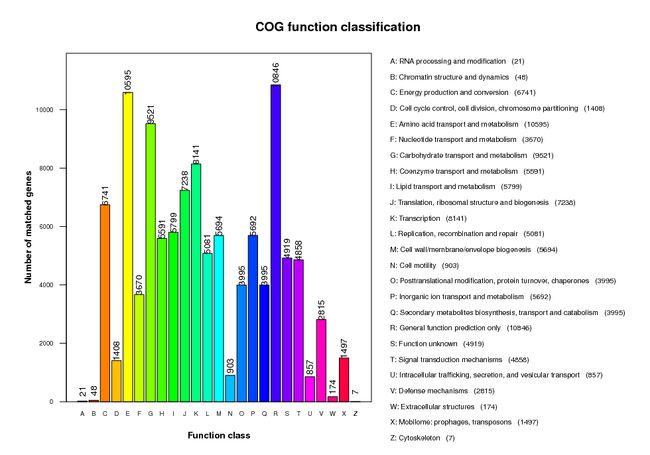
说明 横坐标表示COG功能类型,纵坐标表示注释上的基因个数。
5)从各个分类层级上的丰度表出发,进行注释基因数目统计,相对丰度概况展示,丰度聚类热图展示,PCA和NMDS降维分析,基于功能丰度的Anosim组间(内)差异分析,代谢通路比较分析,组间功能差异的Metastat和LEfSe分析。
宏基因组学那些事之数据库与软件
每日一生信--COG注释(终结版)
diamod安装及使用说明阅读笔记
序列功能注释神器:eggNOG-mapper,KEGG/COG/KOG/GO/BiGG 一网打尽
诺禾致源宏基因组结题报告
诺禾致源扩增子结题报告