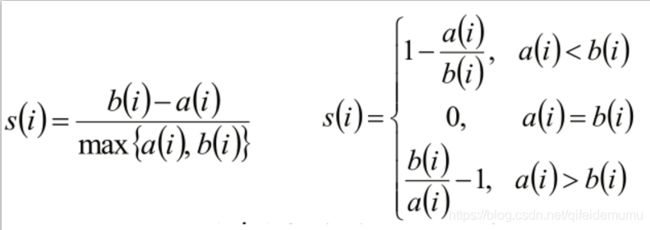k-means算法(DBSCAN算法),聚类算法
一、K-means算法是最经典的聚类算法(无监督学习),
本文对scikit-learn中的kmeans进行说明,以便以后使用。
首先:k-means算法的优劣如下
优势:简单,快速,适合常规数据集
劣势:K值难确定,复杂度与样本呈线性关系,很难发现任意形状的簇(针对凸数据集效果较好:
在欧氏空间中,凸集是对于集合内的每一对点,连接该对点的直线段上的每个点也在该集合内。凹集则不满足)
要使用kmeans算法的话,首先需要进行import:
from sklearn.cluster import KMeansscikit-learn中,通过KMeans进行对象的新建,并传入算法参数进行参数设置。
KMeans传参详解:
1、n_clusters : k值,聚类中心数量(开始时需要产生的聚类中心数量),默认为8
2、max_iter : 算法运行的最大迭代次数,默认300,凸数据集不用管这个数,凹数据集需要指定。
3、tol: 容忍的最小误差,当误差小于tol就会退出迭代(算法中会依赖数据本身),默认为1e-4
4、n_init : (用不同的初始化之心运行计算的次数)k-means算法会随机运行n_init次,最终的结果将是最好的一个聚类结果,默认10
5、init : 即初始值(质心)选择的方式,有三个选择{
优化过的'k-means++', ,一般默认'k-means++' ,
完全随机选择'random': 随机选择k个实例作为聚类中心
自己指定的初始化质心,ndarray:如果传入为矩阵(ndarray),则将该矩阵中的每一行作为聚类中心
初始化过程如下:
从输入的数据点集合(要求有k个聚类)中随机选择一个点作为第一个聚类中心;(2)、对于数据集中的每一个点x,计算它与最近聚类中心(指已选择的聚类中心)的距离D(x);(3)、选择一个新的数据点作为新的聚类中心,选择的原则是:D(x)较大的点,被选取作为聚类中心的概率较大;(4)、重复2和3直到k个聚类中心被选出来
6、algorithm :可选的K-means距离计算算法, 可选{"auto", "full" or "elkan",default="auto"}
"full":传统的距离计算方式.,支持稀疏数据。
"elkan":使用三角不等式,效率更高,但是目前不支持稀疏数据。1、计算任意两个聚类中心的距离;2当计算x点应该属于哪个聚类中心时,当发现2*S(x,K1)
"auto":当为稀疏矩阵时,采用full,否则elkan。
7、precompute_distances : 是否将数据全部放入内存计算,可选{'auto', True, False},开启时速度更快但是更耗内存.
'auto' : 当n_samples * n_clusters > 12million,不放入内存,否则放入内存,double精度下大概要多用100M的内存
True : 进行预计算
False : 不进行预计算
8、n_jobs : 同时进行计算的核数(并发数),n_jobs用于并行计算每个n_init,如果设置为-1,使用所有CPU,若果设置为1,不并行,也可以自定义个数
9、random_state : 用于随机产生中心的随机序列,指定确切的数字后,可以让每次运行程序,产生的结果都一样
10、verbose : 是否输出详细信息,默认为0,值越大,细节打印越多。
● int:冗长度★ 0:不输出训练过程● 1:偶尔输出● >1:对每个子模型都输出
11、copy_x : 是否直接在原矩阵上进行计算。默认为True,会copy一份进行计算。
新建对象后,常用的方法包括fit、predict、cluster_centers_和labels。fit(X)函数对数据X进行聚类,使用predict方法进行新数据类别的预测,使用cluster_centers_获取聚类中心,使用labels_获取训练数据所属的类别,inertia_获取每个点到聚类中心的距离和。
一维数据的聚类:参考链接
from sklearn.cluster import KMeans
import numpy as np
x = np.random.random(10000)
y = x.reshape(-1,1)
km = KMeans()
km.fit(y)
km.cluster_centers_核心的操作是y = x.reshape(-1,1),含义为将一维数据变成只有1列,行数不知道多少(-1代表根据剩下的维度计算出数组的另外一个shape属性值)。
多维数据聚类样例:
import pandas as pd
from sklearn.cluster import KMeans# 第一步 选定要处理的数据集
beer = pd.read_csv('data.txt', sep=' ')
X = beer[["calories","sodium","alcohol","cost"]]# 第二步 是否需要标准化(归一化),以消除数值之间的差异性,一般都是需要归一化处理的,具体后文会有介绍,归一化的判定标准。
#from sklearn.preprocessing import StandardScaler
#scaler = StandardScaler()
#X_scaled = scaler.fit_transform(X) # 找到数据转换规则,进行归一化处理
#km = KMeans(n_clusters=3).fit(X_scaled)# 得到数据归一化之后的模型
#beer["scaled_cluster"] = km.labels_
#beer.sort_values("scaled_cluster")
km = KMeans(n_clusters=3).fit(X) # 数据没有进行标准化
##########
#至于DBSCAN算法,就此处模型不一样,具体如下
#from sklearn.cluster import DBSCAN
#db = DBSCAN(eps=10, min_samples=2).fit(X)# 第三步,将分类好的数据标签在原数据后新加一列
beer['cluster'] = km.labels_
beer.sort_values('cluster')
km.labels_.value_counts() #统计各个类别的数目
cluster_centers = km.cluster_centers_# 可以查看分类后各个类别数据的基本情况(描述性统计)
beer.groupby("cluster").mean()
centers = beer.groupby("cluster").mean().reset_index()# 两个维度数据的可视化展示
import matplotlib.pyplot as plt
plt.scatter(beer["calories"], beer["alcohol"],c=colors[beer["cluster"]])
plt.scatter(centers.calories, centers.alcohol, linewidths=3, marker='+', s=300, c='black')
plt.xlabel("Calories")
plt.ylabel("Alcohol")#对于有多个维度的数据,想查看聚类之后两两特征之间的关系,便于区分k(n_clusters)的取值,可以使用如下的步骤:
from pandas.tools.plotting import scatter_matrix
scatter_matrix(beer[["calories","sodium","alcohol","cost"]],s=100, alpha=1, c=colors[beer["cluster"]], figsize=(10,10))
plt.suptitle("k=?的数据分类特征关系") # 标题自定义scatter_matrix的参数详解:
1。frame,pandas dataframe对象 ,如beer[["calories","sodium","alcohol","cost"]]
2。alpha, 图像透明度,一般取(0,1]
3。figsize,以英寸为单位的图像大小,一般以元组 (width, height) 形式设置
4。ax,可选一般为none
5。diagonal,必须且只能在{‘hist’, ‘kde’}中选择1个,’hist’表示直方图(Histogram plot),’kde’表示核密度估计(Kernel Density Estimation);该参数是scatter_matrix函数的关键参数
6。marker。Matplotlib可用的标记类型,如’.’,’,’,’o’等
7。density_kwds。(other plotting keyword arguments,可选),与kde相关的字典参数
8。hist_kwds。与hist相关的字典参数
9。range_padding。(float, 可选),图像在x轴、y轴原点附近的留白(padding),该值越大,留白距离越大,图像远离坐标原点
10。kwds。与scatter_matrix函数本身相关的字典参数
11。c。颜色
#第四步 聚类评估,轮廓系数
- 计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇。将ai 称为样本i的簇内不相似度。
- 计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样 本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:
- bi =min{bi1, bi2, ..., bik}
- si接近1,则说明样本i聚类合理
- si接近-1,则说明样本i更应该分类到另外的簇
- 若si 近似为0,则说明样本i在两个簇的边界上。
①是否需要归一化的判定
from sklearn import metrics
score_scaled = metrics.silhouette_score(X,beer.scaled_cluster)
# 传入原始值,跟标准化后的聚类标签
score = metrics.silhouette_score(X,beer.cluster)
# 传入原始值, 跟没有标准化聚类后的聚类标签
print(score_scaled, score)
# 查看标准化前后得分的大小,得分越大,越准确,得分越小越不准确,可以用轮廓系数的的分大小来判定是否进行归一化处理
②K值的判定
scores = []
for k in range(2,20):
labels = KMeans(n_clusters=k).fit(X).labels_
score = metrics.silhouette_score(X, labels)
scores.append(score)
plt.plot(list(range(2,20)), scores)
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Sihouette Score")#通过输出不同的k值的得分,来判定K值的选定
k-medoids(k中心点)
kmeans这种方法虽然快速高效,是大规模数据聚类分析中首选的方法,但是它也有一些短板,比如在数据集中有脏数据时,由于其对每一个类的准则函数为平方误差,当样本数据中出现了不合理的极端值,会导致最终聚类结果产生一定的误差,而本篇将要介绍的K-medoids(中心点)聚类法在削弱异常值的影响上就有着其过人之处。
与K-means算法类似,区别在于中心点的选取,K-means中选取的中心点为当前类中所有点的重心,而K-medoids法选取的中心点为当前cluster中存在的一点,准则函数是当前cluster中所有其他点到该中心点的距离之和最小,这就在一定程度上削弱了异常值的影响,但缺点是计算较为复杂,耗费的计算机时间比K-means多。
具体的算法流程如下:
1.在总体n个样本点中任意选取k个点作为medoids
2.按照与medoids最近的原则,将剩余的n-k个点分配到当前最佳的medoids代表的类中
3.对于第i个类中除对应medoids点外的所有其他点,按顺序计算当其为新的medoids时,准则函数的值,遍历所有可能,选取准则函数最小时对应的点作为新的medoids
4.重复2-3的过程,直到所有的medoids点不再发生变化或已达到设定的最大迭代次数
5.产出最终确定的k个类
在Python中关于K-medoids的第三方算法实在是够冷门,经过笔者一番查找,终于在一个久无人维护的第三方模块pyclust中找到了对应的方法KMedoids(),若要对制定的数据进行聚类,使用格式如下:
KMedoids(n_clusters=n).fit_predict(data),其中data即为将要预测的样本集,下面以具体示例进行展示
from pyclust import KMedoids
import numpy as np
from sklearn.manifold import TSNE
import matplotlib.pyplot as plt
'''构造示例数据集(加入少量脏数据)'''
data1 = np.random.normal(0,0.9,(1000,10))
data2 = np.random.normal(1,0.9,(1000,10))
data3 = np.random.normal(2,0.9,(1000,10))
data4 = np.random.normal(3,0.9,(1000,10))
data5 = np.random.normal(50,0.9,(50,10))
data = np.concatenate((data1,data2,data3,data4,data5))
'''准备可视化需要的降维数据
我们总喜欢能够直观地展示研究结果,聚类也不例外。然而,通常来说输入的特征数是高维的(大于3维),一般难以直接以原特征对聚类结果进行展示。而TSNE提供了一种有效的数据降维方式,让我们可以在2维或者3维的空间中展示聚类结果。
'''
data_TSNE = TSNE(learning_rate=100).fit_transform(data)
'''对不同的k进行试探性K-medoids聚类并可视化'''
plt.figure(figsize=(12,8))
for i in range(2,6):
k = KMedoids(n_clusters=i,distance='euclidean',max_iter=1000).fit_predict(data)
colors = ([['red','blue','black','yellow','green'][i] for i in k])
plt.subplot(219+i)
plt.scatter(data_TSNE[:,0],data_TSNE[:,1],c=colors,s=10)
plt.title('K-medoids Resul of '.format(str(i)))
plt.show()
二、DBSCAN聚类算法(无监督学习)
DBSCAN优缺点分析:
优点:不需要指定簇的个数;可以发现任意形状的簇;擅长找到离群点(-1标识);2个参数就够用
缺点:高维数据有些困难(需要降维);参数难以选择,参数的选择对结果影响巨大;效率慢(数据削减策略)
DBSCAN类的重要参数也分为两类,一类是DBSCAN算法本身的参数,一类是最近邻度量的参数,下面我们对这些参数做一个总结。
1)eps: DBSCAN算法参数,即我们的ϵϵ-邻域的距离阈值,和样本距离超过ϵϵ的样本点不在ϵϵ-邻域内。默认值是0.5.一般需要通过在多组值里面选择一个合适的阈值。eps过大,则更多的点会落在核心对象的ϵϵ-邻域,此时我们的类别数可能会减少, 本来不应该是一类的样本也会被划为一类。反之则类别数可能会增大,本来是一类的样本却被划分开。
2)min_samples: DBSCAN算法参数,即样本点要成为核心对象所需要的ϵϵ-邻域的样本数阈值。默认值是5. 一般需要通过在多组值里面选择一个合适的阈值。通常和eps一起调参。在eps一定的情况下,min_samples过大,则核心对象会过少,此时簇内部分本来是一类的样本可能会被标为噪音点,类别数也会变多。反之min_samples过小的话,则会产生大量的核心对象,可能会导致类别数过少。
3)metric:最近邻距离度量参数。可以使用的距离度量较多,一般来说DBSCAN使用默认的欧式距离(即p=2的闵可夫斯基距离)就可以满足我们的需求。可以使用的距离度量参数有:
a) 欧式距离 “euclidean”: ∑i=1n(xi−yi)2−−−−−−−−−−√∑i=1n(xi−yi)2
b) 曼哈顿距离 “manhattan”: ∑i=1n|xi−yi|∑i=1n|xi−yi|
c) 切比雪夫距离“chebyshev”: max|xi−yi|(i=1,2,...n)max|xi−yi|(i=1,2,...n)
d) 闵可夫斯基距离 “minkowski”: ∑i=1n(|xi−yi|)p−−−−−−−−−−−√p∑i=1n(|xi−yi|)pp p=1为曼哈顿距离, p=2为欧式距离。
e) 带权重闵可夫斯基距离 “wminkowski”: ∑i=1n(w∗|xi−yi|)p−−−−−−−−−−−−−−√p∑i=1n(w∗|xi−yi|)pp 其中w为特征权重
f) 标准化欧式距离 “seuclidean”: 即对于各特征维度做了归一化以后的欧式距离。此时各样本特征维度的均值为0,方差为1.
g) 马氏距离“mahalanobis”:(x−y)TS−1(x−y)−−−−−−−−−−−−−−−√(x−y)TS−1(x−y) 其中,S−1S−1为样本协方差矩阵的逆矩阵。当样本分布独立时, S为单位矩阵,此时马氏距离等同于欧式距离。
还有一些其他不是实数的距离度量,一般在DBSCAN算法用不上,这里也就不列了。
4)algorithm:最近邻搜索算法参数,算法一共有三种,第一种是蛮力实现,第二种是KD树实现,第三种是球树实现。这三种方法在K近邻法(KNN)原理小结中都有讲述,如果不熟悉可以去复习下。对于这个参数,一共有4种可选输入,‘brute’对应第一种蛮力实现,‘kd_tree’对应第二种KD树实现,‘ball_tree’对应第三种的球树实现, ‘auto’则会在上面三种算法中做权衡,选择一个拟合最好的最优算法。需要注意的是,如果输入样本特征是稀疏的时候,无论我们选择哪种算法,最后scikit-learn都会去用蛮力实现‘brute’。个人的经验,一般情况使用默认的 ‘auto’就够了。 如果数据量很大或者特征也很多,用"auto"建树时间可能会很长,效率不高,建议选择KD树实现‘kd_tree’,此时如果发现‘kd_tree’速度比较慢或者已经知道样本分布不是很均匀时,可以尝试用‘ball_tree’。而如果输入样本是稀疏的,无论你选择哪个算法最后实际运行的都是‘brute’。
5)leaf_size:最近邻搜索算法参数,为使用KD树或者球树时, 停止建子树的叶子节点数量的阈值。这个值越小,则生成的KD树或者球树就越大,层数越深,建树时间越长,反之,则生成的KD树或者球树会小,层数较浅,建树时间较短。默认是30. 因为这个值一般只影响算法的运行速度和使用内存大小,因此一般情况下可以不管它。
6) p: 最近邻距离度量参数。只用于闵可夫斯基距离和带权重闵可夫斯基距离中p值的选择,p=1为曼哈顿距离, p=2为欧式距离。如果使用默认的欧式距离不需要管这个参数。
以上就是DBSCAN类的主要参数介绍,其实需要调参的就是两个参数eps和min_samples,这两个值的组合对最终的聚类效果有很大的影响。
参考:https://www.cnblogs.com/pinard/p/6217852.html,
备忘,DBSCAN:label为-1的即为离群点,需要删除掉离群点