学会用ggplot2画堆积图就等于学会画饼图,这样一石二鸟的干货,还不继续往下看吗?
堆积柱状图(或百分比堆积图)与饼图都是我们常用的直观描述数据占比的手段,它们的区别主要就在于一个是用柱子的高低来展示占比,另一个是扇形的大小,其实它们本质相同,只是坐标轴的类型不同。那么在绘图的时候其实也是互通的,通过绘制堆积柱状图后将坐标轴稍微替换一下,就能够将饼图绘制出来。下面详细介绍绘制过程。
本文梗概
- 堆积柱状图的画法
- 如何将堆积图“一键”转换为饼图
预处理 - 创建需要输入的数据框
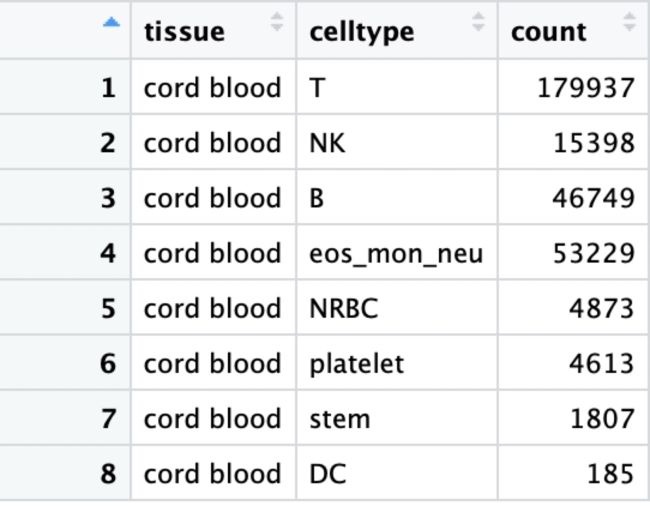
cb = data.frame(tissue = rep("cord blood", 8),
celltype = c('T', 'NK', 'B',
'eos_mon_neu', 'NRBC', 'platelet', 'stem', 'DC'),
count = c(179937, 15398, 46749, 53229,
4873, 4613, 1807, 185))
这个数据框的第一列表示组织类型,第二列celltype是细胞类型,第三列count表示该细胞类型的数目有多少个。
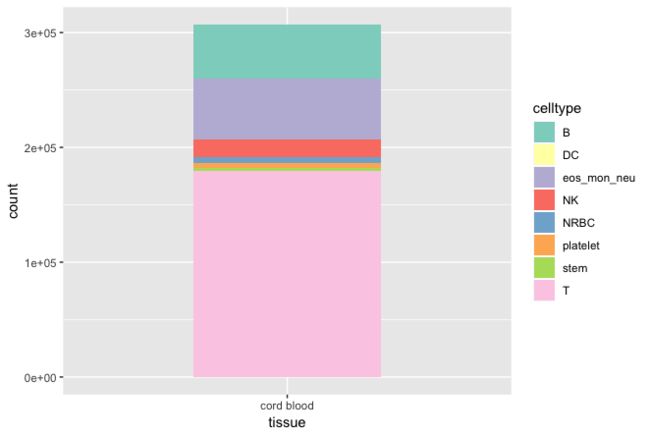
1. 画堆积柱状图
这里注意使用geom_bar(),position = 'stack',就会出现堆积图,柱子的高度指的是比例。另外一种柱状图使用的是geom_col(),但它的高度指的是原始数据的大小,这点需要加以区分。
library(ggplot2)
p = ggplot(cb,aes(x=tissue,y=count,fill=celltype)) +
geom_bar(stat = 'identity', width = 0.5, position = 'stack')
p
现在就画好了最基础的堆积柱状图。我们来美化一下它。
改变配色:
p = p + scale_fill_brewer(palette ="Set3",direction = 1)
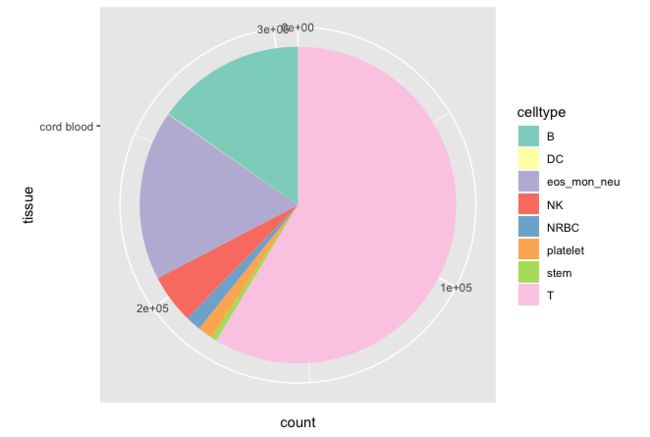
2. 转换成饼图
我们需要做的,就是将上面的堆积柱状图的坐标轴改成极坐标轴:
p = p + coord_polar(theta = 'y', direction = 1)
p
这时候饼图就画出来了。但是这样的图是不能用的,毕竟横纵坐标的“count”和“tissue”都不再适用了,而外圈那些数字也需要去除。
去除横纵坐标标题“tissue”,“count”:
p = p + labs(x="",y="")
p
去除数字和“cord blood”字样:
p = p + theme(axis.text = element_blank())
p
去除左上角那个辣眼睛的小点(其实是刻度):
p = p + theme(axis.ticks = element_blank())
p
将背景设置为透明:
p = p + theme(panel.background = element_rect(I(0)))
p
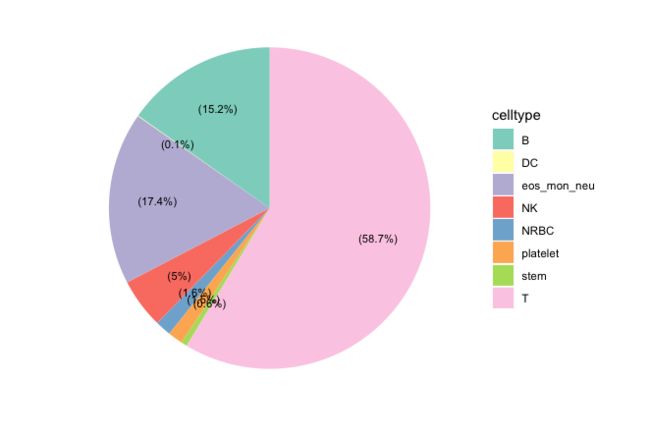
下面我们将这个饼图上面用geom_text注释上每部分的占比,x = 1.1可以调整注释距离圆心的位置,size调整字体大小:
label <- paste0('(', round(cb$count/sum(cb$count) * 100, 1), '%)')
p = p+ geom_text(aes(x=1.1,label=as.character(label)),
position = position_stack(reverse =F,vjust=0.5),size=3)
p