R plot图片背景设置为透明_一文学会网络分析——Cooccurrence网络图在R中的实现...
作者:陈亮 单位:中科院微生物所
编者按:在菌群月坛,在军科院听取王军组陈亮博士分享网络分析的经验,不仅使我对网络的背景知识有了更全面的认识,更使我手上一个关于菌根的课题有极大的启示。这么好的知识,当然希望和大家分享,故约稿陈博士在“宏基因组”发布一下他的经验,感谢陈博士的整理和分享。下面是正文:
网络分析背景知识
近年来,随着计算机技术的发展,网络科学研究在社会网络方面的分析方法已经成熟,从而促进了网络分析方法向其他领域的渗透,例如:信号传导网络、神经网络、代谢通路网络、基因调控网络、生态网络等。
基于图论(Graph theory)的网络科学认为,任何非连续事物之间的关系都可以用网络来表示,通过将互联网内的电脑、社会关系中的个人、生物的基因等不同属性的实体抽象为节点(Node),并用连接(Link)来展示实体之间的关系,通过量化以节点和连接为组件的网络结构指数(Index),从而能够在统一的框架下寻找复杂系统的共性。
目前生态学领域大家用到的网络图多为基于群落数据相关性构建的Co-occurrence网络图。此类网络可以采用R中igraph包构建并实现出图。当然,除此之外,还有一些非命令行的软件,例如cytoscape,gephi,pajek等。但我认为,对于R使用者来说,通过R做图还是最方便的。大致的流程如下图所示:
1)根据观察,实验或者相关性推断来确定物种间的联系。Co-occurrence网络的构建多是基于相关性推断来构建的。常用的相关性推断方法有Pearson,Spearman, Sparcc等方法。
2)通过构建的相关性矩阵或者相互作用列表来构建igraph对象。常用的方法有以下三种,分别由graph_from_incidence_matrix,graph_from_adjacency_matrix,graph_from_edgelist三个函数获得,详细信息参照igraph官方帮助文档。第一种数据格式是普通矩阵,矩阵中数字代表行列所代表的物种间存在联系,这种联系可通过实验或观察来得到。第二种数据格式是邻接矩阵,物种间相关性计算得到的通常为此种形式。第三种为边列表(edgelist),共两列数据,分别代表网络内的节点名称,每一行代表这两个节点间存在着联系。
3)计算网络的各种参数,用以推断网络的性质。
常用网络参数有:
平均路径长度(Average path length):网络中任意两个节点之间的距离的平均值。其反映网络中各个节点间的分离程度。现实网络通常具有“小世界(Small-world)”特性。
聚集系数(Clustering coefficient):分局域聚类系数和全局聚集系数,是反映网络中节点的紧密关系的参数,也称为传递性。整个网络的全局聚集系数C表征了整个网络的平均的“成簇性质”。
介数(Betweenness):网络中不相邻的节点i和j之间的通讯主要依赖于连接节点i和j的最短路径。如果一个节点被许多最短路径经过,则表明该节点在网络中很重要。经过节点n的数量所占比例,介数反映了某节点在通过网络进行信息传输中的重要性。
连接性 (Connectance):网络中物种之间实际发生的相互作用数之和(连接数之和)占总的潜在相互作用数(连接数)的比例,可以反映网络的复杂程度。
此外还包括:度分布(Degree distribution)、平均度(Average degree)、平均介数(Average betweenness)、平均最近邻度(Average nearest-neighbor degree)、直径(Diameter)、介数中心性(Betweenness centralization)和度中心性(Degree centralization)等参数。
各网络参数计算方法及意义参见igraph.org官方帮助文档。
群落数据co-occurrence实例
网络分析需要两个文件,OTU表和OTU的属性;具体格式见测试数据 (后台 回复 网络 获取)
1.最简单的网络图
# 设置工作目录:请修改下方目录或在Rstudio的Session菜单中选择下载测试数据所在的目录
# setwd("~/Downloads/chenliang")
# 安装需要的包,默认不安装,没安装过的请取消如下注释
# install.packages("igraph")
# install.packages("psych")
# 加载包
library(igraph)
library(psych)
# 读取otu-sample矩阵,行为sample,列为otu
otu = read.table("otu_table.txt",head=T,row.names=1)
# 计算OTU间两两相关系数矩阵
# 数据量小时可以用psych包corr.test求相关性矩阵,数据量大时,可应用WGCNA中corAndPvalue, 但p值需要借助其他函数矫正
occor = corr.test(otu,use="pairwise",method="spearman",adjust="fdr",alpha=.05)
occor.r = occor$r # 取相关性矩阵R值
occor.p = occor$p # 取相关性矩阵p值
# 确定物种间存在相互作用关系的阈值,将相关性R矩阵内不符合的数据转换为0
occor.r[occor.p>0.05|abs(occor.r)<0.6] = 0
# 构建igraph对象
igraph = graph_from_adjacency_matrix(occor.r,mode="undirected",weighted=TRUE,diag=FALSE)
igraph
# NOTE:可以设置weighted=NULL,但是此时要注意此函数只能识别相互作用矩阵内正整数,所以应用前请确保矩阵正确。
# 可以按下面命令转换数据
# occor.r[occor.r!=0] = 1
# igraph = graph_from_adjacency_matrix(occor.r,mode="undirected",weighted=NULL,diag=FALSE)
# 是否去掉孤立顶点,根据自己实验而定
# remove isolated nodes,即去掉和所有otu均无相关性的otu 可省略,前期矩阵已处理过
bad.vs = V(igraph)[degree(igraph) == 0]
igraph = delete.vertices(igraph, bad.vs)
igraph
# 将igraph weight属性赋值到igraph.weight
igraph.weight = E(igraph)$weight
# 做图前去掉igraph的weight权重,因为做图时某些layout会受到其影响
E(igraph)$weight = NA
# 简单出图
# 设定随机种子数,后续出图都从同一随机种子数出发,保证前后出图形状相对应
set.seed(123)
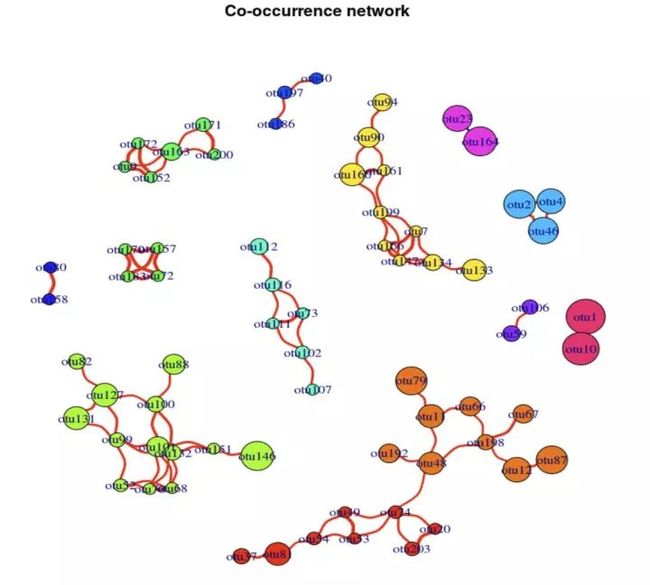
plot(igraph,main="Co-occurrence network",vertex.frame.color=NA,vertex.label=NA,edge.width=1,
vertex.size=5,edge.lty=1,edge.curved=TRUE,margin=c(0,0,0,0))2.按相关类型设置边颜色
# 如果构建网络时,weighted=NULL,此步骤不能统计
sum(igraph.weight>0)# number of postive correlation
sum(igraph.weight<0)# number of negative correlation
# set edge color,postive correlation 设定为red, negative correlation设定为blue
E.color = igraph.weight
E.color = ifelse(E.color>0, "red",ifelse(E.color<0, "blue","grey"))
E(igraph)$color = as.character(E.color)
# 改变edge颜色后出图
set.seed(123)
plot(igraph,main="Co-occurrence network",vertex.frame.color=NA,vertex.label=NA,edge.width=1,
vertex.size=5,edge.lty=1,edge.curved=TRUE,margin=c(0,0,0,0))3.按相关性设置边宽度
# 可以设定edge的宽 度set edge width,例如将相关系数与edge width关联
E(igraph)$width = abs(igraph.weight)*4
# 改变edge宽度后出图
set.seed(123)
plot(igraph,main="Co-occurrence network",vertex.frame.color=NA,vertex.label=NA,
vertex.size=5,edge.lty=1,edge.curved=TRUE,margin=c(0,0,0,0))
边宽度为4倍相关系数绝对值,看看边是不是有粗有细,越粗代表相关绝对值越大
4.设置点的颜色和大小属性对应物种和丰度
# 添加OTU注释信息,如分类单元和丰度
# 另外可以设置vertices size, vertices color来表征更多维度的数据
# 注意otu_pro.txt文件为我随机产生的数据,因此网络图可能不会产生特定的模式或规律。
otu_pro = read.table("otu_pro.txt",head=T,row.names=1)
# set vertices size
igraph.size = otu_pro[V(igraph)$name,] # 筛选对应OTU属性
igraph.size1 = log((igraph.size$abundance)*100) # 原始数据是什么,为什么*100再取e对数
V(igraph)$size = igraph.size1
# set vertices color
igraph.col = otu_pro[V(igraph)$name,]
levels(igraph.col$phylum)
levels(igraph.col$phylum) = c("green","deeppink","deepskyblue","yellow","brown","pink","gray","cyan","peachpuff") # 直接修改levles可以连值全部对应替换
V(igraph)$color = as.character(igraph.col$phylum)
set.seed(123)
plot(igraph,main="Co-occurrence network",vertex.frame.color=NA,vertex.label=NA,
edge.lty=1,edge.curved=TRUE,margin=c(0,0,0,0))5.调整布局样式
# 改变layout,layout有很多,具体查看igraph官方帮助文档。
set.seed(123)
plot(igraph,main="Co-occurrence network",layout=layout_with_kk,vertex.frame.color=NA,vertex.label=NA,
edge.lty=1,edge.curved=TRUE,margin=c(0,0,0,0))
set.seed(123)
plot(igraph,main="Co-occurrence network",layout=layout.fruchterman.reingold,vertex.frame.color=NA,vertex.label=NA,
edge.lty=1,edge.curved=TRUE,margin=c(0,0,0,0))6.按模块着色
# 模块性 modularity
fc = cluster_fast_greedy(igraph,weights =NULL)# cluster_walktrap cluster_edge_betweenness, cluster_fast_greedy, cluster_spinglass
modularity = modularity(igraph,membership(fc))
# 按照模块为节点配色
comps = membership(fc)
colbar = rainbow(max(comps))
V(igraph)$color = colbar[comps]
set.seed(123)
plot(igraph,main="Co-occurrence network",vertex.frame.color=NA,vertex.label=NA,
edge.lty=1,edge.curved=TRUE,margin=c(0,0,0,0))7.显示标签和点轮廓
# 最后添加删除color和label项可显示标签和点颜色边框
plot(igraph,main="Co-occurrence network",vertex.frame.color=NA,vertex.label=NA,
edge.lty=1,edge.curved=TRUE,margin=c(0,0,0,0))8.常用网络属性
# network property
# 边数量 The size of the graph (number of edges)
num.edges = length(E(igraph)) # length(curve_multiple(igraph))
num.edges
# 顶点数量 Order (number of vertices) of a graph
num.vertices = length(V(igraph))# length(diversity(igraph, weights = NULL, vids = V(igraph)))
num.vertices
# 连接数(connectance) 网络中物种之间实际发生的相互作用数之和(连接数之和)占总的潜在相互作用数(连接数)的比例,可以反映网络的复杂程度
connectance = edge_density(igraph,loops=FALSE)# 同 graph.density;loops如果为TRUE,允许自身环(self loops即A--A或B--B)的存在
connectance
# 平均度(Average degree)
average.degree = mean(igraph::degree(igraph))# 或者为2M/N,其中M 和N 分别表示网络的边数和节点数。
average.degree
# 平均路径长度(Average path length)
average.path.length = average.path.length(igraph) # 同mean_distance(igraph) # mean_distance calculates the average path length in a graph
average.path.length
# 直径(Diameter)
diameter = diameter(igraph, directed = FALSE, unconnected = TRUE, weights = NULL)
diameter
# 群连通度 edge connectivity / group adhesion
edge.connectivity = edge_connectivity(igraph)
edge.connectivity
# 聚集系数(Clustering coefficient):分局域聚类系数和全局聚集系数,是反映网络中节点的紧密关系的参数,也称为传递性。整个网络的全局聚集系数C表征了整个网络的平均的“成簇性质”。
clustering.coefficient = transitivity(igraph)
clustering.coefficient
no.clusters = no.clusters(igraph)
no.clusters
# 介数中心性(Betweenness centralization)
centralization.betweenness = centralization.betweenness(igraph)$centralization
centralization.betweenness
# 度中心性(Degree centralization)
centralization.degree = centralization.degree(igraph)$centralization
centralization.degree通过以上的学习,大家是不是可以一步步基于OTU表和注释,用R实现高大上的网络分析和绘制了呢?想要提高,还得多读,多练,多思考,解决科学问题是关键!
编辑:刘永鑫
R统计和作图
Graphpad,经典绘图工具初学初探
在R中赞扬下努力工作的你,奖励一份CheatShet
别人的电子书,你的电子书,都在bookdown
R语言 - 入门环境Rstudio
R语言 - 热图绘制 (heatmap)
R语言 - 基础概念和矩阵操作
R语言 - 热图简化
R语言 - 热图美化
R语言 - 线图绘制
R语言 - 线图一步法
R语言 - 箱线图(小提琴图、抖动图、区域散点图)
R语言 - 箱线图一步法
R语言 - 火山图
R语言 - 富集分析泡泡图
R语言 - 散点图绘制
R语言 - 韦恩图
R语言 - 柱状图
R语言 - 图形设置中英字体
R语言 - 非参数法生存分析
R语言 - 绘制seq logo图
WGCNA分析,简单全面的最新教程
一文看懂PCA主成分分析
富集分析DotPlot,可以服
基因共表达聚类分析和可视化
R中1010个热图绘制方法
还在用PCA降维?快学学大牛最爱的t-SNE算法吧, 附Python/R代码
一个函数抓取代谢组学权威数据库HMDB的所有表格数据
文章用图的修改和排版
network3D: 交互式桑基图
network3D 交互式网络生成
Seq logo 在线绘制工具——Weblogo
生物AI插图素材获取和拼装指导
ggplot2高效实用指南 (可视化脚本、工具、套路、配色)
图像处理R包magick学习笔记
SOM基因表达聚类分析初探
利用gganimate可视化全球范围R-Ladies(R社区性别多样性组织)发展情况
一分钟绘制磷脂双分子层:AI零基础入门和基本图形绘制
AI科研绘图(二):模式图的基本画法
你知道R中的赋值符号箭头(
R语言可视化学习笔记之ggridges包
利用ComplexHeatmap绘制热图(一)
ggplot2学习笔记之图形排列
R包reshape2,轻松实现长、宽数据表格转换
用R在地图上绘制网络图的三种方法
PCA主成分分析实战和可视化 附R代码和测试数据
iTOL快速绘制颜值最高的进化树!
12个ggplot2扩展包帮你实现更强大的可视化
编程模板-R语言脚本写作:最简单的统计与绘图,包安装、命令行参数解析、文件读取、表格和矢量图输出
R语言统计入门课程推荐——生物科学中的数据分析Data Analysis for the Life Sciences
数据可视化基本套路总结
你知道R中的赋值符号箭头
和等号=的区别吗?使用dplyr进行数据操作30例
交集intersect、并集union、找不同setdiff
R包reshape2,轻松实现长、宽数据表格转换
1数据类型(向量、数组、矩阵、 列表和数据框)
2读写数据所需的主要函数、与外部环境交互
3数据筛选——提取对象的子集
4向量、矩阵的数学运算
5控制结构
6函数及作用域
7认识循环函数lapply和sapply
8分解数据框split和查看对象str
9模拟—随机数、抽样、线性模型
1初识ggplot2绘制几何对象
2图层的使用—基础、加标签、注释
3工具箱—误差线、加权数、展示数据分布
4语法基础
5通过图层构建图像
6标度、轴和图例
7定位-分面和坐标系
8主题设置、存储导出
9绘图需要的数据整理技术
创建属于自己的调色板
28个实用绘图包,总有几个适合你
热图绘制
R做线性回归
绘图相关系数矩阵corrplot
相关矩阵可视化ggcorrplot
绘制交互式图形recharts
交互式可视化CanvasXpress
聚类分析factoextra
LDA分析、作图及添加置信-ggord
解决散点图样品标签重叠ggrepel
添加P值或显著性标记ggpubr
Alpha多样性稀释曲线rarefraction curve
堆叠柱状图各成分连线画法:突出组间变化
冲击图展示组间时间序列变化ggalluvial
桑基图riverplot
微生物环境因子分析ggvegan
五彩进化树与热图更配ggtree
多元回归树分析mvpart
随机森林randomForest 分类Classification 回归Regression
加权基因共表达网络分析WGCNA
circlize包绘制circos-plot
R语言搭建炫酷的线上博客系统
往期精品(点击图片直达文字对应教程)
后台回复“生信宝典福利第一波”或点击