遗传算法求解TSP问题
一、导论
演化算法是一类模拟自然界遗传进化规律的仿生学算法,它不是一个具体的算法,而是一个算法簇。遗传算法是演化算法的一个分支,由于遗传算法的整体搜索策略和优化计算是不依赖梯度信息,所以它的应用比较广泛。
本次使用遗传算法(用Python编写)来解决TSP问题。
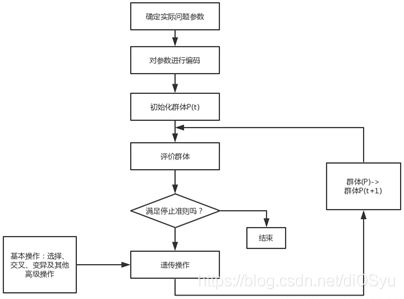
二、算法流程
(1)首先,对城市进行编号0,1,2,3……30。
(2)采用十进制编码:直接用城市的编号进行编码,染色体{0,1,2,……30}表示路径为0-1-2-……-30。
(3)参数设计:种群规模count=300,染色体长度length=31,进化次数epoch_time=2000。
(4)初始化种群:为了加快程序的运行速度,在初始种群的选取中要选取一些较优秀的个体。使用改良圈算法求得一个较好的初始种群。随机生成一个染色体,比如{0,1,2,……30},任意交换两城市顺序位置,如果总距离减小,则更新改变染色体,如此重复,直至不能再做修改。
(5)采用次序交叉法。首先 随机地在双亲中选择两个杂交点,再交换杂交段,其他位置根据双亲城市的相对位置确定。
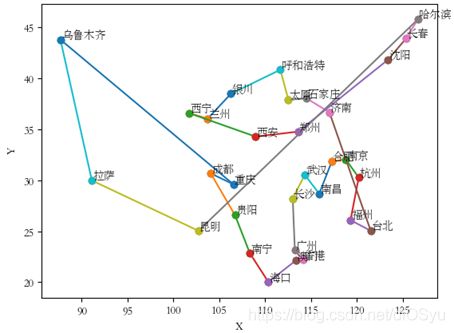
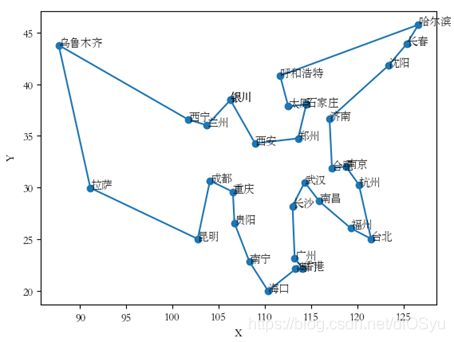
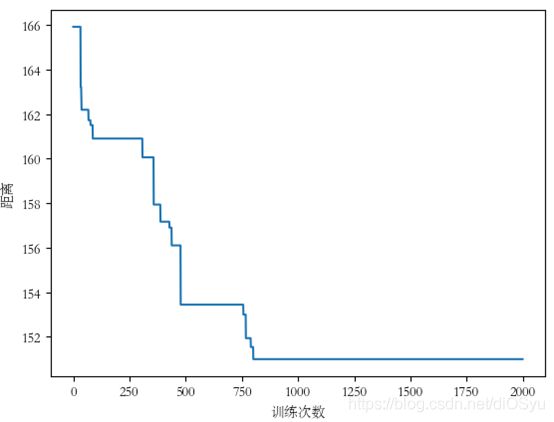
(6)变异也是实现群体多样性的一种手段,同时也是全局寻优的保证。具体设计如下, 按照给定的变异率,对选定变异的个体,随机地取三个整数,满足 1 文件 定位 作用 data.txt 数据文件 存放城市信息 main.py 主程序 程序入口,遗传算法求解TSP问题 data.txt:每一行为一条城市信息,第一列为城市名,第二列、第三列为城市横坐标、纵坐标。一共有31条城市信息。 main.py:主程序 贪心法求解TSP问题(用于与遗传算法比较): 起点为城市0,最短长度为188.23。路径为:0-> 26-> 27-> 5-> 23-> 30-> 29-> 21-> 19-> 20-> 18-> 15-> 14-> 16-> 17-> 22-> 12-> 9-> 10-> 4-> 3-> 24-> 11-> 25-> 13-> 8-> 7-> 6-> 28-> 1-> 2-> 0。 遗传算法求解TSP问题 运行结果1: 起点城市为3,最短路径长度为150.98。路径为:[3, 24, 11, 2, 1, 28, 26, 0, 27, 5, 23, 30, 29, 21, 19, 20, 18, 17, 22, 16, 14, 15, 12, 8, 7, 6, 4, 10, 9, 13, 25, 3]。 该结果为迭代次数为2000次时运行结果。 算法收敛情况如下: 横坐标为遗传算法的训练次数,纵坐标为最短路径长度。 (1)算法评价 算法 优点 缺点 贪心法 计算量小,运行时间较短 答案固定,不能获得更加优秀的解 遗传算法 收敛条件不够明确、计算量大、运行时间长、很多参数的确定需要多次测试 与贪心法相比,可以获得更优的路径解 动态规划 点较少时,运行时间可以接受,可以获得最优解 点数目较多时,运行时间过长 (2)遗传算法分析 1) 遗传算法终止条件: 2) 可以通过分析算法收敛情况,修改迭代次数,减少计算量,提高运行效率。 3) 遗传算法解决TSP问题,收敛情况多种多样。可以通过修改起始点、多次运行获得更优的路径解。 (3)总结 由于遗传算法的整体搜索策略和优化计算是不依赖梯度信息,只需要影响搜索方向的目标函数和相应的适应度函数,所以它的应用比较广泛。 利用遗传算来进行大规模问题的组合优化是一种比较有效的方法,因为在目前的计算上利用枚举法求解最优解有一定的难度。 但是遗传算法也有不足之处,它对算法的精确度、可行度、计算复杂性等方面还没有有效的定量分析方法。 [1] 遗传算法解决TSP问题. Springtostring. https://blog.csdn.net/springtostring/article/details/82429604 [2] 遗传算法. 维基百科. https://zh.wikipedia.org/wiki/遗传算法三、代码清单
重庆,106.54,29.59
拉萨,91.11,29.97
乌鲁木齐,87.68,43.77
银川,106.27,38.47
呼和浩特,111.65,40.82
南宁,108.33,22.84
哈尔滨,126.63,45.75
长春,125.35,43.88
沈阳,123.38,41.8
石家庄,114.48,38.03
太原,112.53,37.87
西宁,101.74,36.56
济南,117,36.65
郑州,113.6,34.76
南京,118.78,32.04
合肥,117.27,31.86
杭州,120.19,30.26
福州,119.3,26.08
南昌,115.89,28.68
长沙,113,28.21
武汉,114.31,30.52
广州,113.23,23.16
台北,121.5,25.05
海口,110.35,20.02
兰州,103.73,36.03
西安,108.95,34.27
成都,104.06,30.67
贵阳,106.71,26.57
昆明,102.73,25.04
香港,114.1,22.2
澳门,113.33,22.13# -*- coding:utf-8 -*-
import numpy as np
import matplotlib.pyplot as plt
import matplotlib
import math
from random import random, randint, shuffle
# 设置字体
matplotlib.rcParams['font.family'] = 'STSong'
# 载入数据
def read_data():
# 城市名
_name = []
# 城市坐标
_condition = []
# 读取数据
with open('data.txt', 'r', encoding='utf-8') as f:
lines = f.readlines()
for line in lines:
line = line.split('\n')[0]
line = line.split(',')
_name.append(line[0])
_condition.append([float(line[1]), float(line[2])])
_condition = np.array(_condition)
return _name, _condition
class TSP:
def __init__(self, _origin):
# 起始点
self.origin = _origin
# 进化次数
self.epochTime = 2000
# 种群数
self.populationCount = 300
# 保持率
self.retainRate = 0.3
# 变异率
self.mutationRate = 0.2
# 改良次数
self.improveCount = 10000
# 读取数据
self.cityNames, self.condition = read_data()
# 城市数目
self.cityCount = len(self.cityNames)
# 生成距离矩阵
self.mat = self.get_distance_matrix()
# 城市索引
self.cityIndex = [_ for _ in range(self.cityCount)]
self.cityIndex.remove(_origin)
# 距离矩阵
def get_distance_matrix(self):
# 使用短变量
_cnt = self.cityCount
_cdt = self.condition
# 生成矩阵
_mat = np.zeros([_cnt, _cnt])
for i in range(_cnt):
for j in range(i, _cnt):
if i == j:
_mat[i][j] = 0
else:
x2 = (_cdt[i][0] - _cdt[j][0]) ** 2
y2 = (_cdt[i][1] - _cdt[j][1]) ** 2
_mat[j][i] = _mat[i][j] = math.sqrt(x2 + y2)
return _mat
# 由路径求TSP路径总长度
def get_total_distance(self, _x):
# 使用短变量
_mat = self.mat
_origin = self.origin
# 计算长度
_dis = 0
_dis += _mat[_origin][_x[0]]
for _i in range(len(_x)):
if _i == len(_x) - 1:
_dis += _mat[_origin][_x[_i]]
else:
_dis += _mat[_x[_i]][_x[_i + 1]]
return _dis
# 生成一些较优秀的个体
def improve(self, _x):
# 改良次数
improve_count = self.improveCount
i = 0
_dis = self.get_total_distance(_x)
while i < improve_count:
u = randint(0, len(_x) - 1)
v = randint(0, len(_x) - 1)
if u != v:
new_x = _x.copy()
t = new_x[u]
new_x[u] = new_x[v]
new_x[v] = t
new_distance = self.get_total_distance(new_x)
if new_distance < _dis:
_dis = new_distance
_x = new_x.copy()
else:
continue
i += 1
return _x
# 自然选择
def selection(self, _population):
# 保持率
retain_rate = self.retainRate
# 对总距离从小到大进行排序
graded = [[self.get_total_distance(_x), _x] for _x in _population]
graded = [_x[1] for _x in sorted(graded)]
# 选出适应性强的染色体
retain_length = int(len(graded) * retain_rate)
_parents = graded[:retain_length]
# 设置弱者的存活概率
random_select_rate = 0.2
# 选出适应性不强,但是幸存的染色体
for chromosome in graded[retain_length:]:
if random() < random_select_rate:
_parents.append(chromosome)
return _parents
# 交叉
def crossover(self, _parents):
# 生成子代的个数,保证种群数目稳定
target_count = self.populationCount - len(_parents)
# 孩子列表
cs = []
while len(cs) < target_count:
male_index = randint(0, len(_parents) - 1)
female_index = randint(0, len(_parents) - 1)
if male_index != female_index:
male = _parents[male_index]
female = _parents[female_index]
left = randint(0, len(male) - 2)
right = randint(left + 1, len(male) - 1)
# 交叉片段
gene1 = male[left:right]
gene2 = female[left:right]
child1_c = male[right:] + male[:right]
child2_c = female[right:] + female[:right]
child1 = child1_c.copy()
child2 = child2_c.copy()
for o in gene2:
child1_c.remove(o)
for o in gene1:
child2_c.remove(o)
child1[left:right] = gene2
child2[left:right] = gene1
child1[right:] = child1_c[0:len(child1) - right]
child1[:left] = child1_c[len(child1) - right:]
child2[right:] = child2_c[0:len(child1) - right]
child2[:left] = child2_c[len(child1) - right:]
cs.append(child1)
cs.append(child2)
return cs
# 变异
def mutation(self, _cs):
# 变异率
mutation_rate = self.mutationRate
# 生成变异后结果
cs = []
for i in range(len(_cs)):
child = _cs[i]
if random() < mutation_rate:
u = randint(1, len(child) - 4)
v = randint(u + 1, len(child) - 3)
w = randint(v + 1, len(child) - 2)
child = child[0:u] + child[v:w] + child[u:v] + child[w:]
cs.append(child)
return cs
# 得到最佳纯输出结果
def get_best_result(self, _population):
graded = [[self.get_total_distance(_x), _x] for _x in _population]
graded = sorted(graded)
return graded[0][0], graded[0][1]
# 进化
def evolution(self):
# 使用短变量
_origin = self.origin
# 初始化种群
population = []
for i in range(self.populationCount):
# 随机生成个体
x = self.cityIndex.copy()
shuffle(x)
# 优化个体
x = self.improve(x)
# 添加
population.append(x)
# 遗传优化
register = []
i = 0
distance, result_path = self.get_best_result(population)
while i < self.epochTime:
# 选择繁殖个体群
parents = self.selection(population)
# 交叉繁殖
cs = self.crossover(parents)
# 变异操作
cs = self.mutation(cs)
# 更新种群
population = parents + cs
distance, result_path = self.get_best_result(population)
register.append(distance)
i += 1
result_path = [_origin] + result_path + [_origin]
# 输出
print(distance)
print(result_path)
print(register)
# 绘图
xs = list()
ys = list()
_names = self.cityNames
_condition = self.condition
for j in result_path:
# 画注解
plt.annotate(_names[j], [_condition[j, 0], _condition[j, 1]])
xs.append(_condition[j, 0])
ys.append(_condition[j, 1])
# 绘图
plt.xlabel('X')
plt.ylabel('Y')
plt.plot(xs, ys, '-o')
plt.show()
plt.xlabel('训练次数')
plt.ylabel('距离')
plt.plot(list(range(len(register))), register)
plt.show()
if __name__ == '__main__':
t1 = TSP(3)
t1.evolution()四、运行结果
五、分析与思考
六、参考文献