用R和BioConductor进行基因芯片数据分析(二):缺失值填充
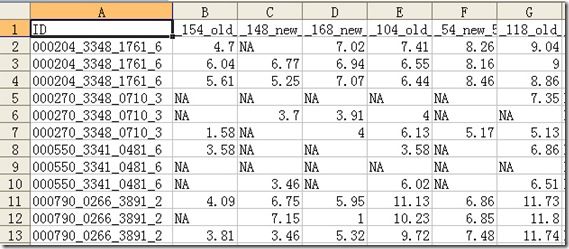
以下分析用到的数据可以在这里(http://dl.getdropbox.com/u/308058/blog/raw_data_3_replicates.txt )下载,这个数据来自关于基因对蝴蝶迁移性的研究,样本是20个蝴蝶个体,其中10个是当地固有个体(old),另外10个是新迁入的个体(new),old和new个体两两随机配对,分别用不同颜色染料(波长分别为555和647nm)标记后,在同一张基因芯片上杂交;此外,每个基因在每张芯片上都重复点样3次,因此此数据是有3个replicates及10张芯片的双通道芯片。数据是样点的信号强度值,没有经过标准化处理的。
拿到数据你会看到许多”NA”,这是因为我把缺失的空白值替换成NA了, 以便用R进行缺失值填充。
说到缺失值填充,通常有3种方法:
A. 用此基因的平均表达值填充;如果有多张重复芯片,可以取不同芯片上的平均值;对于时间序列芯片,可以通过插值法填充。此方法很简单,也比较常用,但是效果不及下面2种方法
B. 基于SVD(即单值分解)方法的填充:简单地讲,此方法是通过描述基因表达谱的几个基本模式来对缺失值进行填充。
C. 基于KNN(最近邻)方法的填充: 此方法是寻找和有缺失值的基因的表达谱相似的其他基因,通过这些基因的表达值(依照表达谱相似性加权)来填充缺失值。KNN法是这3种方法里效果最好的,因此对本数据的缺失值用KNN法填充。
对以上3种方法的比较,这篇paper提供了清晰的说明: Troyanskaya, O., Cantor, M., Sherlock, G., Brown, P., Hastie, T., Tibshirani, R., Botstein, D., and Altman, R. B. (2001), Missing value estimation methods for DNA microarrays, Bioinformatics 17(6):520-525. 其中关于KNN的介绍如下:
The KNN-based method selects genes with expression profiles similar to the gene of interest to impute missing values. If we consider gene A that has one missing value in experiment 1, this method would find K other genes, which have a value present in experiment 1, with expression most similar to A in experiments 2–N (where N is the total number of experiments). A weighted average of values in experiment 1 from the K closest genes is then used as an estimate for the missing value in gene A.
In the weighted average, the contribution of each gene is weighted by similarity of its expression to that of gene A.
下面分析数据
首先要安装 R (http://www.r-project.org/)
然后下载安装叫做impute的package
> source("http://bioconductor.org/biocLite.R")
> biocLite("impute")
impute是专门用KNN法进行缺失值填充的R package:
设置好当前工作目录( Windows是在R的菜单栏->文件->改变工作目录…设置,Linux下用setwd()函数)
然后在R控制台输入以下代码:
library(impute)
#导入impute package
raw<-read.table('raw_data_3_replicates.txt',header=TRUE)
rawexpr<-raw[,-1]
#移除第一列ID列
if(exists(".Random.seed")) rm(.Random.seed)
#必须,如果没有这句话会出错,原因不知-,-请高手指教
imputed<-impute.knn(as.matrix(rawexpr) ,k = 10, rowmax = 0.5, colmax = 0.8, maxp = 1500, rng.seed=362436069)
#impute.knn() 使用一个矩阵作为第一个参数,其他参数这里使用的是默认值
write.table(imputed$data,file='imputed_data.txt')
#write.table() 把数据保存在当前工作目录下的文件中,文件名用file=’ ‘指定,这一步不是必须的
imputeddata<-imputed$data
#imputed$data是在R中储存imputed后的数据的矩阵
现在在R里输入imputed,即填充好的数据矩阵,是不是NA值全都没了?
关于impute package的详细Documentation在http://bioconductor.fhcrc.org/packages/release/bioc/html/impute.html
全部数据文件:http://files.cnblogs.com/emanlee/R_bioconductor_genechip_data_process.zip
from : http://azaleasays.com/tag/r/