3D U-Net论文笔记
3D U-Net论文笔记
原文地址:Learning Dense Volumetric Segmentation from Sparse Annotation
Abstract
本文介绍了一种从稀疏标注的体图像中学习的体分割网络。我们概述了这种方法的两个有吸引力的用例:(1)在半自动设置中,用户在要分割的volume(立方体)中注释一些切片。该网络从这些稀疏注释中学习,并提供密集的3D分割。(2) 在完全自动化的设置中,我们假设存在一个具有代表性的、稀疏注释的训练集。在这个数据集上训练,网络密集地分割新的体积图像。提议的网络通过将所有2D操作替换为3D操作,扩展了Ronneberger等人之前的u-net架构。该实现在训练期间执行动态弹性变形,以有效地增加数据。它是从头开始端到端training的,即不需要预先training的网络。我们在一个复杂的、高度可变的3D结构——爪蟾肾上测试了所提出的方法的性能,并在两个用例中都取得了良好的结果。
Keywords: Convolutional Neural Networks, 3D, Biomedical Volumet-ric Image Segmentation, Xenopus Kidney, Semi-automated, Fully-automated,
Sparse Annotation
**关键词:**卷积神经网络、3D、生物医学体积图像分割、爪蟾肾、半自动、全自动、稀疏注释
1、Introduction
在生物医学数据分析中,体积数据非常丰富。由于计算机屏幕上只能显示二维切片,因此用分段标签标注此类数据会带来困难。因此,以逐片的方式对大量卷进行注释是非常乏味的。由于相邻切片显示的信息几乎相同,因此这种方法也很低效。特别是对于需要大量注释数据的基于学习的方法,三维体积的完整注释并不是创建能够很好推广的大型丰富训练数据集的有效方法。
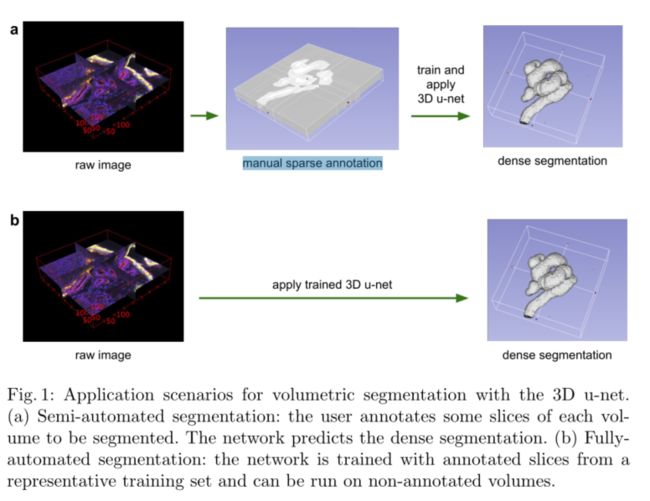
在本文中,我们提出了一种深度网络,它可以学习生成密集的体积分割,但只需要一些带注释的2D切片进行训练。如图1所示,该网络可以以两种不同的方式使用:第一个应用案例的目标是densification(加密/封闭)一个稀疏注释数据集 (注释一部分数据集切片);第二种方法从多个稀疏注释的数据集中学习,以推广到新数据(输入数据不注释,根据具有具有代表性的数据源推断)。这两个案例都非常相关。
该网络基于之前的u-net架构,该架构由收缩编码器部分和连续扩展解码器部分组成,前者用于分析整个图像,后者用于生成全分辨率分割[11]。虽然u-net是一个完全二维的体系结构,但本文提出的网络以三维体作为输入,并用相应的三维操作处理它们,特别是三维卷积、三维最大池和三维上卷积层。此外,我们还避免了网络架构中的瓶颈[13],并使用批处理规范化[4]来加快收敛速度
在许多生物医学应用中,只需要很少的图像就可以训练出一个概括得相当好的网络。这是因为每个图像已经包含了具有相应变化的重复结构。在体积图像中,这种效果更加明显,因此我们可以在两个体积图像上训练一个网络,以便推广到第三个。加权损失函数和特殊的数据扩充使我们能够仅使用少量手动注释的切片(即从稀疏注释的训练数据)来训练网络。
我们展示了该方法在非洲爪蟾肾脏的困难共焦显微数据集上的成功应用。在其发育过程中,非洲爪蟾肾脏形成了一个复杂的结构[7],这限制了预定义参数模型的适用性。首先,我们提供定性结果,以证明几个注释切片的致密化质量。这些结果得到了定量评估的支持。我们还提供了实验,展示了注释切片的数量对网络性能的影响。基于Caffe[5]的网络实现作为OpenSource提供。
https://lmb.informatik.uni-freiburg.de/resources/opensource/unet.en.html
1.1、Related Work
如今,CNNs可以以接近人类表现的精度分割具有挑战性的生物医学2D图像[11,12,3]。由于这一成功,人们已经多次尝试将3D CNN应用于生物医学体积数据。Milleri等人[9]提出了一种CNN结合Hough投票的3D分割方法。然而,他们的方法不是端到端的,只适用于紧凑的水滴状结构。Kleesiek等人[6]的方法是用于3D分割的少数端到端3D CNN方法之一。然而,他们的网络并不深,在第一次卷积之后只有一个最大池;因此,它无法在多个尺度上分析结构。我们的工作是基于2D u-net[11],它在2015年赢得了多个国际细分和跟踪比赛。u-net的体系结构和数据扩充允许仅从少数带注释的样本中学习具有良好泛化性能的模型。它利用了一个事实,即正确应用刚性变换和轻微弹性变形仍然可以产生生物学上合理的图像。向上卷积结构,如用于语义分割的完全卷积网络[8]和u-net,目前还没有广泛推广,我们只知道有一次尝试将这种结构推广到3D[14]。在Tran等人的这项工作中,该体系结构被应用于视频,完整的注释可用于training.。本论文的亮点在于,它可以从零开始在稀疏注释的volumes上进行训练,并且由于其无缝平铺策略,可以在任意大的volumes上工作。
2、Network Architecture
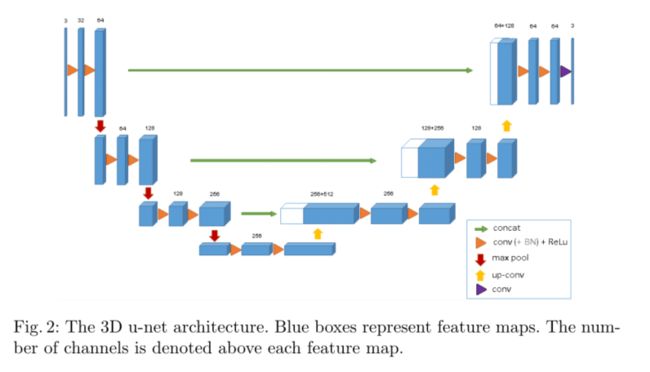
图2展示了网络架构。与标准u-net一样,它有一个分析(下采样)和一个合成(上采样)路径,每个路径有四个解析步骤。在分析路径中,每一层包含两个3×3×3卷积,每个卷积后跟一个校正线性单元(ReLu),然后是一个2×2×2最大池,每个维度的步幅为2。在合成路径中,每一层由一个2×2×2的上卷积和两个3×3×3的卷积组成,每个卷积后面是一个ReLu。分析路径中分辨率相同的图层之间的快捷连接为合成路径。在最后一层中,1×1×1卷积将输出通道的数量减少到标签的数量,在我们的例子中是3。该架构共有19069955个参数。就像[13]中建议的那样,我们通过将最大池之前已经存在的通道数量增加一倍来避免瓶颈。在合成路径中,我们也采用了这种方案。
网络的输入是带有3个通道的图像的132×132×116体素(立体像素)块。我们在最后一层的输出分别是x、y和z方向上的44×44×28个体素。当体素(立体像素)大小为1.76×1.76×2.04µm3时,预测分割中每个体素的近似感受野为155×155×180µm3。因此,每个输出体素都有足够的上下文来有效地学习。
我们还在每个ReLU之前引入批处理规范化(“BN”)。在[4]中,每个批次在训练期间用其平均值和标准偏差进行标准化,并使用这些值更新全局统计数据。这之后是一个层来学习规模和偏见明确。在测试时,通过这些计算出的全局统计数据和学习到的量表和偏差进行归一化。然而,我们的批量只有一个样本,而且样本很少。在这样的应用程序中,在测试时使用当前统计数据效果最好。
该体系结构的重要部分是加权softmax损失函数,它允许我们对稀疏注释进行训练。将未标记像素的权重设置为零,可以仅从标记像素中学习,从而推广到整个体积。
3、Implementation Details
3.1、Data
我们有三个爪蟾肾胚胎样本,处于Nieuwkoop-Faber 36-37期[10]。其中一个如图1(左)所示。3D数据已记录在使用蔡司LSM 510 DUO倒置共焦显微镜,配备平面复消色差40x/1.3油浸物镜,四块瓷砖,三个通道,体素大小为0.88×0.88×1.02µm3。我们使用XuvTools[1]将瓷砖缝成了大体积。第一个通道显示番茄凝集素在488nm激发波长下与荧光素偶联。第二个通道显示405nm激发下DAPI染色的细胞核。第三个通道显示β-连环蛋白,使用564nm激发标记细胞膜的Cy3标记的二级抗体。我们使用Slicer3D(3D标注软件)[2]手动注释了每个volume中的一些正交xy、xz和yz切片(分别三个角度分别进行切片)。根据良好的数据表示选择注释位置,即在所有3维中尽可能均匀地采样注释切片。不同的结构被标记为0:“小管内”;1.“小管”;2:“背景”,3:“未标记”。未标记切片中的所有体素也会得到标签3(“未标记”)。我们在原始分辨率的下采样版本上进行了所有的实验,每个维度都是2倍。因此,在我们的样本1、2和3的x×y×z维度上,实验中使用的数据大小分别为248×244×64、245×244×56和246×244×59。对于样本1、2和3,正交(yz、xz、xy)切片中手动注释切片的数量分别为(7、5、21)、(6、7、12)和(4、5、10)。
3.2、Training
除了旋转、缩放和灰度值增强外,我们还在数据和ground truth labels上应用了平滑的密集变形场。为此,我们在每个方向上间隔32个体素的网格中,从标准偏差为4的正态分布中采样随机向量,然后应用B-spline interpolation。使用带加权cross-entropy loss的softmax对网络输出和ground truth labels进行比较,我们减少了常见背景的权重,增加了内微管的权重,以达到微管和背景体素对损失的平衡影响。标签为3(“未标记”)的体素不参与损失计算,即权重为0(使得网络可以仅从有标记的像素中学习)。我们使用Caffe[5]框架的随机梯度下降求解器进行网络训练。为了能够训练大型3D网络,我们使用了内存高效的cuDNN3卷积层实现。数据扩充是动态完成的,这会产生与训练迭代一样多的不同图像。我们在NVIDIA TitanX GPU上运行了70000次训练迭代,大约花了3天时间。
4、Experiments
4.1 Semi-Automated Segmentation
对于半自动分割,我们假设用户需要对少量体积图像进行完整分割,并且没有先验分割知识。所提出的网络允许用户注释每个体积中的几个切片,并让网络创建密集的体积分割 。
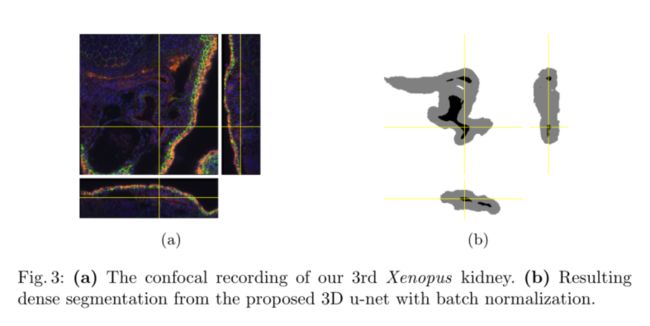
为了进行定性评估,我们在所有三个稀疏注释的样本上对网络进行了训练。图3显示了第三个样本的分割结果。该网络可以从几个带注释的切片中找到整个三维体积分割,并节省了专家进行完整体积注释的时间。
为了评估半自动设置中的定量性能,我们将来自所有3个样本的所有77个手动注释切片统一划分为三个子集,并在有和没有批次标准化的情况下进行3倍交叉验证。为此,我们移除了测试切片,让它们保持未标记状态。这模拟了一个应用程序,其中用户提供了更稀疏的注释。为了测量使用完整3D环境的增益,我们将结果与纯2D实现进行比较,后者将所有带标签的切片视为独立图像。我们在表1中给出了实验结果。(Intersection over Union)联合体上的交点(IoU)被用作精度度量,用于将丢失的ground truth切片与预测的3D体积进行比较。IoU被定义为**true positives/(true positives + false negatives + false positives)**真阳性/(真阳性+假阴性+假阳性)。结果表明,我们的方法已经能够从很少的注释切片中进行推广,从而在几乎不需要注释的情况下实现非常精确的三维分割。
我们还分析了注释切片的数量对网络性能的影响。为此,我们模拟了一个单样本半自动分割。我们开始在每个正交方向上使用1个带注释的切片,并逐渐增加带注释切片的数量。我们在表2中报告了每一个样本(S1、S2和S3)的网络高性能增益,以及每增加几块ground truth(“GT”)切片。结果来自于经过10小时批量标准化训练的网络。为了进行测试,我们使用了本实验任何设置中未使用的切片。
4.2 Fully-automated Segmentation
全自动分割设置假设用户想要分割在类似设置中记录的大量图像。我们进一步假设可以组装一个具有代表性的训练数据集
为了评估这种设置的性能,我们对两个(部分注释的)肾脏体积进行了训练,并使用训练过的网络分割第三个volumes。我们报告了所有3种可能的训练量和测试量组合的结果。表3总结了上一节中遗漏(未使用)的volume.的所有带注释的2D切片的IoU。在这个实验中,BN也改善了结果,但第三种情况除外,在第三种情况下,BN的效果适得其反。我们认为数据集的巨大差异是造成这种影响的原因。全自动分割的典型用例将在更大的样本上工作,其中相同数量的稀疏标签可以很容易地分布在更多的数据集上,以获得更具代表性的训练数据集。
5、Conclusion
我们介绍了一种端到端学习方法,该方法可以半自动和全自动地从稀疏注释中分割三维体积。它为非洲爪蟾肾脏高度可变的结构提供了精确的分割。在半自动装置的3倍交叉验证实验中,我们的平均IoU为0.863。在一个完全自动化的设置中,我们展示了3D架构相对于等效2D实现的性能增益。该网络是从零开始训练的,并且没有以任何方式针对该应用进行优化。我们预计它将适用于许多其他生物医学体积分割任务。它的实现是开源的。