图基准数据集(OGB)
作者论文:https://arxiv.org/abs/2005.00687
作者代码:https://github.com/snap-stanford/ogb
OGB官网:https://ogb.stanford.edu/
目录
0.概述
0.1.目前数据集的问题
0.2 OGB数据集
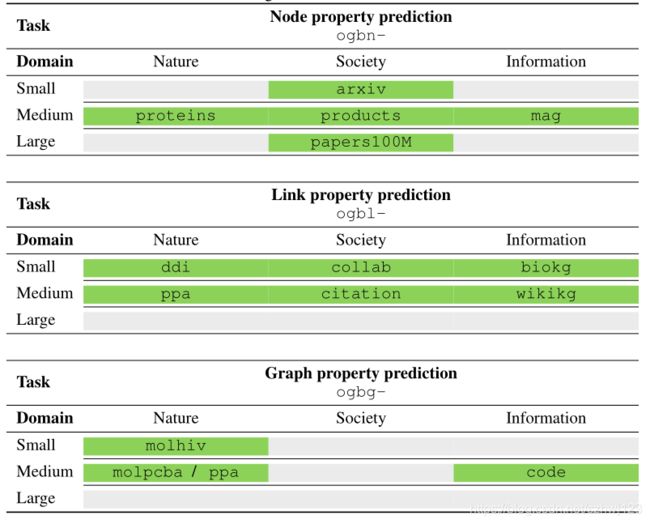
1.OGB节点预测(5个数据集)
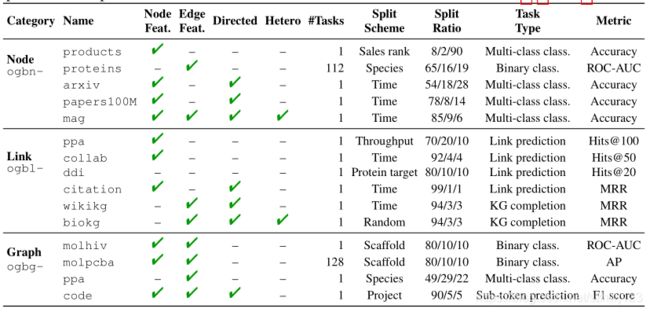
1.1ogbn-products--亚马逊产品联合采购网络
1.2ogbn-papers100M--论文引用网络
1.3ogbn-arxiv--arXiv论文引文网络
1.4ogbn-mag--异构的学术图
1.5 ogbn-proteins--蛋白质相互关系网络
2.OGB链路预测(6个数据集)
2.1 ogbl-ppa --蛋白质关系网络
2.2 ogbl-collab --作家协作网络
2.3 ogbl-ddi --药物相互作用网络
2.4 ogbl-citation --论文引用网络
2.5 ogbl-biokg --异构知识图谱汇编自大量生物医学知识库
2.6 ogbl-wikikg --维基知识图谱
3.OGB图属性预测(4个数据集)
3.1 ogbg-mol* --分子图
3.2 ogbg-ppa --蛋白质关系子图
3.3 ogbg-code --抽象语法树的源代码
0.概述
0.1.目前数据集的问题
规模小,数据集存在质量问题(信息泄露比如直接用标签作为特征,有重复),划分数据集不合理,可能存在分布偏移
0.2 OGB数据集
数据集有小中大三种规模,涵盖领域广,可用于节点预测,链路预测,图属性预测。并且提供了相应的评估标准,加载方式也可适用于pyg,dgl等流行的图神经网络框架(作者代码中都有相应的例子)
以pyg为例
>>> from ogb.graphproppred import PygGraphPropPredDataset
>>> dataset = PygGraphPropPredDataset(name="ogbg-molpcba")
# Pytorch Geometric dataset object
>>> split_idx = dataset.get_idx_split()
# Dictionary containing train/valid/test indices.
>>> train_idx = split_idx["train"]
# torch.tensor storing a list of training indices.#评估方式
>>> from ogb.graphproppred import Evaluator
# Get Evaluator for ogbg-molpcba
>>> evaluator = Evaluator(name = "ogbg-molpcba")
# Learn about the specification of input to the Evaluator.
>>> print(evaluator.expected_input_format)
# Prepare input that follows input spec.
>>> input_dict = {"y_true": y_true, "y_pred": y_pred}
# Get the model performance.
result_dict = evaluator.eval(input_dict)1.OGB节点预测(5个数据集)
1.1ogbn-products--亚马逊产品联合采购网络
无权无向图(ClusterGCN-master)
节点代表产品,边代表2个产品被一起购买。节点特征为产品说明书中的单词,并进行了降维操作。共有47种类型。
数据的分割处理:对产品进行排序,销售量前8%用于训练,接下来的2%用于验证,剩余的用于测试
1.2ogbn-papers100M--论文引用网络
有向图,包括1.11亿篇论文。构造方式与ogbn-arxiv一样。
1.3ogbn-arxiv--arXiv论文引文网络
有向图,表示arxiv上计算机科学论文之间的引用关系。节点表示论文,边表示论文的引用关系,每篇论文都有一个将词在标题和摘要中的嵌入量平均得到128维特征向量。通过 WORD 2 VEC 模型获得嵌入。测试是预测40个关于CS的领域。
分割通过年份分割,2017年之前的用于训练,2018年的用于验证,2019年的用于测试
1.4ogbn-mag--异构的学术图
包含不同的节点类型(论文、作者、机构和主题)及其关系。
它包含四种类型,论文(736,389个节点),作者(1,134,649个节点),机构(8,740个节点)和研究领域(59,965个节点),以及4种关系--一个作者"附属于"一个机构,一个作者"写"一篇论文,一篇论文"引用"一篇论文,一篇论文"的主题是"一个研究领域"。每篇论文由一个128的向量构成。
测试的任务是根据论文的内容、参考文献、作者和作者所属的机构来预测每篇论文的发表会议,共有349个会议。
分割通过年份分割,208年之前的用于训练,2018和2019年的用于验证和测试
1.5 ogbn-proteins--蛋白质相互关系网络
节点表示蛋白质,边表示蛋白质之间不同类型的有生物学意义的联系,边的维度为8维,其中每个维表示单个关联类型的强度,值在0到1之间。
该任务是预测在一个多标签二分类系统中蛋白质功能的存在,在这个系统中总共有112种标签需要预测。(二分类,即共有112个功能,看某个蛋白质是否有这个功能)。
数据集分割。我们根据蛋白质来源的种类将蛋白质节点分成训练/验证/测试集。
2.OGB链路预测(6个数据集)
2.1 ogbl-ppa --蛋白质关系网络
ogbl-ppa数据集是一个无向、无加权的图。节点代表58种不同物种的蛋白质,而边表示蛋白质之间具有生物学意义的联系,如物理相互作用、共表达、同源性或基因组邻近(Szklarczyk等,2019)。
我们提供了一个由训练边构造的图形对象(即不包含验证边和测试边)。每个节点都包含一个58维特征向量,表明了相应蛋白质的来源。
预测任务:任务是预测新的关联边给定训练边。评估是基于一个模型对正样本与负样本的排名情况。具体来说,我们将验证/测试集中的每个正边与随机抽样的300万条负边进行排序,并计算排在第K位或以上的正边的比例
2.2 ogbl-collab --作家协作网络
ogbl-collab数据集是一个无向图,表示作者之间协作网络的一个子集,每个节点代表一个作者,边表示作者之间的协作。所有节点都具有128维的特征,这是通过平均作者发表的论文的词的嵌入度得到的。所有的边都与两种元信息相关联:年份和边的权值,表示当年发表的合著论文的数量。
预测任务。任务是根据过去的合作,预测作者在特定年份的合作关系。评估指标类似于附录5.1中的ogbl-ppa,我们希望模型将真正的合作排名高于虚假的合作。
数据集分割。
为了模拟一个真实的协作推荐应用,我们根据时间对数据进行了分割。
具体来说,我们将2017年之前的合作作为训练边,2018年的合作作为验证边,2019年的合作作为测试边。
2.3 ogbl-ddi --药物相互作用网络
ogbl-ddi数据集是一个同构、无加权、无向图,代表药物-药物相互作用网络,每个节点代表fda批准的或实验药物。边表示药物之间的相互作用,可以解释为一种现象,即两种药物一起服用的联合效果与药物相互独立作用的预期效果有很大不同。
预测任务。这项任务是根据已知的药物相互作用的信息来预测药物相互作用。
2.4 ogbl-citation --论文引用网络
ogbl-引文数据集是一个有向图,表示从MAG中提取的论文子集之间的引文网络,每个节点是一篇具有128维word2VEC特征的论文,这些特征总结了其标题和摘要,每个有向边表示一篇论文引用了另一篇论文。所有节点还附带了表明相应论文发表年份的元信息。
预测任务。任务是根据已有的引文预测缺失的引文。具体来说,对于每一篇源论文,有两篇参考文献被随机删除,我们希望模型将缺失的两篇参考文献排名高于1000篇负参考文献候选文献。
2.5 ogbl-biokg --异构知识图谱汇编自大量生物医学知识库
ogbl-biokg数据集是一个知识图(KG),它是我们使用来自大量生物医学数据存储库的数据创建的。它包含5种实体:疾病(10,687个节点)、蛋白质(17,499个节点)、药物(10,533个节点)、副作用(9,969个节点)和蛋白质功能(45,085个节点)。连接两类实体的定向关系有51种,其中药物-药物相互作用39种,蛋白-蛋白相互作用8种,以及药物-蛋白、药物-副作用、药物-蛋白、功能-功能关系。所有关系都被建模为有向边,其中连接相同实体类型(如蛋白质-蛋白质、药物-药物、功能-功能)的关系总是对称的,即边是双向的。
任务是根据训练三元组预测新的三元组
2.6 ogbl-wikikg --维基知识图谱
从Wikidata知识库中提取的知识图(KG)。它包含一组三元组边缘(头部、关系、尾部)其捕获了世界各实体之间的不同类型的关系。我们检索了Wikidata中的所有关系语句,并过滤掉稀有实体。该KG中包含了2,500,604个实体和535个关系类型。
预测任务:在给定训练边缘的情况下预测新的三元组边缘。
3.OGB图属性预测(4个数据集)
3.1 ogbg-mol* --分子图
Moleculenet: a benchmark for molecular machine learning (论文)
ogbg-molhiv数据集小,ogbg-molpcbaz数据集大
所有的分子都是用rdkit预处理的,每个图表示一个分子,节点表示原子,边表示化学键。输入的节点特征为九维。边的特征为三维,包含键类型,键立体化学以及附加键特征,表明键是否共轭。
任务是尽可能准确地预测目标分子的性质(是否有此性质),对于molhiv用ROC-AUC评估,molpcba用 Average Precision (AP)评估
3.2 ogbg-ppa --蛋白质关系子图
每个蛋白质关联图中的节点表示蛋白质,而边表示蛋白质之间有生物学意义的关联。边缘与7维特征相关联,其中每个元素取0到1之间的值,代表特定类型蛋白质的强度蛋白质关联,如基因共现、基因融合事件和共表达。
预测任务: 给定一个蛋白质关联邻域图,任务是37种方法的多类分类,以预测该图来源于什么分类群。
3.3 ogbg-code --抽象语法树的源代码
ogbg-code数据集是从大约45万个Python方法定义中获得的抽象语法树(ast)集合。
任务是预测组成方法名的子标记,给定由AST表示的Python方法体及其节点特性