实践 | k-means聚类(使用鸢尾花数据集)
使用K-means聚类算法筛实现鸢尾花聚类
- 鸢尾花数据集描述
- 导入相关包
- 直接从sklearn.datasets中加载数据集
- 绘制二维数据分布图
- 实例化K-means类&定义训练函数
- 训练
- 可视化展示
对于给定的样本集,按照样本之间的距离大小,将样本集划分为K个簇,让簇内的点尽量紧密的连在一起,而让簇间的距离尽量的大
鸢尾花数据集描述
- 包含3种类型数据集,共150条数据
- 包含4项特征:花萼长度、花萼宽度、花瓣长度、花瓣宽度
导入相关包
import matplotlib.pyplot as plt
import numpy as np
from sklearn.cluster import KMeans
from sklearn import datasets
直接从sklearn.datasets中加载数据集
# 直接从sklearn中获取数据集
iris = datasets.load_iris()
X = iris.data[:, :4] # 表示我们取特征空间中的4个维度
print(X.shape)
绘制二维数据分布图
每个样本使用两个特征,绘制其二维数据分布图
# 取前两个维度(萼片长度、萼片宽度),绘制数据分布图
plt.scatter(X[:, 0], X[:, 1], c="red", marker='o', label='see')
plt.xlabel('sepal length')
plt.ylabel('sepal width')
plt.legend(loc=2)
plt.show()
实例化K-means类&定义训练函数
def Model(n_clusters):
estimator = KMeans(n_clusters=n_clusters)# 构造聚类器
return estimator
def train(estimator):
estimator.fit(X) # 聚类
训练
# 初始化实例,并开启训练拟合
estimator=Model(3)
train(estimator)
可视化展示
label_pred = estimator.labels_ # 获取聚类标签
# 绘制k-means结果
x0 = X[label_pred == 0]
x1 = X[label_pred == 1]
x2 = X[label_pred == 2]
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o', label='label0')
plt.scatter(x1[:, 0], x1[:, 1], c="green", marker='*', label='label1')
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='+', label='label2')
plt.xlabel('sepal length')
plt.ylabel('sepal width')
plt.legend(loc=2)
plt.show()
# 法一:直接手写实现
# 欧氏距离计算
def distEclud(x,y):
return np.sqrt(np.sum((x-y)**2)) # 计算欧氏距离
# 为给定数据集构建一个包含K个随机质心centroids的集合
def randCent(dataSet,k):
m,n = dataSet.shape #m=150,n=4
centroids = np.zeros((k,n)) #4*4
for i in range(k): # 执行四次
index = int(np.random.uniform(0,m)) # 产生0到150的随机数(在数据集中随机挑一个向量做为质心的初值)
centroids[i,:] = dataSet[index,:] #把对应行的四个维度传给质心的集合
return centroids
# k均值聚类算法
def KMeans(dataSet,k):
m = np.shape(dataSet)[0] #行数150
# 第一列存每个样本属于哪一簇(四个簇)
# 第二列存每个样本的到簇的中心点的误差
clusterAssment = np.mat(np.zeros((m,2)))# .mat()创建150*2的矩阵
clusterChange = True
# 1.初始化质心centroids
centroids = randCent(dataSet,k)#4*4
while clusterChange:
# 样本所属簇不再更新时停止迭代
clusterChange = False
# 遍历所有的样本(行数150)
for i in range(m):
minDist = 100000.0
minIndex = -1
# 遍历所有的质心
#2.找出最近的质心
for j in range(k):
# 计算该样本到4个质心的欧式距离,找到距离最近的那个质心minIndex
distance = distEclud(centroids[j,:],dataSet[i,:])
if distance < minDist:
minDist = distance
minIndex = j
# 3.更新该行样本所属的簇
if clusterAssment[i,0] != minIndex:
clusterChange = True
clusterAssment[i,:] = minIndex,minDist**2
#4.更新质心
for j in range(k):
# np.nonzero(x)返回值不为零的元素的下标,它的返回值是一个长度为x.ndim(x的轴数)的元组
# 元组的每个元素都是一个整数数组,其值为非零元素的下标在对应轴上的值。
# 矩阵名.A 代表将 矩阵转化为array数组类型
# 这里取矩阵clusterAssment所有行的第一列,转为一个array数组,与j(簇类标签值)比较,返回true or false
# 通过np.nonzero产生一个array,其中是对应簇类所有的点的下标值(x个)
# 再用这些下标值求出dataSet数据集中的对应行,保存为pointsInCluster(x*4)
pointsInCluster = dataSet[np.nonzero(clusterAssment[:,0].A == j)[0]] # 获取对应簇类所有的点(x*4)
centroids[j,:] = np.mean(pointsInCluster,axis=0) # 求均值,产生新的质心
# axis=0,那么输出是1行4列,求的是pointsInCluster每一列的平均值,即axis是几,那就表明哪一维度被压缩成1
print("cluster complete")
return centroids,clusterAssment
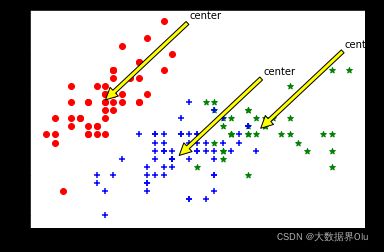
def draw(data,center,assment):
length=len(center)
fig=plt.figure
data1=data[np.nonzero(assment[:,0].A == 0)[0]]
data2=data[np.nonzero(assment[:,0].A == 1)[0]]
data3=data[np.nonzero(assment[:,0].A == 2)[0]]
# 选取前两个维度绘制原始数据的散点图
plt.scatter(data1[:,0],data1[:,1],c="red",marker='o',label='label0')
plt.scatter(data2[:,0],data2[:,1],c="green", marker='*', label='label1')
plt.scatter(data3[:,0],data3[:,1],c="blue", marker='+', label='label2')
# 绘制簇的质心点
for i in range(length):
plt.annotate('center',xy=(center[i,0],center[i,1]),xytext=\
(center[i,0]+1,center[i,1]+1),arrowprops=dict(facecolor='yellow'))
# plt.annotate('center',xy=(center[i,0],center[i,1]),xytext=\
# (center[i,0]+1,center[i,1]+1),arrowprops=dict(facecolor='red'))
plt.show()
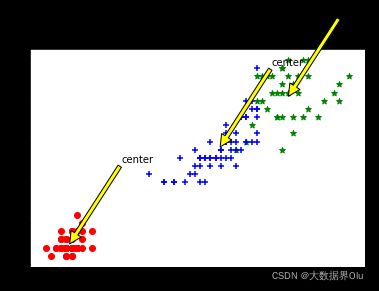
# 选取后两个维度绘制原始数据的散点图
plt.scatter(data1[:,2],data1[:,3],c="red",marker='o',label='label0')
plt.scatter(data2[:,2],data2[:,3],c="green", marker='*', label='label1')
plt.scatter(data3[:,2],data3[:,3],c="blue", marker='+', label='label2')
# 绘制簇的质心点
for i in range(length):
plt.annotate('center',xy=(center[i,2],center[i,3]),xytext=\
(center[i,2]+1,center[i,3]+1),arrowprops=dict(facecolor='yellow'))
plt.show()
dataSet = X
k = 3
centroids,clusterAssment = KMeans(dataSet,k)
draw(dataSet,centroids,clusterAssment)