芯片数据分析笔记【01】 | 基因芯片的基本原理
基因芯片(genechip)(又称DNA芯片、生物芯片)的原型是80年代中期提出的。基因芯片的测序原理是杂交测序方法,即通过与一组已知序列的核酸探针杂交进行核酸序列测定的方法,在一块基片表面固定了序列已知的靶核苷酸的探针。当溶液中带有荧光标记的核酸序列TATGCAATCTAG,与基因芯片上对应位置的核酸探针产生互补匹配时,通过确定荧光强度最强的探针位置,获得一组序列完全互补的探针序列。据此可重组出靶核酸的序列。
1. Affymetrix生物芯片介绍
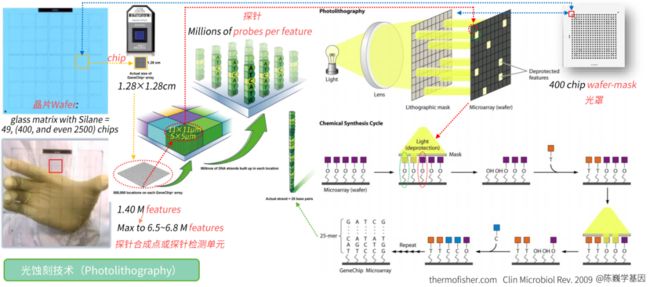
美国Affymetrix公司是目前全球基因芯片行业的领头羊,以其专利的寡聚核苷酸原位光刻合成技术,年产各类寡聚核苷酸基因芯片达到几十万张,占据了表达谱基因芯片科研市场的一半以上,经过了将近十年的研究和开发,已经在国际上赢得了很高的盛誉,同时也成为为数极少的已经盈利的生物芯片公司。Affymetrix公司的基因芯片为寡核苷酸芯片(Oligo芯片),这种类型的芯片具有极高的特异性和灵敏度,重复性好,假阳性率非常低,是目前世界上最先进的基因芯片。Affymetrix所利用的原位光刻专利技术可使一张芯片上合成多达500,000 个寡核苷酸。该系统可以分析样品中DNA或者RNA序列的相对含量。
Affymetrix公司率先开发的寡聚核苷酸原位光刻专利技术,是生产高密度寡核苷酸基因芯片的核心关键技术。采用的技术原理是在合成碱基单体的5'羟基末端连上一个光敏保护基。首先使支持物羟基化,并用光敏保护基团将其保护起来。每次选取适当的蔽光膜(mask)使需要聚合的部位透光,其它部位不透光。这样,光通过蔽光膜照射到支持物上,受光部位的羟基脱保护而活化。因为合成所用的单体分子一端按传统固相合成方法活化,另一端受光敏保护基的保护,所以发生偶联的部位反应后仍旧带有光敏保护基团。因此,每次通过控制蔽光膜的图案(透光与不透光)决定哪些区域应被活化,以及所用单体的种类和反应次序就可以实现在待定位点合成大量预定序列寡聚体的目的。使用多种蔽光膜能以更少的合成步骤生产出高密度的阵列,在合成循环中探针数目呈指数增长。某一含N个核苷酸的寡聚核苷酸,通过4×N个化学步骤能合成出4N个可能结构。例如:一段8个碱基的寡核苷酸有65,536种排列的可能,通过32个化学步骤,8个小时就能合成65,536个探针。其基本原理如图所示:
视频讲解:https://v.qq.com/x/page/j0173fqkgbb.html
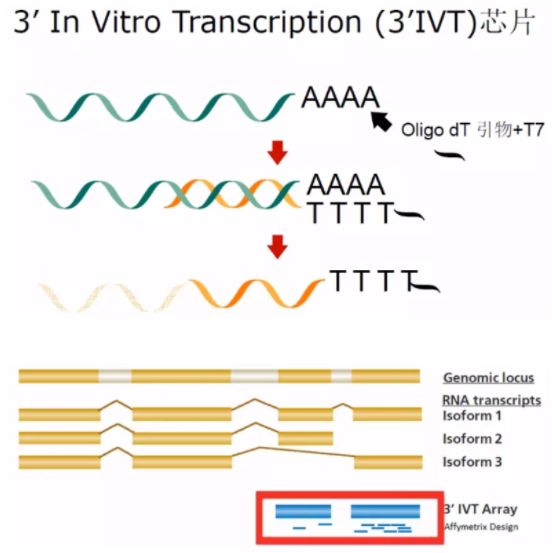
昂飞的表达谱芯片,分成传统的 「In Vitro Transcription」 芯片,简称 IVT 芯片和新一代的 「Whole Transcriptome」 芯片,简称 WT 芯片。
IVT 芯片用 Oligo dT 引物和 T7 逆转录酶来得到 cDNA 链,所以,它得到的 cDNA 主要是靠近 mRNA 3'位末端的 cDNA。
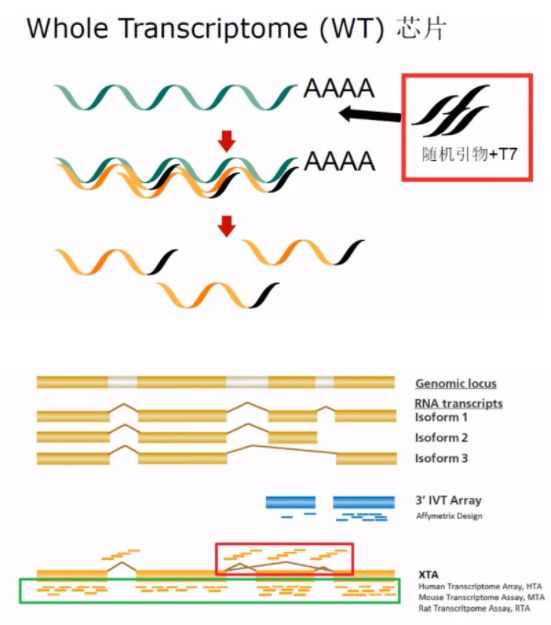
WT 芯片用随机引物和 T7 逆转录酶来得到 cDNA 链,所以其 cDNA 会覆盖转录本上更多区域。
WT 芯片的好处:
可以覆盖转录本上更多的区域,实验结果的代表性就会更强(如图绿框)。
可以针对因为差异剪接所形成的不同转录本分别设计探针,方便得到不同转录本的表达量变化情况(如图红框)。
可以检测到 lncRNA。
比较著名的 WT 芯片有 HTA 2.0、Exon 1.0、Gene 2.0/2.1 等。
参考文章:https://mp.weixin.qq.com/s/OURXVzaQF05ykQEWpXsLGw
2. illumina的原理
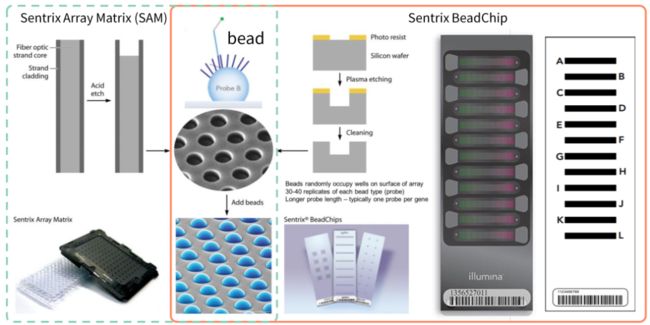
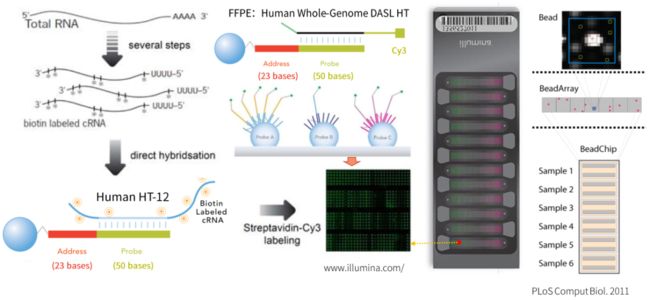
illumina 方案的测序芯片称为「Infinium」(曾用名「Bead Array」),顾名思义由玻璃基片和微珠组成。
玻璃基片形状大小与普通载玻片相近,其作用主要是为了给测序微珠提供容器。
基片内表面,通过光蚀刻的方法,做出许多排列整齐的微米级小孔,作为容纳测序微珠的空间。两者大小恰好匹配,一个小孔正好能容纳一个微珠。
微珠则为芯片的核心部分,每个微珠的表面,都各偶联某种序列的 DNA 片段。每个微珠上,有几十万个片段,而一个珠子上的片段,都是同一种序列。
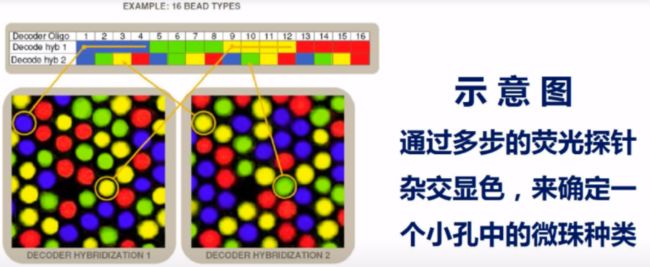
其序列设计如图所示,由靠近微珠端的 Address 序列和 Probe 序列构成。Address 序列是标识微珠的标签序列,通过碱基的排列组合组成该微珠的 ID。在 illumina 生产芯片的过程当中,是把要做芯片的几十万种微珠,按设定的比例或者客户需求进行混合,撒到玻璃基片上。在一张芯片的一个反应当中,每种珠子平均有 15 颗或以上。
微珠随机地落入基片的小孔当中,再通过检测芯片上每个小孔当中的微珠上的 Address 序列,就可以知道,这个小孔当中是哪种微珠。
所以,illumina 公司出厂的每一张芯片,都要跟一个.dmap文件,标注每一张芯片的每一个微孔当中,分别是哪种微珠。自然地,获得芯片相应的地图文件之后,才能解读测序数据。
Probe 序列:作为探针序列,其作用是与目标 DNA 互补杂交;且与 Address 序列存在一一对应的关系。
检测样本的制备和 Affymetrix 芯片基本相似,也是要产生带标记的 cRNA,再和探针杂交,这里我们常用的标记有两种,一种是生物素(biotin)标记,再加荧光结合的抗生物素蛋白与生物素结合即可,见于最常用的 Human HT-12 系列,目前为 V4 版本。
另一种是待检测 cRNA 上直接标记荧光,比如专供石蜡包埋(FFPE)标本的 Human Whole Genome DASL HT 芯片就是这种。大家可以看到,标记了荧光的 cRNA 被探针“毛珠”粘住了,表达量越高的 RNA 被粘住的机会也越多,荧光就越强。这些荧光的点阵正是一个个长满探针排列在硅片孔里的微珠。
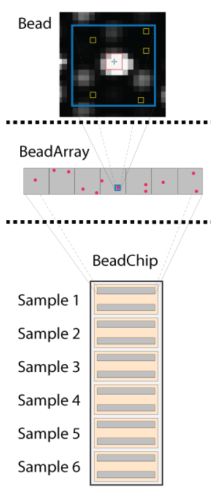
上图显示了 WG-6 BeadChip 芯片中典型珠子(顶部)的示意图,其中标记了阳性(红色正方形)和局部背景(黄色正方形)信号的像素。许多包含 50-mer 探针的微珠位于每个 BeadArray 反应池中。图示为了清晰一些,大大降低了微珠的密度。
视频讲解:https://v.qq.com/x/page/d0171ihr74i.html
参考文章:https://mp.weixin.qq.com/s/7D0TmxWubcMnnaXOtn4bow
3. Agilent 生物芯片原理
Agilent芯片的基片是一个玻璃片。它的大小和一张标准的病理载玻片一样大小。
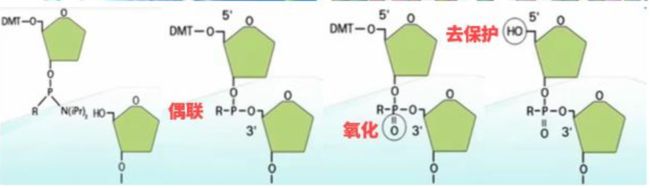
它的芯片制作过程,是用和喷墨打印一样的技术来进行制作的。喷墨打印机,是在墨盒里面是装了“红、黄、蓝、黑”四种颜色的墨水。而Agilent打印生物芯片的墨盒里面,是用带保护基团的A/C/G/T四种碱基底物,来代替了颜色墨水。分别含有4种碱基底物的小液滴,被按照设计的探针序列,依次、层叠地喷到玻璃板的确定的位置上。
在每一个碱基的延伸过程当中都有 3 个步骤,分别是偶联、氧化、 去除保护基团, 即先把一个核苷酸喷到玻璃板上,然后再喷上第二个碱基,让两个碱基之间发生偶联,继而 经过氧化,把亚磷酸基团氧化成磷酸基团。然后,把连在第二个碱基 5’ 位羟基上的 DMT 保 护基团去掉, 留下一个自由的 5’ 位羟基,再进行下一步的延伸反应。不断重复这个过程, DNA 链就会不断地延长。Agilent 的这个 DNA 链合成技术,每一步的合成效率都非常高,可 以达到 99%以上。这让 Agilent 可以在芯片上得到很长的 DNA 链, 最长可以达到 300 个碱基 的长度。
Agilent 表达谱芯片的检测原理也是基于 3’ IVT 原理。首先用 Oligo dT 引物和 T7 逆转录 酶来得到第一链的 cDNA, 然后再转录或复制出第二链的 cDNA,得到双链 cDNA,这个双链 cDNA 是带有 T7 启动子的。接下来, 如果是单色(或单通道)芯片杂交流程,我们只进行这 张图的左边的蓝色部分,使用 T7 逆转录酶,以带有 A、 T、 U 碱基的核苷酸和标有 Cy5 荧光 基团 CTP 为底物逆转录出 cRNA 来;如果是双色(或双通道)杂交流程,要加上图示的右半 边红色的部分,即对于另一个样本(参照组 reference),就用标有 Cy3 荧光基团 CTP 做为 底物之一。这样, Cy3(或 Cy5) 荧光基团就在体外逆转录过程当中,被带入到新合成的 cRNA链当中去了。经纯化后,将这个制备好的 cRNA 与芯片探针进行杂交,在激光扫描仪下检测 每个点的荧光强度,并计算出对应基因的 RNA 表达量来。不同于单色芯片以计算荧光强度作 为表达值,双色芯片的表达值则计算的是实验组和参照组的比值。
参考视频连接:https://v.qq.com/x/page/h0174dobvcb.html
参考文章:https://mp.weixin.qq.com/s/8qUblDoNV3AobGdiwl8Aig
需要注意的是,对于双色芯片,我们称标记第二种颜色的样本为参照组(reference),它可 以是对照组样本(如用同一个患者的癌旁组织作为癌的对照),也可以仅仅是一个基因表达 的参考,如使用一种或几种肿瘤细胞系的 RNA 混合物作为参照组。GEO 上使用 Agilent 双色 芯片的数据集的表达谱,常常给出的是两种基因的表达强度比值,并取 log2;但也有些数 据集虽然用的也是这款芯片,但只使用了单色单样本,这时候就直接和普通单色芯片一样分 析即可。