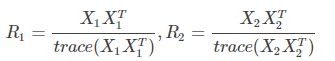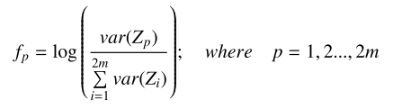matlab csp共空间代码,运动想象中共空间模式算法(CSP)的实现
最近在研究运动想象算法,其中CSP来提取特征用的比较多,尤为是在二分类的问题中,以前写过一篇如何在MNE库中实现CSP算法的博客,用的是MNE库中已经写好的算法,如今想本身实现该算法,研究了几天发现坑仍是比较多的。。。先列出一些参考的博客文章,里面有对应的算法在matlab中的实现,后面主要是解释算法中的一些细节以及本身对该算法的理解。强烈建议下载本博客的资源包对照着看,里面是整理好的博客中用到的资料。html
CSP原理:算法
CSP在Python MNE库中的实现编程
matlab代码实现:函数
上面既然都有实现好的版本了,本着拿来主义的思想原本不打算本身开发的,可是考虑到后面须要支持多种编程语言的实现,以及验证算法的准确性,我在python中也实现了一遍,并尝试理解每一条语句的意义,及其对应的原理。不弄不知道啊,一弄发现了不少有趣的细节。3d
一句话总结CSP算法:共空间模式算法的基本原理是利用矩阵的对角化。找到一组最优空间滤波器进行投影,使两类信号的方差差别化最大,从而获得具备较高区分度的特征向量。
上面所说的特征向量也就至关于滤波器,输入数据通过和特征向量的运算就会获得最大差别化方差的信号,这个信号我理解为是“原始波形”,测量结果=原始波形 * 电极位置,这一点在后面会详细解释。
算法的实现原理在前面的博客中都有所介绍,但愿你们能对照着看,公式的推导这里就再也不赘述,下面主要说一下对应的代码实现中遇到的问题。
问题1 求协方差矩阵
全部的资料第一步都是求对应数据的协方差矩阵,其对应的计算公式为
不知道你们有没有注意到这根本不是协方差矩阵的标准形式,协方差矩阵的计算方法见:https://blog.csdn.net/Mr_HHH/article/details/78490576。协方差的计算应该是去均值后的数据,而且是对列的运算,矩阵X的协方差计算公式为:
那为何在这里会这么计算呢?对行计算这个问题好解释,由于脑波数据通常都是按行存储的,这就是转置的问题。但是为何它不用减去均值,而且也没有求平均。这个问题困扰了我很久,最后在一篇文献中找到了想要的答案。
《Designing optimal spatial filters for single-trial EEG classification in a movement task》文章的796页有以下两段话。
第一段话主要的意思是说,EEG数据是滤波后的,均值为零,看到这恍然大悟,滤波后的数据没有直频份量,均值那可不就是零么,那也就不用减均值了。
第二段话主要的意思是说,正则化是为了消除不一样实验对值的影响,因此使用的是迹。
问题2 特征值排序获取白化矩阵
排序是为了找到影响大的特征向量这个好理解,在文章《Multiclass Common Spatial Pattern for EEG based Brain Computer Interface with Adaptive Learning Classifier》中第3页有提到这个步骤。
上文提到的matlab代码中有一个小问题,代码中直接对特征值进行了排序,我我的的理解是应该对特征值的绝对值排序,由于特征值有可能出现负值的。
![]()
问题3 为何最后提取的特征值是和方差有关的一个量
提取特征的计算公式以下,可为何是方差呢?
这个问题在《Optimizing spatial filters for robust EEG single-trial analysis》一文中有所解释,文章的第4页有以下一段话,大体的意思是说带通滤波后的信号方差和能量有关,虽然仍是不太理解,不过总算找到一些理论依据了。
在上面提到的matlab代码中,在提取特征的时候,还对方差求了一个log:
![]()
我想做者多是借鉴了《Multiclass Common Spatial Pattern for EEG based Brain Computer Interface with Adaptive Learning Classifier》这篇文章,文章第3页中有以下公式:
matlab代码的Python实现
至此流程上算是理解了,参考matlab中的实现,实现了该算法的python版,为了对比计算结果,保留了特征值排序的问题,至关于matlab代码中一比一的实现。
EEG_Channels = trainData.shape[1]
EEG_Trials = trainData.shape[0]
classLabels = np.unique(trainLabels)
EEG_Classes = len(classLabels)
covMatrix = list(range(EEG_Classes))
trialCov = np.zeros([EEG_Channels, EEG_Channels, EEG_Trials])
for i in range(EEG_Trials):
E = trainData[i]
EE = np.dot(E, E.T)
trialCov[:, :, i] = EE/np.trace(EE)
for i in range(EEG_Classes):
covMatrix[i] = np.mean(trialCov[:, :, trainLabels == classLabels[i]], 2)
print('a' ,covMatrix[0])
print(covMatrix[1])
covTotal = covMatrix[0] + covMatrix[1]
#########特征值降序
eigenvalues, featurevectors = np.linalg.eig(covTotal)
egIndex = np.argsort(eigenvalues)[::-1]
eigenvalues = np.sort(eigenvalues)[::-1]
Ut = featurevectors[:, egIndex]
# 矩阵白化
P = np.dot(np.diag(np.sqrt(1/eigenvalues)), Ut.T)
# 矩阵P做用求公共特征向量transformedCov1
transformedCov1 = P.dot(covMatrix[0]).dot(P.T)
# 计算公共特征向量transformedCov1的特征向量和特征矩阵
eigenvalues, featurevectors = np.linalg.eig(transformedCov1)
egIndex = np.argsort(eigenvalues)[::-1]
eigenvalues = np.sort(eigenvalues)[::-1]
U1 = featurevectors[:, egIndex]
# 计算投影矩阵W
CSPMatrix = U1.T.dot(P)
# 计算特征矩阵
FilterPairs = 2
feature_train = np.zeros([EEG_Trials, 2*FilterPairs+1])
feature_test = np.zeros([EEG_Trials, 2*FilterPairs+1])
Filter = np.r_[CSPMatrix[:FilterPairs], CSPMatrix[-FilterPairs:]]
#从每一次实验中提取CSP特征
for t in range(EEG_Trials):
# 将数据投影到CSP空间
projectedTrial_train = Filter.dot(trainData[t])
projectedTrial_test = Filter.dot(trainData[t])
# 投影后信号的方差做为特征
variances_train = np.var(projectedTrial_train, 1)
variances_test = np.var(projectedTrial_test, 1)
for f in range(len(variances_train)):
feature_train[t, f] = np.log(variances_train[f])
for f in range(len(variances_test)):
feature_test[t, f] = np.log(variances_test[f])
CSP_Train_faetures = feature_train[:, 0:4]
CSP_Test_features = feature_test[:, 0:4]
与Python中MNE库的对比
到目前为止算法算是已经实现了,但是这么作对不对呢,MNE库中有写好的CSP算法,对比一下看看结果。不比不知道啊,一比呵呵了,结果彻底不同啊,只好查看MNE库的源码看看了。主要的计算流程在csp.py中的fit函数中,这里截取其中的部分代码进行解释说明。
![]()
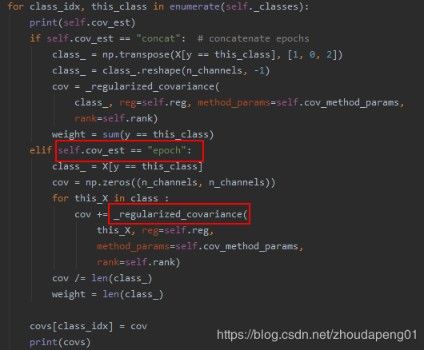
在MNE库中第一步也是求协方差矩阵,这里有两种计算方式“concact”和“epoch”,concact是拼接后再求方差。
对应上面的程序,至关于使用的是epoch方法,
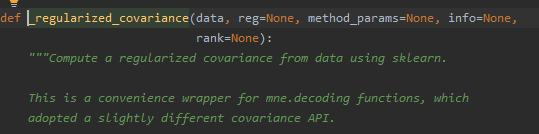
但是这个协方差的计算看起来也很诡异啊,_regularized_covariance是什么鬼,python中有cov函数啊,但是为何这里使用的倒是另外一个函数呢?跳转代码瞅瞅,原来使用的不是标准的协方差计算公式啊。
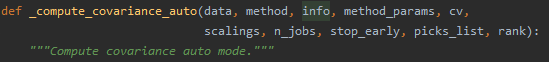
继续追踪,跳转到了_compute_covariance_auto函数,在这个函数中有不少协方差的计算方法,涨知识了协方差还有这么多计算的道道,原来这叫经验协方差。上面的注释中说是使用sklearn中的计算方式,还真特么有。。。。
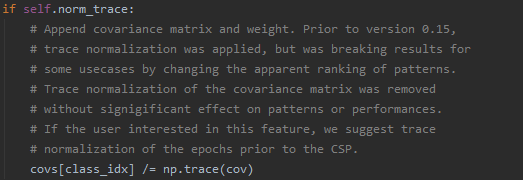
好吧,不纠结了怎么说这也算是协方差,能够接受,继续往下看,看到这部分多少有些安心了,MNE中也提供了利用迹进行正则化的方法,看来整体的思路没错。
下面就是求特征值和特征向量了,这里使用了abs绝对值,也对应了我上面说的特征值排序应该取绝对值。这里为何-0.5就不清楚了。特征提取采用的矩阵相乘,这点前面也解释过,滤波后的数据求协方差不须要减去均值,这里用的也是协方差(能量)的均值做为特征。
不知道你们发现没有这里少了一个步骤就是白化处理,这一点尚未没想明白?难道就是由于它用的协方差计算方法比较牛,而且特征值减去了0.5就不用白化了么?牛皮就是任性啊。。。
patterns和filter的理解
参考说一下我的对这两个参数的理解,测得的数据通过filter滤波后的结果至关因而获得了原始波,而patterns就是源分布信息。
原始波 * 源位置 = 测得的数据
《Optimizing spatial filters for robust EEG single-trial analysis》
《Designing optimal spatial filters for single-trial EEG classification in a movement task》