一文看懂如何用 Python 查看三维数据 (nii.gz格式) 的各种图像参数
Python 如何读取CT/MRI数据的大小,值范围,层厚,分辨率等信息
一文看懂如何用 Python 查看三维数据(nii.gz格式)的各种图像参数
编程环境: jupyter notebook
导入所有安装包
import numpy as np
import nibabel as nib
from ipywidgets import interact, interactive, IntSlider, ToggleButtons
import matplotlib.pyplot as plt
%matplotlib inline
import seaborn as sns
sns.set_style('darkgrid')
- 读取图像
使用 nibabel 库
image_path = "./image/92_v.nii.gz"
image_obj = nib.load(image_path)
print(f'Type of the image {type(image_obj)}')
此时,得到的是 ‘nibabel.nifti1.Nifti1Image’
- 提取 numpy 数组
使用 get_fdata()方法从 image_obj中提取 data
image_data = image_obj.get_fdata()
type(image_data)
$: numpy.ndarray
- 查看图像大小
height, width, depth = image_data.shape
print(f"The image object height: {height}, width:{width}, depth:{depth}")
$: The image object height: 512, width:512, depth:161
- 查看图像值范围
print(f'image value range: [{image_data.min()}, {image_data.max()}]')
$: image value range: [-1024.0, 3071.0]
- 查看图像成像信息
上述中,我们获取了图像矩阵的信息。接下来查看图像成像信息,如 层厚,平面(in-plane)分辨率等
矩阵以外的信息可以通过 image_obj.header 获取
header是键值对,查看 header 包含的所有信息
print(image_obj.header.keys())
其中,最感兴趣的是图像的分辨率 在 pixdim 里面
pixdim = image_obj.header['pixdim']
print(f'z轴分辨率: {pixdim[3]}')
print(f'in plane 分辨率: {pixdim[1]} * {pixdim[2]}')
z轴分辨率: 1.0
in plane 分辨率: 0.568 * 0.568
单位是: mm
我们知道层厚信息,以及矩阵大小,就可以求出实际的扫描范围。
z_range = pixdim[3] * depth
x_range = pixdim[1] * height
y_range = pixdim[2] * width
print(x_range, y_range, z_range)
$: 291.0 291.0 161.0
比如我们要知道z轴方向实际扫了多少mm, 用z轴方向的分辨率 * 矩阵大小。 示例中为 161mm。
- 可视化图像
大家都知道,python是没法直接显示三维图像的,如果非常想要看一下图像什么样,可以一层一层的显示。
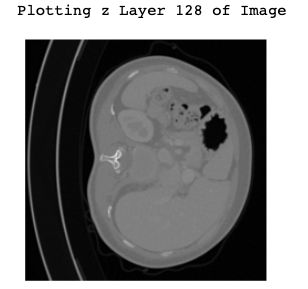
6.1 随机单独显示某一层
maxval = 177
i = np.random.randint(0, maxval)
# Define a channel to look at
print(f"Plotting z Layer {i} of Image")
plt.imshow(image_data[:, :, i], cmap='gray')
plt.axis('off');
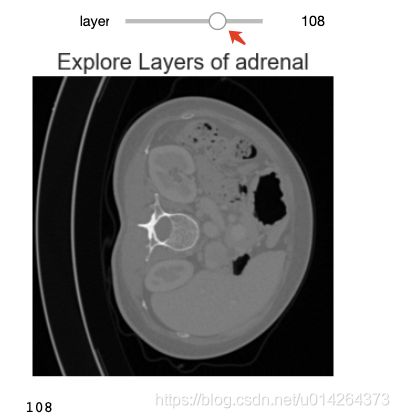
6.2 交互显示
所谓交互显示,实际也是显示某一层,只不过可以通过鼠标拉动,依次显示每一层图像。
def explore_3dimage(layer):
plt.figure(figsize=(10, 5))
plt.imshow(image_data[:, :, layer], cmap='gray');
plt.title('Explore Layers of adrenal', fontsize=20)
plt.axis('off')
return layer
定义一个函数,通过 layer 传参,显示某个层面
interact(explore_3dimage, layer=(0, image_data.shape[-1]));
接下来探索标签
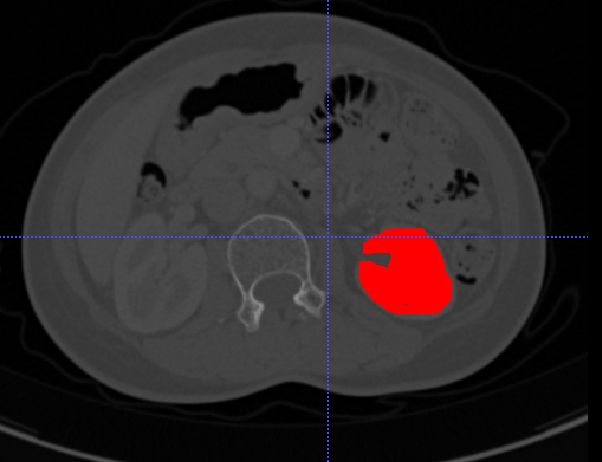
- 获取 label 信息
想读取图像一样读取数据数据
label_path = "../data/adrenal_demo/mask/92_mask.nii.gz"
label_obj = nib.load(label_path)
label_array = label_obj.get_fdata()
7.1 查看 label 里面有几种值
使用 np.unique()
print(f'With the unique values: {np.unique(label_array)}')
$: With the unique values: [0. 1. 2.]
更进一步,查看每个标签对应多少像素
np.unique(label_array, return_counts=True)
$: (array([0., 1., 2.]), array([42179843, 14269, 11072]))
返回独一无二的标签,以及对应的像素数量
怎么样,你学会了,赶紧动手试一试吧。
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连
![]()