机器学习sklearn----KMeans评估指标
引言
前面介绍了KMeans的基础知识。了解了KMeans的基础用法。但是具体需要怎么来评估我们的模型还是一个未知数。今天这节我们会介绍再不考虑实际需求的情况下来评估我们的KMeans模型。实际的应用中是按照我们的需求来评估模型聚类效果的。
数据准备
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
import warnings
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
warnings.filterwarnings("ignore") # 忽略警告
# 创建数据集
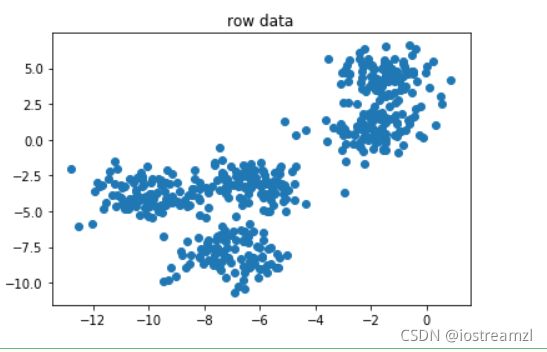
X, y = make_blobs(n_samples = 500, n_features=2, centers=5, random_state=1)
plt.scatter(X[:, 0], X[:, 1])
plt.title("row data")
inertia指标
inertia表示的是每个样本点到其所在质心的距离之和。按照inertia的定义来说inertia是越小越好。 但是实际上我们可以发现当分簇的数量越来越多的时候inertia的值自然越来越小。 甚至再极端的情况下,簇的数量等于样本数的时候,每一个样本就是一个簇,那么inertia就为0。 所以说inertia虽然是一个不错的珩琅标准,单数却很难界定界限在哪里。
示例:查看inertia随n_clusters的变化
# 查看inertia随着n_clusters的变化
for n in range(1, 10) :
km = KMeans(n_clusters=n)
km.fit(X, y)
print("n_clusters: {}\tinertia: {}".format(n, km.inertia_))
# 可以看出inertia的值是越来越小的。但是我们观察原始数据
# 显然分为2-5类是最好的结果,所以这里的inertia作为指标不太适用
轮廓系数silhouette_score
轮廓系数用两个指标来同时评估样本的簇内差异和簇间差异
轮廓系数的计算公式如下:
s = b − a m a x ( a , b ) s = \frac {b - a} {max(a, b)} s=max(a,b)b−a
其中 a 表示样本与它所在的簇之间样本的相似性,等于样本与同一簇之间的其他样本的平均距离
b 表示样本与其他簇的相似新,等于样本与离其最近的簇的所有样本的平均距离
我们希望簇内差异小,簇间差异大。那么就是希望b大于a,且越大越好。
轮廓系数s的取值再-1,1之间,s越靠近1越好
from sklearn.metrics import silhouette_score, silhouette_samples
# silhouette_score 返回所有样本的轮廓系数的均值
# silhouette_samples 返回每个样本的轮廓系数
# inertia和轮廓系数的对比
inertia_scores = []
sil_scores = []
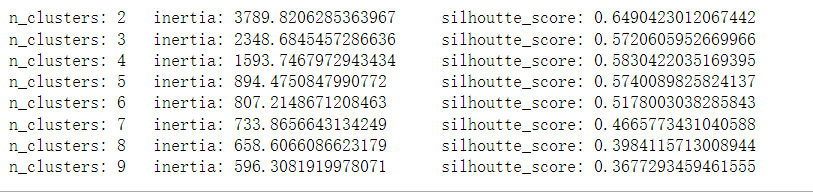
for n in range(2, 10) :
km = KMeans(n_clusters=n).fit(X, y)
inertia_scores.append(km.inertia_)
# 轮廓系数接收的参数中,第二个参数至少有两个分类
sc = silhouette_score(X, km.labels_)
sil_scores.append(sc)
print("n_clusters: {}\tinertia: {}\tsilhoutte_score: {}".format(
n, km.inertia_, sc))