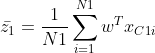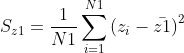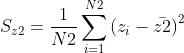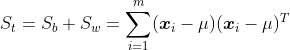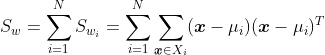线性判别分析(LDA)详解
入门小菜鸟,希望像做笔记记录自己学的东西,也希望能帮助到同样入门的人,更希望大佬们帮忙纠错啦~侵权立删。
目录
一、LDA简介
二、数学原理(以二分类为例子)
1、设定
2、每一类的均值和方差
3、目标函数
4、目标函数的求解
5、最终的实践所求
三、多分类LDA
四、LDA用途与优缺点
1、用途
2、优点
3、缺点
五、LDA的python应用
1、调用函数LinearDiscriminantAnalysis
2、常用参数意义
3、常用返回值
4、利用LDA进行二分类实例
一、LDA简介
LDA(线性判别分析)是一个经典的二分类算法。
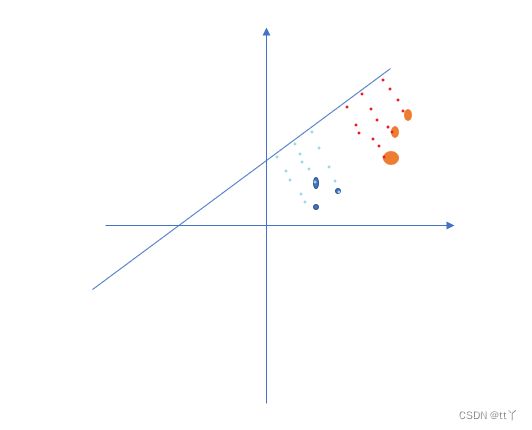
主要思想:以一种基于降维的方式将所有的样本映射到一维坐标轴上,然后设定一个阈值,将样本进行区分
如下图所示,把红蓝两类的点投影在了一条直线(向量a)上,即二维变一维(本来一个点要用(x,y)来表示,投影到直线后就用一个维度来描述)。
二、数学原理(以二分类为例子)
1、设定
首先我们假设整个样本空间分为两个类别,分别是1、-1;N1、N2分别代表1,-1类别样本的个数;样本为X。
那么有:![]() ;
;![]()
设定z为映射后的坐标(即投影后的坐标)
2、每一类的均值和方差
将样本数据X向w向量(设定w的模长为1)做投影,则有:![]()
接下来求出映射后的均值和方差(用来衡量样本的类间距离和类内距离)
3、目标函数
想要得到好的分类模型,即要求类内间距小,类间间距大。即:
类内间距小:![]() ;两个类的方差越小,说明样本越密集
;两个类的方差越小,说明样本越密集
类间间距大:![]() ;用两个类的均值的距离说明两个类之间的距离
;用两个类的均值的距离说明两个类之间的距离
根据这样的思路构建目标函数:
J(w)越大越好,即我们要求的是:![]()
4、目标函数的求解
化简目标函数:(将w向量与原数据的运算分隔开)
令类间散度矩阵:![]() ;类内散度矩阵:
;类内散度矩阵:![]() ,则有:
,则有:
方法一:
为了解决
,则对J(w)求导:
化简得到:
又因为
,
,
都是标量,w前面我们已经约定它的模长为1,所以我们不关心它的长度,只关心他的方向,所以把标量都摘掉,得:
方法二:
J(w)的分子分母都是关于w的二次项,因此J(w)的解与w的长度无关,只与它的方向有关。所以这里为例简单处理也可以令
,故求
,利用拉格朗日乘子法可得:
又因为
方向恒为
,所以令
,因此有
5、最终的实践所求
为得到数值解的稳定性,通常对![]() 进行奇异值分解(
进行奇异值分解(![]() ),再由
),再由![]() 得到
得到![]() 。
。
三、多分类LDA
假定存在N个类,且第i类示例数为![]() 。
。
然后与上面的二分类类似:目标函数为:
类似可得:![]()
所以W的解为![]() 的特征向量组成的矩阵。
的特征向量组成的矩阵。
四、LDA用途与优缺点
1、用途
LDA既可以用来降维(将W视为投影矩阵),又可以用来分类,但主要还是用于降维。
2、优点
与另一个降维算法PCA对比
(1)在降维过程中可以使用类别的先验知识经验,而PCA(无监督学习)无法使用类别先验知识
(2)LDA样本分类依赖的是均值而不是方差,比PCA算法更优
3、缺点
(1)LDA不适合对非高斯分布的样本降维
(2)LDA降维最多降到类别数N-1的维数,如果我们降维的维度大于N-1,则不能使用LDA
(3)LDA可能会过度拟合数据
五、LDA的python应用
1、调用函数LinearDiscriminantAnalysis
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis2、常用参数意义
(1)solver:字符串类型,指定求解最优化问题的算法
'svd':奇异值分解。对于有大规模特征的数据,推荐用这种算法
'lsqr':最小平方差,可以结合skrinkage参数
'eigen' :特征分解算法,可以结合shrinkage参数
(2)skrinkage:取值:字符串‘auto’或者浮点数或者None。
该参数通常在训练样本数量小于特征数量的场合下使用。
‘auto’:自动决定shrinkage参数的大小
None:不使用shrinkage参数
浮点数(位于0~1之间):自己指定的shrinkage参数
(3)n_components:(整数类型)指定了数组降维后的维度(该值必须小于n_classes-1)
(4)priors:一个数组,数组中的元素依次指定了每个类别的先验概率。如果为None,则认为每个类的先验概率都是等可能的
3、常用返回值
coef_:权重向量
intercept:b值
covariance_:一个数组,依次给出了每个类别的协方差矩阵
means_:一个数组,依次给出了每个类别的均值向量
xbar_:给出了整体样本的均值向量
4、利用LDA进行二分类实例
来个简单的小栗子
我们使用sklearn里的乳腺癌数据集
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()然后对数据进行一个处理,让我们看起来舒服点,计算机处理也舒服点
data=cancer["data"]
col = cancer['feature_names']
x = pd.DataFrame(data,columns=col)#就是那些个特征
target = cancer.target.astype(int)
y = pd.DataFrame(target,columns=['target'])#对应特征组合下的类别标签训练集测试集分分类
from sklearn.model_selection import train_test_split
x_train,x_test,y_train,y_test=train_test_split(x,y,test_size=0.3,random_state=1)直接进入训练
clf = LinearDiscriminantAnalysis(n_components=1)
model=clf.fit(x_train,y_train)训练出来的模型对test集进行一个预测
y_pred = model.predict(x_test)
print(classification_report(y_test, y_pred))完整代码
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from sklearn import metrics
import matplotlib.pyplot as plt
from sklearn.metrics import classification_report
import pandas as pd
import warnings
warnings.filterwarnings('ignore')
cancer = load_breast_cancer()
data=cancer["data"]
col = cancer['feature_names']
x = pd.DataFrame(data,columns=col)
target = cancer.target.astype(int)
y = pd.DataFrame(target,columns=['target'])
x_train,x_test,y_train,y_test=train_test_split(x,y,test_size=0.3,random_state=1)
clf = LinearDiscriminantAnalysis(n_components=1)
model=clf.fit(x_train,y_train)
y_pred = model.predict(x_test)
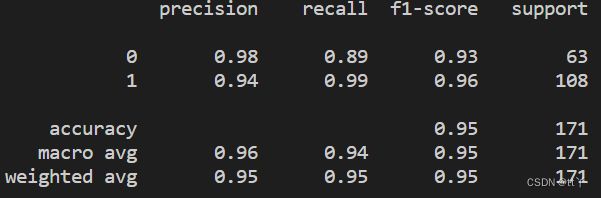
print(classification_report(y_test, y_pred))结果
欢迎大家在评论区批评指正,谢谢~