将批量图片转换成NIFTI(.nii)格式
NIFTI (Neuroimaging Informatics Technology Initiative) 是一种常用的医学图片保存格式(NIfTI-1 Data Format — Neuroimaging Informatics Technology Initiative (nih.gov))。可以用软件FSL (参考:(66条消息) 如何处理.nii文件_暮日落流年的博客-CSDN博客_nii文件用什么打开)和 ITK-SNAP (怎样打开并查看.nii和DICOM格式的医学图像_zuzhiang的博客-CSDN博客_dicom格式怎么打开)进行查看。
本文目的是展示如何将nii格式的文件提取出一张张图片并保存,然后再展示如何将这些保存的图片再转化回nii格式保存。
1. 将nii保存为tif图片
在下面的例子中,nii文件是一个大脑CT图片的图像分割的标签文件,其标签值为0,1,2,4。相应的数据是在Kaggle中的一个项目里下载的(3D MRI Brain tumor segmentation || U-NET | Kaggle)。
import numpy as np
import os
import matplotlib.pyplot as plt
import cv2
import SimpleITK as sitk
# 数据
path = './'
file = 'BraTS20_Training_001_seg.nii'
# 目标路径
save_path = './tif_from_nii'
if not os.path.exists(save_path):
os.mkdir(save_path)
itk_img = sitk.ReadImage(os.path.join(path,file))
data_np = sitk.GetArrayFromImage(itk_img)
# save extracted image
for idx in range(data_np.shape[0]):
fname = str(idx).zfill(3)+'.tif'
# plt.imsave(os.path.join(save_path,fname), data_np[idx]) # 保存后像素0~255
cv2.imwrite(os.path.join(save_path,fname), data_np[idx]) # 像素值为原始值(0,1,2,4)
需要注意的是,应该用cv2.imwrite()来保存图片,使得像素值保留为原始的标签值(0,1,2,4)。如果用plt.imsave()的话,则像素值被转换为(0,255,128,64)。
提取的tif图片如下图所示(因为像素值低,所以看上去是全黑的):
2. 批量将tif图片转换成一个nii文件
上面这一步已经从原始的nii文件中提取了图片并保存为tif格式(应该也可以保存为png,jpg格式,但本人没有尝试过)。下面的代码显示如何进行这种转换:
import SimpleITK as sitk
import os
save_path = './tif_from_nii'
file_list = os.listdir(save_path)
file_list.sort()
file_names = [os.path.join(save_path,f) for f in file_list]
newspacing = [1, 1, 1] # 设置x,y, z方向的空间间隔
reader = sitk.ImageSeriesReader()
reader.SetFileNames(file_names)
vol = reader.Execute()
vol.SetSpacing(newspacing)
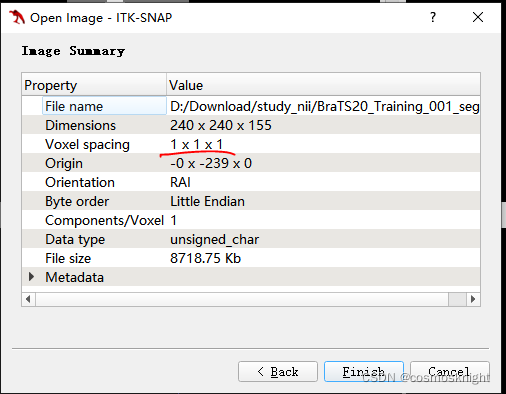
sitk.WriteImage(vol, 'volume.nii') # 保存为volume.nii.gz也可需要说明的是,有必要设置newspacing, 否则,x,y,z方向的间隔不对的话,结果就会和原来的nii文件不同。这里之所以设置为[1,1,1],是我用ITK-SNAP软件查看 'BraTS20_Training_001_seg.nii' 文件得到的:
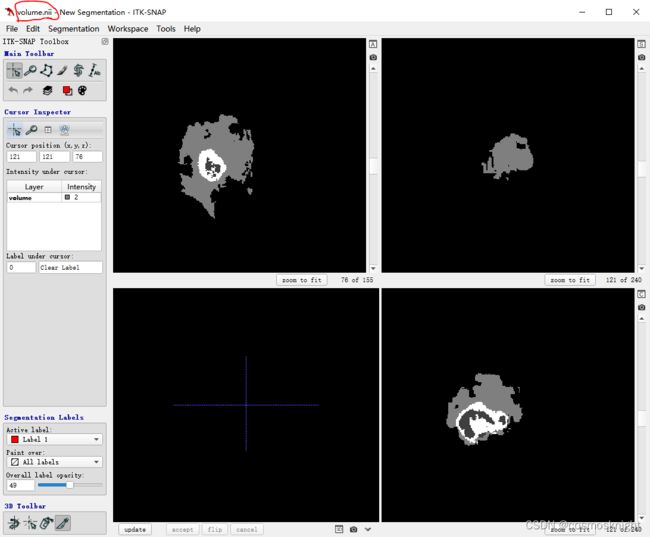
NIFTI格式除了图片的像素信息外,还包含很多其他信息,这些信息应该也是可以通过代码直接获取,本文未做这些尝试,感兴趣的读者可以自行百度或Google。因为本文只是为了提取像素信息,所以最终生成的volume.nii文件中已丢失了原始nii中的一些信息(如CT切片方向等)。下图展示最终的结果:
本文部分参考:
python 3.x - How to convert Multiple png or Jpeg images into one nifti image by python3? - Stack Overflow