limma | 配对样本的差异分析怎么搞!?(一)

1写在前面
最近在用limma包做配对样本的差异分析,在这里和大家分享一下吧。
大家可以先思考一下,配对和非配对的结果一样吗??
应用场景: 同一病人的癌和癌旁样本,同一样品的多时间点测序等。
2用到的包
rm(list = ls())
library(tidyverse)
library(limma)
library(GEOquery)
3示例数据
这里我从GEO数据库上download了一个dataset。
在3个样本中对T细胞和B细胞分别进行了转录组分析。
每个样本的细胞都分为Control或anti-BTLA组。
我们先常规下载数据吧,boxplot不是很齐啊,强迫症的我必须标准化!
GSE194314 <- getGEO('GSE194314', destdir=".",getGPL = F)
exprSet <- exprs(GSE194314[[1]])
boxplot(log2(exprSet))

exprSet <- normalizeBetweenArrays(exprSet) %>%
log2(.)
boxplot(exprSet)
nice!~ 齐了,接着做吧。
4获取分组数据
pdata <- pData(GSE194314[[1]])

5整理分组数据
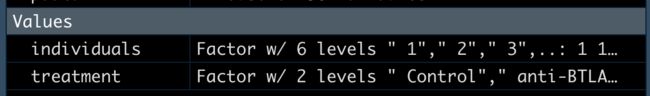
这里我们提取出分组数据后转为factor。
individuals <- factor(unlist(lapply(pdata$characteristics_ch1.1,function(x) strsplit(as.character(x),":")[[1]][2])))
treatment <- unlist(lapply(pdata$characteristics_ch1.2,function(x) strsplit(as.character(x),":")[[1]][2]))
treatment <- factor(treatment,levels = unique(treatment))
6非配对处理
6.1 整理分组矩阵
这里我们只把treatment作为分组信息纳入design中,不进行配对。
design_non_paried <- model.matrix(~ 0 + treatment)
colnames(design_non_paried) <- c("Control","anti-BTLA")
fit1 <- lmFit(exprSet,design_non_paried)
fit1 <- eBayes(fit1)
6.2 差异分析
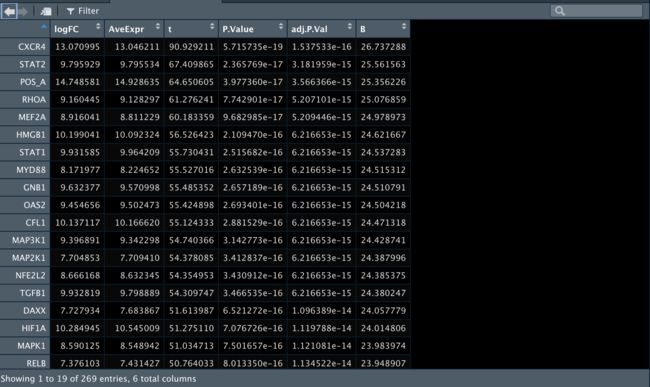
allDiff_non_paired <- topTable(fit1,
adjust = 'BH',
coef = "anti-BTLA",
n = Inf,
#p.value = 0.05
)
7配对处理
7.1 整理分组矩阵
design_paried <- model.matrix(~ individuals + treatment)
fit2 <- lmFit(exprSet,design_paried)
fit2 <- eBayes(fit2)
7.2 差异分析
allDiff_paired <- topTable(fit2,
adjust = 'BH',
coef = "treatment anti-BTLA",
n = Inf)
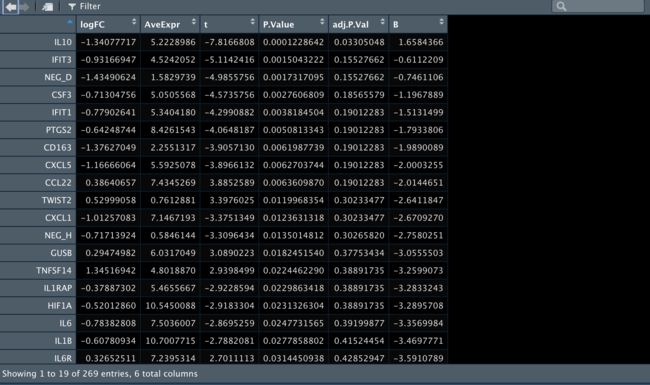
8对比结果
我们直接用火山图可视化对比一下吧。 这里我们把阈值设置为|logFC|>1,pvalue<0.05。
library(EnhancedVolcano)
library(patchwork)
p1 <- EnhancedVolcano(allDiff_non_paired,
lab = rownames(allDiff_non_paired),
x = 'logFC',
y = 'P.Value',
title = 'non_paired',
pointSize = 3.0,
labSize = 6.0,
legendPosition = 'right',
pCutoff = 0.05,
FCcutoff = 1)
p2 <- EnhancedVolcano(allDiff_paired,
lab = rownames(allDiff_paired),
x = 'logFC',
y = 'P.Value',
title = 'paired',
pointSize = 3.0,
labSize = 6.0,
legendPosition = 'right',
pCutoff = 0.05,
FCcutoff = 1)
p1 + p2
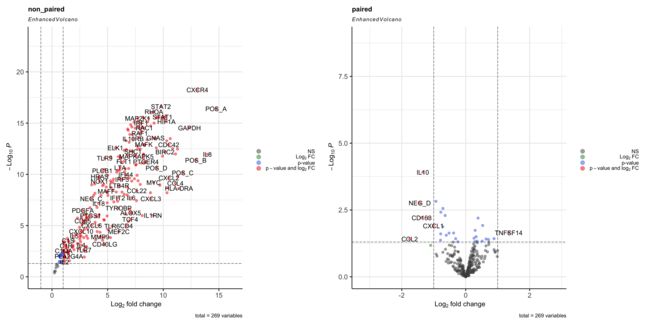
这配对和非配对的区别还是挺大的!
9小彩蛋
细心的小伙伴肯定发现了,这里我们假设T细胞和B细胞是同一个细胞,不进行区分。
但实际上需要进行T细胞和B细胞分层对比,下期我们再介绍Multi-level如何处理吧。

点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰

本文由 mdnice 多平台发布