医学图像处理_医学图像处理案例(二十)——医学图像处理案例代码详解
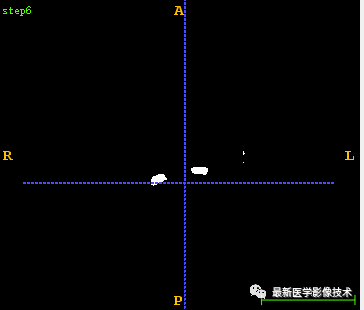
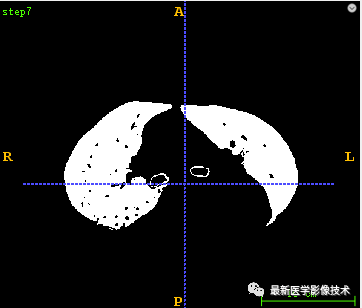
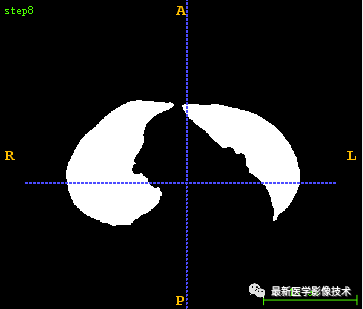
在前面分享的医学图像处理案例中,给出了很多具体案例,但有些读者还是渴望可以深入分享案例代码详解。那么今天我将从骨骼分割,气管分割,肺组织分割,血管分割这四个具体案例来详细讲解如何来实现。 1、骨骼分割代码详解 1.1、采用固定阈值分割,一般骨骼的HU值在300以上,但是发现心脏和主动脉的HU值与一些骨骼的HU比较接近,所以我们设置固定阈值为100,把骨骼和心脏及主动脉都分割出来。
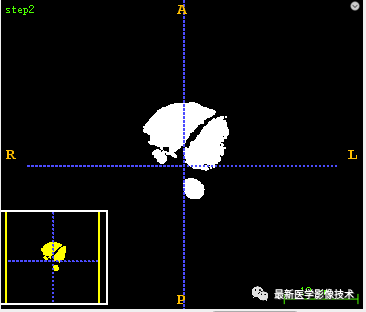
1.2、 首先采用形态学开操作,将骨骼和心脏和主动脉连接的部分断开,然后再取最大连通域可以得到粗略的心脏和主动脉图像。
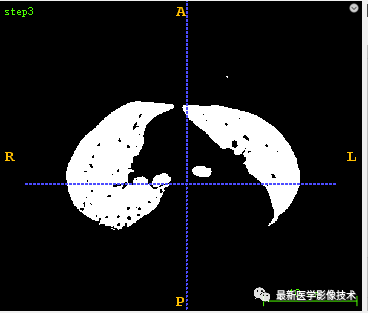
1.3、 再将步骤1.1的结果与步骤1.2的结果相减。
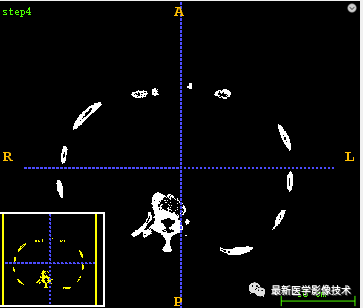
1.4、 对步骤1.3的结果求取最大连通域得到骨骼Mask区域。
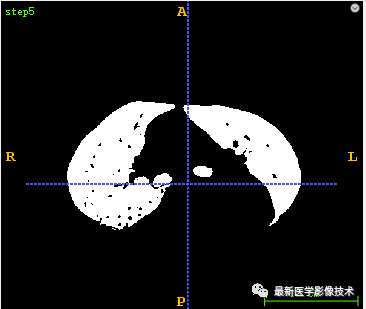
1.5、将得到的Mask图像与原始图像进行逻辑与操作得到最后的气管分割结果图像。
代码:
def skeletonSegment(pathDicom): sitk_src = dicomseriesReader(pathDicom) # 1 sitk_seg = BinaryThreshold(sitk_src, lowervalue=100, uppervalue=3000) sitk.WriteImage(sitk_seg, 'step1.mha') # 2 sitk_open = MorphologicalOperation(sitk_seg, kernelsize=2, name='open') sitk_open = GetLargestConnectedCompont(sitk_open) sitk.WriteImage(sitk_open, 'step2.mha') # 3 array_open = sitk.GetArrayFromImage(sitk_open) array_seg = sitk.GetArrayFromImage(sitk_seg) array_mask = array_seg - array_open sitk_mask = sitk.GetImageFromArray(array_mask) sitk_mask.SetDirection(sitk_seg.GetDirection()) sitk_mask.SetSpacing(sitk_seg.GetSpacing()) sitk_mask.SetOrigin(sitk_seg.GetOrigin()) sitk.WriteImage(sitk_mask, 'step3.mha') # 4 skeleton_mask = GetLargestConnectedCompont(sitk_mask) sitk.WriteImage(skeleton_mask, 'step4.mha') # 5 sitk_skeleton = GetMaskImage(sitk_src, skeleton_mask, replacevalue=-1500) sitk.WriteImage(sitk_skeleton, 'step5.mha') return sitk_skeletondef tracheaSegment(pathDicom): sitk_src = dicomseriesReader(pathDicom) lstSeeds = [] seed1 = [246, 268, 835] seed2 = [241, 287, 798] seed3 = [272, 277, 798] lstSeeds.append(seed1) lstSeeds.append(seed2) lstSeeds.append(seed3) sitk_mask = RegionGrowThreshold(sitk_src, lstSeeds, -1024, -900) sitk.WriteImage(sitk_mask, 'tracheamask.mha') sitk_trachea = GetMaskImage(sitk_src, sitk_mask, replacevalue=-1500) return sitk_tracheadef lungSegment(pathDicom): sitk_src = dicomseriesReader(pathDicom) # 1 sitk_seg = BinaryThreshold(sitk_src, lowervalue=-300, uppervalue=2000) sitk.WriteImage(sitk_seg, 'step1.mha') # 2 sitk_floodfilled = FloodFilled(sitk_seg) sitk.WriteImage(sitk_floodfilled, 'step2.mha') # 3 sitk_xorop = sitk.XorImageFilter() sitk_mask1 = sitk_xorop.Execute(sitk_seg, sitk_floodfilled) sitk_notop = sitk.NotImageFilter() sitk_mask2 = sitk_notop.Execute(sitk_mask1) sitk.WriteImage(sitk_mask2, 'step3.mha') # 4 sitk_mask3 = FillHole(sitk_mask2) sitk.WriteImage(sitk_mask3, 'step4.mha') # 5 sitk_mask4 = RemoveSmallConnectedCompont(sitk_mask3, 0.2) sitk.WriteImage(sitk_mask4, 'step5.mha') # 6 segtrachea lstSeeds = [] seed1 = [246, 268, 835] seed2 = [241, 287, 798] seed3 = [272, 277, 798] lstSeeds.append(seed1) lstSeeds.append(seed2) lstSeeds.append(seed3) sitk_tracheamask = RegionGrowThreshold(sitk_src, lstSeeds, -1024, -900) sitk.WriteImage(sitk_tracheamask, 'step6.mha') # 7 lung reduce trachea array_tracheamask = sitk.GetArrayFromImage(sitk_tracheamask) array_mask4 = sitk.GetArrayFromImage(sitk_mask4) array_mask4 = array_mask4 - array_tracheamask sitk_mask4 = sitk.GetImageFromArray(array_mask4) sitk_mask4.SetDirection(sitk_tracheamask.GetDirection()) sitk_mask4.SetSpacing(sitk_tracheamask.GetSpacing()) sitk_mask4.SetOrigin(sitk_tracheamask.GetOrigin()) sitk.WriteImage(sitk_mask4, 'step7.mha') # 8 sitk_mask4 = MorphologicalOperation(sitk_mask4, kernelsize=3, name='open') sitk_mask5 = MorphologicalOperation(sitk_mask4, kernelsize=9, name='close') sitk.WriteImage(sitk_mask5, 'step8.mha') # 9 sitk_lung = GetMaskImage(sitk_src, sitk_mask5, replacevalue=-1500) sitk.WriteImage(sitk_lung, 'step9.mha') return sitk_lungdef vessleSegment(pathDicom): sigma_minimum = 0.2 sigma_maximum = 3. number_of_sigma_steps = 8 lowerThreshold = 40 output_image = 'vessel.mha' input_image = itk.imread(pathDicom, itk.F) # 1 ImageType = type(input_image) Dimension = input_image.GetImageDimension() HessianPixelType = itk.SymmetricSecondRankTensor[itk.D, Dimension] HessianImageType = itk.Image[HessianPixelType, Dimension] objectness_filter = itk.HessianToObjectnessMeasureImageFilter[HessianImageType, ImageType].New() objectness_filter.SetBrightObject(True) objectness_filter.SetScaleObjectnessMeasure(True) objectness_filter.SetAlpha(0.5) objectness_filter.SetBeta(1.0) objectness_filter.SetGamma(5.0) multi_scale_filter = itk.MultiScaleHessianBasedMeasureImageFilter[ImageType, HessianImageType, ImageType].New() multi_scale_filter.SetInput(input_image) multi_scale_filter.SetHessianToMeasureFilter(objectness_filter) multi_scale_filter.SetSigmaStepMethodToLogarithmic() multi_scale_filter.SetSigmaMinimum(sigma_minimum) multi_scale_filter.SetSigmaMaximum(sigma_maximum) multi_scale_filter.SetNumberOfSigmaSteps(number_of_sigma_steps) itk.imwrite(multi_scale_filter.GetOutput(), "step1.mha") # 2 OutputPixelType = itk.UC OutputImageType = itk.Image[OutputPixelType, Dimension] rescale_filter = itk.RescaleIntensityImageFilter[ImageType, OutputImageType].New() rescale_filter.SetInput(multi_scale_filter) itk.imwrite(rescale_filter.GetOutput(), "step2.mha") # 3 thresholdFilter = itk.BinaryThresholdImageFilter[OutputImageType, OutputImageType].New() thresholdFilter.SetInput(rescale_filter.GetOutput()) thresholdFilter.SetLowerThreshold(lowerThreshold) thresholdFilter.SetUpperThreshold(255) thresholdFilter.SetOutsideValue(0) thresholdFilter.SetInsideValue(255) itk.imwrite(thresholdFilter.GetOutput(), "step3.mha")