MICCAI2022 | NestFormer用于脑肿瘤分割的嵌套模态感知的Transformer
NestedFormer:用于脑肿瘤分割的嵌套模态感知转换器
摘要:多模态磁共振成像通过提供丰富的互补信息,在临床实践中经常被用于诊断和研究脑肿瘤。以前的多模态MRI分割方法通常是在网络的早期/中期通过串联(cat)多模态MRI来执行模态融合,这很难探索模态之间的非线性依赖关系。论文提出一种新的嵌套的模态感知transformer(NestedFormer)来显式地探索用于脑肿瘤分割的多模态磁共振成像的模态内和模态间的关系。在基于transformer的多编码器和单解码器结构的基础上,对不同模态的高层表示进行嵌套的多模态融合,并且在较低的尺度上应用模态敏感门控(MSG)以实现更有效的跳层连接。具体地说,多模态融合是在提出的嵌套的模态感知特征聚集(NMaFA)模块中进行的,该模块通过一个三维的空间注意力transformer来增强单个模态的长距离相关性,并通过跨模态注意力transformer进一步补充模态之间的关键上下文信息。在BraTS2020基准和私有的脑膜瘤分割(MeniSeg)数据集上的广泛实验表明,NestedFormer明显优于最先进的分割。
1 引言
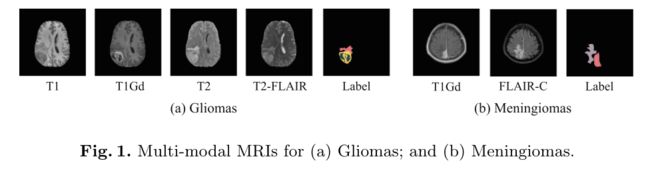
脑肿瘤是世界上最常见的癌症之一,其中胶质瘤是最常见的恶性脑肿瘤,具有不同程度的侵袭性,脑膜瘤是成人最常见的原发性颅内肿瘤。多模态磁共振成像(MRI)通过为分析脑肿瘤提供丰富的补充信息而在临床上被常规使用。具体而言,对于胶质瘤,常用的MRI序列是T1加权(T1-weighted,T1)、对比后T1加权(post-contrast T1-weighted,T1Gd)、T2加权( T2-weighted,T2)和T2流体衰减反转恢复(T2 Fluid Attenuation Inversion Recovery,T2-FLAIR)图像;每个序列在区分肿瘤、瘤周水肿和肿瘤核心方面具有不同的作用,如(a)
对于脑膜瘤,它们在T1Gd和增强T2-FLAIR(简称FLAIR-C)MRI图像上具有不同的特征性表现;见(b)。因此,从多模态MRI中自动分割脑肿瘤结构对于临床诊断和治疗规划非常重要。
近年来,卷积神经网络(CNNs)在脑肿瘤分割中取得了巨大的成功。主流模型建立在具有跳过连接的编码器-解码器架构[22]上,包括S3DUNet、SegResNet、HPU-Net等。最近的工作有工作探索了Transformer,以对图像内的长期依赖性进行建模。例如,TransBTS利用3D-CNN提取局部空间特征,并应用Transformer对高级特征的全局依赖性进行建模。UNETR使用ViT转换器作为编码器来学习上下文信息,该信息通过多分辨率(resolutions)的跳过连接与基于CNN的解码器合并。然而,这些方法中的Transformer用于增强编码路径,而无需为多模态融合进行特定设计。
为了利用多模态信息,大多数现有方法采用早期融合策略,其中多模态图像被连接作为网络输入。然而,这种策略很难探索不同模态之间的非线性关系。为了缓解这个问题,最近的工作遵循分层融合策略,其中不同编码器提取的特定于模型的特征在网络的中间层进行融合,并共享相同的解码器。在HyeprDenseNet中,每个模态都有一个单独的流,并且在同一流内的层之间以及不同流之间引入了密集连接。MAML通过不同模态特定FCN嵌入多模态图像,然后应用模态感知模块回归注意力图,以融合模态特定特征。然而,这些多模态融合方法没有在模态内和跨模态内建立长距离空间依赖关系,因此它们不能充分利用不同模态的互补信息。
动机
- 从多模态磁共振成像对中自动分割脑瘤结构有助于临床诊断和治疗规划。
- 现有多模态MRI分割方法难以探索不同模态间的非线性关系,通道融合方法没有建立通道内和通道间的长期空间依赖关系,没有针对多模态融合进行具体设计,不能充分利用不同通道间的互补信息。
贡献
-
提出了一种新的嵌套式感知转换器(NestedFormer),用于有效和鲁棒的多模态脑肿瘤分割。首先设计了一个有效的Global Poolformer,从不同的MRI模式中提取有区别的体积空间特征,并更加强调全局相关性。
-
为了更好地提取互补特征,并支持任意数量的模态进行融合,我们提出了一种新的嵌套模态感知特征聚合(NMaFA)模块。它明确地考虑了单模态空间一致性和跨模态一致性,并利用嵌套Transformer来建立模态内和模态间的长程相关性,从而获得更有效的特征表示。
-
此外,我们设计了一个计算效率高的三维空间注意力(TSA)范式来加速3D空间一致性计算。为了提高解码中的特征重用效果,开发了一种新的模态敏感门控(MSG)模块,用于动态过滤模态感知的低分辨率特征,实现有效的跳跃连接。
2 方法
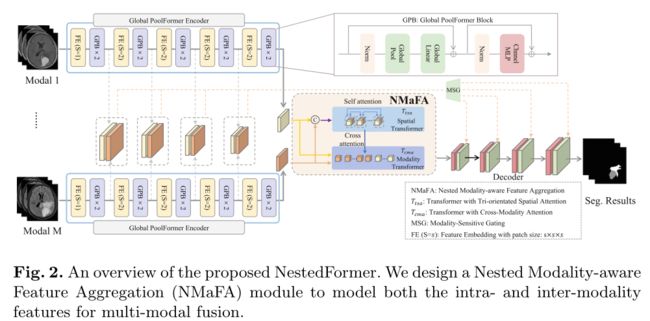
三个组件组成:
1)多个编码器,以获得不同模态的多尺度表示;
2)NMaFA融合模块,以探索多模态高级嵌入内部和之间的相关特征;
3)选通策略,以选择性地将模态敏感的低分辨率特征传输到解码器。
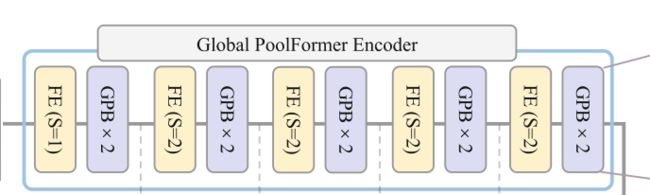
2.1 Global Poolformer编码器
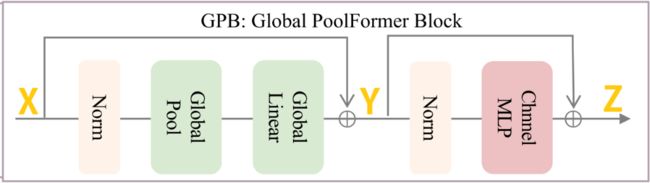
最近的工作表明,与CNN相比,transformer更有利于对全局信息进行建模。更好地提取每个模态的局部上下文信息,我们将Poolformer[26]扩展为模态特定编码器。如[26]中所讨论的,用平均池代替Transformer中的计算密集型注意力模块可以获得比最近的Transformer和MLP类模型更好的性能。因此,为了增强全局信息,我们设计了全局PoolFormer Block(GPB),它利用全局池而不是PoolFormer中的平均池,然后是完全连接的层。
Global Poolformer Encoder,每个包含五组,每组一个特征嵌入(FE)层和两个GPB块。 FE是一个三维卷积。编码器提取出多尺度特征。
如图2所示,给定输入特征嵌入X,GPB块由可学习全局池(GP)和MLP子块组成。输出Z被计算为,
2.2 嵌套模态感知特征聚合
给定高级特征F_1,…,F_M,NMaFA以嵌套方式,利用基于空间注意力的Transformer :T_tsa和基于跨模态注意力的Transformer:T_cma;参见图3。
-
首先,Transformer Ttsa利用自注意力来计算每个模态内空间中不同patch之间的长距离相关性。具体将F_1,…,F_M,先通道cat拼接得到
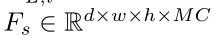
利用patch embedding layer 映射成 token sequence
。T_tsa以 和位置编码作为输入,输出空间增强的特征
和位置编码作为输入,输出空间增强的特征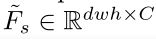
-
其次,Transformer Tcma利用交叉注意力计算计算不同模态之间的全局关系,实现模态间融合。将F_1,…,F_M在空间维度中拼接,以获得flatten 序列

。这里,P(P=32)表示通过令牌学习器策略学习的主要令牌的数量,这有助于减少计算范围,尤其是当令牌数量随着更多模态而大幅增加时。之后,将
和
都馈送到T_cma中,以获得模态增强的特征嵌入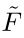
-
还要注意,我们的两个模块不同于传统的通道空间注意力网络,后者重新加权通道和空间的特征图。两个Transformer块,不同于以往的串联或者并联的通道、空间注意力,而是使用嵌套形式融合。
具有三维空间注意力的Transformer
为了提高体积嵌入的空间注意力的计算效率,受到Axial Transformer和Swin Transformer的启发,我们利用轴向注意力MHA_z、平面注意力MHA_xy和窗口注意力MHA_w。具体而言,MHA_z模拟了沿垂直方向的特征标记之间的远程关系;MHA_xy对每个切片内的远程关系进行建模;MHA_w使用滑动窗口来建模局部3D窗口之间的关系。我们分别对MHA_z和MHA_xy使用轴向和平面可学习的绝对位置编码,并对窗口式注意力MHA_z使用相对位置编码。所得注意力计算如下,
该模型不仅增强了局部重要区域的特征提取,而且计算全局特征依赖关系的计算量更小。
具有跨模态注意力的Transformer
通过在通道维度中串联特征,T_tsa主要增强了每个模态内的相关性,并产生了![]()
,尽管模态间的集成也通过patch嵌入进行。为了明确地探索模态间的关系,我们沿着空间维度连接不同模态的特征token,从而产生![]() ;然后使用交叉注意变换器T_cma将模态依赖信息增强为
;然后使用交叉注意变换器T_cma将模态依赖信息增强为![]() ;见图3(c)。交叉关注的输入三元组(Query,Key,Value)计算如下
;见图3(c)。交叉关注的输入三元组(Query,Key,Value)计算如下
![]()
T_cma生成的令牌序列[外链图片转存失败,源站可能有防盗链机制,建议将图片保存下来直接上传![]() 融合并增强了输入特征,增加了接收域和跨通道的全局相关性。
融合并增强了输入特征,增加了接收域和跨通道的全局相关性。
2.3 模态敏感门控
在特征解码中,我们首先将token![]() 折叠回高级4D特征图
折叠回高级4D特征图![]()
。RL通过3D卷积和2×上采样操作以规则的自下而上的方式逐步处理,以恢复全分辨率特征图![]()
用于分段,其中Nc是分段数。请注意,编码器功能是多模态的。因此,我们在跳跃连接中设计了一种模态敏感门控策略,以过滤编码器特征![]()
。具体而言
3 实验
3.1 实现细节
我们的NestedFormer在NVIDIA GTX 3090 GPU上的PyTorch1.7.0中实现。通过Xavier初始化参数。损失函数是soft dice loss和cross-entropy loss的组合,我们采用了权重衰减为10的-5次方的AdamW优化器。根据经验,学习率设置为10−4,我们依次采用了两个T_tsa和一个T_cma。在MHAw中,BraTS2020的窗口大小设置为(2,2,2),MeniSeg的窗口大小为(2、4,4)。
3.2 数据集和评估指标
为了进行评估,我们使用了从天津大学脑医学中心天津环湖医院收集的公共脑肿瘤分割数据集BraTS2020和私人3D脑膜瘤分割数据集(MeniSeg)。采用Dice评分和95%Hausdorff距离(HD95)进行定量比较。
BraTS2020数据集。BraTS2020训练数据集包含369个对齐的四模态MRI数据(即T1、T1Gd、T2、T2-FLAIR),以及专家分割掩码(即GD增强肿瘤、瘤周水肿和肿瘤核心)。每个模态具有155×240×240的体积,并且已经重新采样和共同注册。
分割任务旨在分割整个肿瘤(WT)、增强肿瘤(ET)和肿瘤核心(TC)区域。在最近的工作之后,我们将数据集随机分为训练(315)、验证(17)和测试(37)。
脑膜瘤数据集。MeniSeg数据集包含来自脑膜瘤患者的110个带注释的模态MRI(即T1Gd和FLAIR-C),这些患者在2016年3月至2021年3月期间接受了肿瘤切除。MRI扫描使用四个3.0T MRI扫描仪(Skyra、Trio、Avanto、Prisma,西门子)进行。两位放射科医生在T1Gd和FLAIR-C核磁共振成像上对脑膜瘤肿瘤和水肿进行了标注,第三位经验丰富的放射科医生进行了检查。每个模态数据的体积为32×256×256,并对齐到同一空间中,并采样到[32,192,192]的体积大小进行训练。对所有比较方法进行了双重交叉验证。
3.3 与SOTA方法的比较
所有方法在BraTS2020上最多训练300个epoch,在MeniSeg上训练200个epoch。
BraTS2020。表1报告了三个区域(WT、TC和ET)的Dice和HD95得分,以及BraTS2020上所有方法的平均得分。
显然,我们的NestedFormer在WT上获得了最大的Dice分数,在TC上获得了最高的Dice得分,在TC中获得了最小的HD95分数,并且我们的方法在ET上也排名第二,在WT和ET上排名第二。更重要的是,我们的方法具有最佳的定量性能,Dice和HD95的平均得分分别为0.861和5.051。值得注意的是,HD95是两组点之间的距离差,比Dice敏感。因此,Dice经常被用作主要度量,HD95作为参考。我们还对UNETR、TransBTS和我们的方法进行了双重交叉验证,而我们的方法在WT和TC方面优于两种方法,并且非常接近ET中的最佳结果。
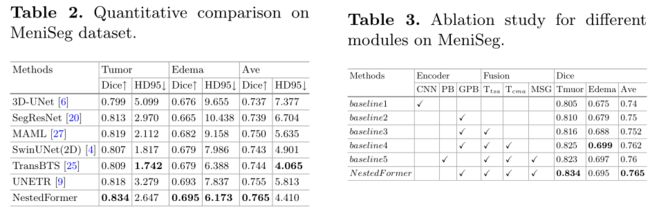
MeniSeg。在表2中,我们列出了我们网络的Dice和HD95评分,并比较了MeniSeg数据集上肿瘤和水肿区域的方法以及平均指标。在所有比较方法中,MAML在肿瘤分割处的Dice评分最大,为0.819,而UNETR在水肿分割处的Dice评分最大为0.693,平均Dice评分为0.755。
相比之下,我们的方法对脑膜瘤肿瘤有1.5%的Dice改善,对水肿有0.2%的Dice改进,平均Dice改善1.0%。关于HD95,我们的方法在肿瘤分割上获得了2.647的第四小分数,在水肿分割上获得6.173的最小分数。
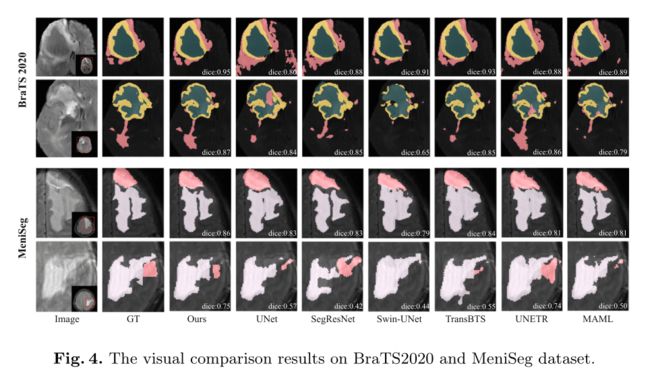
BraTS2020和MeniSeg的视觉比较。图4直观地比较了我们的网络和SOTA方法在BraTS2020和MeniSeg上预测的分割结果。从这些可视化结果中,我们可以发现与所有比较方法相比,我们的方法可以更准确地分割脑肿瘤和瘤周水肿区域。背后的原因是,我们的方法能够通过明确探索多模态之间的模态内和模态间关系来更好地融合多模态MRI。
3.4 消融研究
我们对MeniSeg数据集进行消融研究,以评估我们方法中主要模块的贡献;参见表3。我们不仅比较了基于CNN、PB和GP的三种不同编码器主干的效果,还验证了我们提出的融合模块的效果。其中,baseline1使用多个U-Net编码器来提取不同模态图像的特征,并通过级联来执行特征融合。baseline2-baseline4使用多个GPB编码器来提取特征,并分别通过简单卷积(w/o Ttsa和Tcma)进行跳过连接(见图3)。baseline5使用提议的NMaF A模块(包括Ttsa和Tcma)以及MSG,用编码器中的原始PoolFormer块(PB)替换GPB块。可以清楚地观察到,与基线2相比,使用NMaFA模块增强了远距离相关性信息的提取,并有效地改进了分割结果,而GPB通过考虑全局信息而优于PB。此外,添加了MSG模块以提高跳过连接的特征重用能力,这进一步提高了分割效果,在MeniSeg数据集上实现了最佳平均分割Dice(0.765)。
4 结论
我们提出了一种新的多模态分割框架,称为NestedFormer。该架构通过使用多个Global Poolformer Encoder 来提取M个模态的特征。然后,高级特征由NMaFA模块有效融合,低级特征由模态敏感门(MSG)模块选择。通过这些提出的模块,网络有效地从不同模态中提取并分层融合特征。
我们提出的NestedFormer的有效性在BraTS2020和MeniSeg数据集上得到了验证。我们的框架与模态无关,可以扩展到其他多模态医学数据。在未来的工作中,我们将探索更有效的低级别特征融合,以进一步提高分割性能。